1. 领域背景与文献引入
文献英文标题:A DNA methylation-based test for esophageal cancer detection;发表期刊:Biomarker Research;影响因子:未公开;研究领域:食管癌的DNA甲基化生物标志物研究。
食管癌是全球第七大常见恶性肿瘤及第六大癌症相关死亡原因,多数患者确诊时已处于局部晚期或转移阶段,5年生存率仅5-20%。尽管手术、放化疗等治疗手段有所进步,但晚期诊断仍是改善预后的核心障碍,亟需能够早期检测的生物标志物。表观遗传(epigenetic)改变尤其是DNA甲基化,因是食管癌发生的早期事件(存在于癌前病变如Barrett食管)、稳定性高且可定量检测,成为近年来的研究热点。现有研究表明,食管癌的不同组织学亚型(食管腺癌EA、食管鳞癌ESCC)具有不同的甲基化模式,但多数研究聚焦于单个基因或小样本,缺乏针对不同亚型的特异性检测组合,也较少关注治疗后残留病灶的检测。在此背景下,本研究旨在验证COL14A1、GPX3、ZNF569三个基因的甲基化水平作为食管癌早期检测及治疗反应预测生物标志物的潜力,为临床提供更精准的检测策略。
2. 文献综述解析
作者的综述逻辑围绕“临床需求-表观遗传 biomarker 潜力-现有研究局限-本研究创新”展开:首先强调食管癌晚期诊断的困境,指出早期检测对改善预后的重要性;接着阐述DNA甲基化作为早期事件的优势,提到CDKN2A、APC等常见甲基化基因,但不同亚型(EA/ESCC)有特异性甲基化模式;随后总结现有研究的局限性:样本量小(n≤40)、检测方法不一致(如MSP vs 定量甲基化特异性PCR)、人群差异(亚洲vs高加索)、缺乏治疗后残留病灶研究;最后提出本研究的创新——验证三个高特异性基因的甲基化,分层分析亚型,评估治疗后样本。
现有研究的关键结论:DNA甲基化是食管癌早期事件,可存在于癌前病变;不同亚型有不同甲基化模式(EA涉及Barrett食管相关基因,ESCC涉及鳞状上皮基因);局限性:部分研究样本量小、方法差异导致结果不可比、人群适用性有限。本研究的创新点:① 选择高特异性基因避免随机性;② 分层分析亚型提出特异性 panel;③ 验证治疗后样本的甲基化水平,监测残留病灶。
3. 研究思路总结与详细解析
本研究遵循“文献筛选-样本收集-甲基化检测-临床关联分析-性能评估-治疗后验证”的逻辑,目标是验证三个基因甲基化作为食管癌早期检测及治疗反应预测的生物标志物;核心科学问题包括:三个基因甲基化能否区分癌与正常组织、是否随亚型变化、治疗后能否反映残留病灶。技术路线通过定量甲基化特异性PCR(QMSP)定量检测,结合ROC曲线、Kruskal-Wallis检验等评估性能。
3.1 样本收集与临床数据整理
实验目的:获取食管癌及正常组织样本,整理临床病理信息(亚型、TNM分期、治疗史)。方法细节:收集葡萄牙波尔图肿瘤研究所2007-2017年的124例食管癌福尔马林固定石蜡包埋(FFPE)样本(88例治疗 naive、36例新辅助治疗后),及56例正常食管FFPE样本(来自无食管癌的胃癌患者);由病理学家确认组织学亚型(EA40例、ESCC48例),记录临床数据。结果解读:三组样本年龄无显著差异(p=0.06),排除年龄对结果的干扰。
3.2 DNA提取与甲基化水平检测
实验目的:定量检测三个基因的甲基化水平。方法细节:用Norgen Biotek的FFPE RNA/DNA提取试剂盒提取DNA,Zymo Research的EZ DNA甲基化试剂盒进行亚硫酸氢盐修饰(未甲基化C转化为U,甲基化C保留);使用Roche LightCyler 480 II仪器进行QMSP,反应体系含2μL修饰DNA、5μL GRiSP的Xpert Fast SYBR试剂,引物针对甲基化CpG位点;以β-actin归一化DNA输入量,用Merck Millipore的CpGenome甲基化DNA制作标准曲线确保PCR效率。结果解读:所有样本成功检测,重复样本一致性良好(CV<5%),方法可靠。
实验所用关键产品:Norgen Biotek的FFPE RNA/DNA Purification Plus Kit、Zymo Research的EZ DNA Methylation-Gold™ Kit、Roche的LightCyler 480 II、GRiSP的Xpert Fast SYBR试剂、Merck Millipore的CpGenome™ Universal Methylated DNA。
3.3 甲基化水平与临床病理特征的关联分析
实验目的:分析甲基化水平与亚型、TNM分期的关系。方法细节:用Kruskal-Wallis检验比较多组差异,Mann-Whitney U检验比较两组差异;按亚型分层分析。结果解读:ZNF569甲基化(ZNF569me)在食管癌与正常样本中差异极显著(p<0.001,图1);COL14A1甲基化(COL14A1me)在EA与正常中差异显著(p=0.002),GPX3甲基化(GPX3me)在ESCC与正常中差异显著(p=0.009);ESCC的COL14A1me、GPX3me高于EA(p=0.001、p=0.024),EA的ZNF569me高于ESCC(p=0.020,图3),表明甲基化具有亚型特异性。
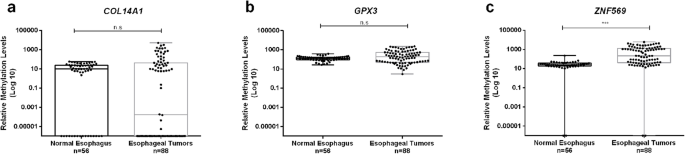
(图1:COL14A1、GPX3、ZNF569在正常与食管癌样本中的甲基化水平,ZNF569me差异极显著)
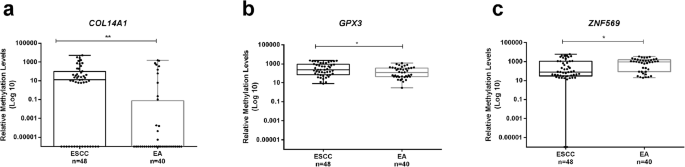
(图3:COL14A1、GPX3、ZNF569在EA与ESCC中的甲基化水平,ESCC的COL14A1me更高)
3.4 生物标志物性能评估
实验目的:评估单个基因及组合 panel 的检测性能。方法细节:用ROC曲线计算AUC,Youden指数确定最佳 cutoff,计算敏感性、特异性、准确性;组合 panel 以至少一个基因阳性为判定标准。结果解读:ZNF569me单独检测食管癌的AUC=0.8467,特异性>90%,敏感性69.3%;COL14A1me+ZNF569me检测EA的敏感性97.5%,准确性82.29%(AUC=0.91,图4a);GPX3me+ZNF569me检测ESCC的准确性81.73%(AUC=0.79,图4b),组合 panel 显著提升性能。
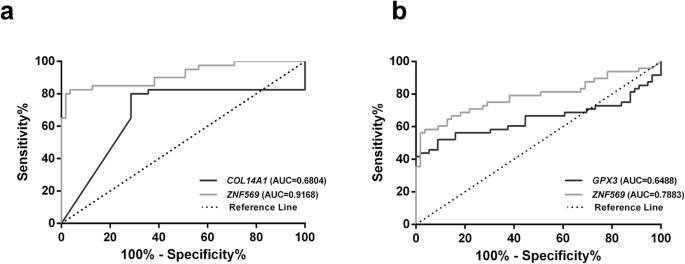
(图4:EA和ESCC的组合 panel ROC曲线,EA的COL14A1+ZNF569 AUC更高)
3.5 治疗后样本的甲基化验证
实验目的:验证治疗后样本的甲基化水平反映残留病灶。方法细节:收集36例治疗后样本(16例EA、20例ESCC),比较与正常样本的甲基化差异。结果解读:EA治疗后样本的ZNF569me、GPX3me显著高于正常(p<0.001),ESCC治疗后样本的GPX3me显著高于正常(p=0.001,图5),提示甲基化可监测残留病灶。
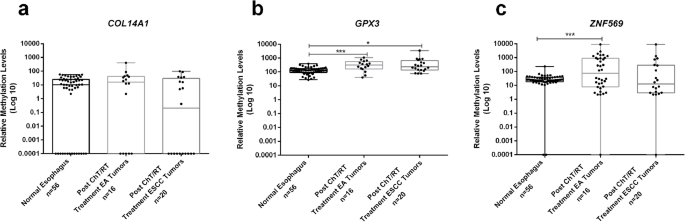
(图5:治疗后EA/ESCC与正常样本的甲基化水平,EA治疗后ZNF569me更高)
4. Biomarker研究及发现成果解析
本研究的Biomarker为COL14A1甲基化(COL14A1me)、GPX3甲基化(GPX3me)、ZNF569甲基化(ZNF569me),均为DNA甲基化生物标志物。筛选逻辑为“文献筛选(高特异性>98%)→排除无引物基因→组织样本验证→分层分析亚型”,研究成果包括通用标志物、亚型特异性 panel 及治疗后残留病灶检测。
Biomarker定位与筛选逻辑
三个基因的甲基化均为食管癌生物标志物,其中ZNF569me为通用标志物,COL14A1me针对EA,GPX3me针对ESCC。筛选逻辑:先通过文献筛选高特异性候选基因(5个),排除无法设计引物的ZNF345、EPB41L3后,选择COL14A1、GPX3、ZNF569;随后通过组织样本(治疗 naive、治疗后、正常)的QMSP检测,验证与食管癌的关联;最后分层分析亚型,确定各亚型的特异性组合。
研究过程与核心成果
Biomarker来源为食管癌及正常组织的FFPE样本;验证方法为QMSP,通过β-actin归一化确保定量准确。特异性与敏感性数据:ZNF569me单独检测食管癌的特异性>90%,敏感性69.3%(AUC=0.8467);COL14A1me+ZNF569me检测EA的敏感性97.5%,准确性82.29%(AUC=0.91);GPX3me+ZNF569me检测ESCC的准确性81.73%(AUC=0.79);治疗后EA样本的ZNF569me、GPX3me显著高于正常(p<0.001),ESCC治疗后样本的GPX3me显著高于正常(p=0.001)。
核心成果:① ZNF569me作为通用标志物区分食管癌与正常;② 针对EA的COL14A1me+ZNF569me高敏感性 panel;③ 针对ESCC的GPX3me+ZNF569me高准确性 panel;④ 治疗后甲基化水平反映残留病灶,为避免不必要手术提供依据。创新性:首次提出食管癌亚型特异性甲基化 panel,拓展了生物标志物的临床应用(早期检测+残留病灶监测)。统计学结果均显著(p<0.05),其中ZNF569me的差异极显著(p<0.001),组合 panel 准确性均超80%。
