1. 领域背景与文献引入
文献英文标题:Elevation of brain-enriched miRNAs in cerebrospinal fluid of patients with acute ischemic stroke;发表期刊:Biomarker Research;影响因子:未公开;研究领域:急性缺血性脑卒中生物标志物研究。
缺血性脑卒中是全球范围内导致死亡和残疾的主要原因之一,其诊断在非典型症状(如短暂性脑缺血发作)或病因不明(约25%患者)时存在挑战[1]。当前用于亚型分类的TOAST系统依赖临床与辅助检查,仅具中等观察者间一致性[6,7],难以精准指导二级预防(如大动脉粥样硬化需抗血小板、心源性栓塞需抗凝)。MicroRNAs(miRNAs)作为稳定存在于体液中的非编码RNA,是潜在生物标志物[8-10],但既往研究多聚焦血液miRNAs,而血液miRNAs易受血细胞干扰(约58%血浆miRNAs来自血细胞)[36]。相比之下,脑脊液(CSF)因血脑屏障(BBB)隔离,更能特异性反映中枢神经系统(CNS)病理[37]。但目前CSF中miRNAs的研究极少,尤其是脑富集miRNAs(如miR-9、miR-124)的表达变化未明确,且技术差异可能影响结果可靠性。因此,本研究通过三种独立技术(下一代测序(NGS)、两种定量PCR(qPCR)),探讨CSF中脑富集miRNAs作为急性缺血性脑卒中生物标志物的潜力。
2. 文献综述解析
本文综述以“缺血性脑卒中诊断挑战→miRNAs生物标志物优势→血液与CSF miRNAs优劣→既往研究局限→本研究技术设计”为核心逻辑,系统评述领域现状:
现有研究关键结论:血液miRNAs易受血细胞干扰,无法准确反映脑内病理;CSF因BBB更具CNS特异性,但相关研究少且未聚焦脑富集miRNAs[27,49]。
现有研究局限性:样本量小、技术单一(多为qPCR)、未按梗死灶大小分层、未验证脑富集miRNAs。
本研究创新价值:首次采用三种技术(NGS、Exiqon qPCR、Applied Biosystems qPCR)分析CSF中miRNAs,按梗死灶大小分组以关联脑损伤程度,减少技术偏差对结果的影响。
3. 研究思路总结与详细解析
本研究整体框架为“标准化样本收集→多技术miRNA分析→分层结果解读→诊断模型验证”,核心目标是明确CSF中脑富集miRNAs作为急性缺血性脑卒中生物标志物的潜力,核心科学问题是脑富集miRNAs表达与脑损伤程度(梗死灶大小)的关联,技术路线遵循“假设-实验验证-数据建模-结论”闭环。
3.1 样本收集与患者分组
实验目的是获取标准化CSF样本及临床信息,确保组间可比性。方法细节:纳入21例急性缺血性脑卒中患者(发病3天内腰穿取CSF)和21例对照(非脑卒中神经系统疾病患者);所有患者行3T MRI(Philips Achieva)计算梗死灶体积(扩散加权成像),结合颈动脉超声、心脏遥测行TOAST分类。结果解读:两组基线特征匹配(Table 1);脑卒中患者按梗死灶大小分为>2cm³(n=7,大动脉粥样硬化/心源性栓塞,平均体积16.5cm³)和<2cm³(n=12,小动脉闭塞,平均体积0.7cm³)。产品关联:文献未提及具体产品,领域常规使用3T MRI扫描仪、0.7mm腰椎穿刺针、细胞离心机等。
3.2 三种miRNA分析技术的应用
实验目的是通过多技术并行,规避技术差异影响。方法细节:
1. 下一代测序(NGS):Trizol试剂(Invitrogen)提取100μl CSF RNA,Illumina TruSeq小RNA文库试剂盒构建文库,NextSeq500测序;sRNAbench比对hg19基因组/miRbase v20,edger软件TMM归一化。
2. Exiqon定量PCR(qPCR):miRCURY RNA提取试剂盒(Exiqon)提取200μl CSF RNA,miRCURY LNA Universal RT试剂盒反转录,372个miRNApanel(Human panel I)在LightCycler 480(Roche)上扩增;NormFinder筛选9个内参(如miR-15a-5p),2^-∆∆Ct法计算相对表达量(REL)。
3. Applied Biosystems定量PCR(qPCR):Norgen Biotek Total RNA Purification Kit提取200μl CSF RNA,TaqMan MicroRNA Reverse Transcription Kit反转录,TaqMan PreAmp Master Mix预扩增,24个候选miRNA在BioMark系统(Fluidigm)上验证;以miR-320a为内参计算REL。
结果解读:NGS检测到71个高频miRNAs(>90%样本),Exiqon qPCR检测到21个,Applied Biosystems qPCR验证21个;三种技术检测结果重叠但qPCR检测率更低(Additional file 4)。产品关联:实验所用关键产品:Trizol试剂(Invitrogen)、Illumina NextSeq500测序仪、miRCURY RNA提取试剂盒(Exiqon)、LightCycler 480 Real-Time PCR System(Roche)、Norgen Biotek Total RNA Purification Kit、TaqMan MicroRNA Reverse Transcription Kit(Applied Biosystems)、BioMark real-time microfluidic PCR system(Fluidigm)。
3.3 差异miRNAs分析与梗死灶大小关联
实验目的是明确miRNAs表达与脑卒中及梗死灶大小的关联。方法细节:edgeR分析NGS差异miRNAs(BH校正P<0.05),按梗死灶大小分组比较,两种qPCR验证。结果解读:NGS显示仅miR-1246、miR-128-3p在脑卒中组显著升高(BH校正后);大梗死灶患者中8个miRNAs(miR-9-5p、miR-128-3p等)显著升高(n=7 vs n=12,P<0.05);qPCR显示miR-9-3p、miR-124-3p在大梗死灶患者中升高,但未达统计学显著性。图片插入:
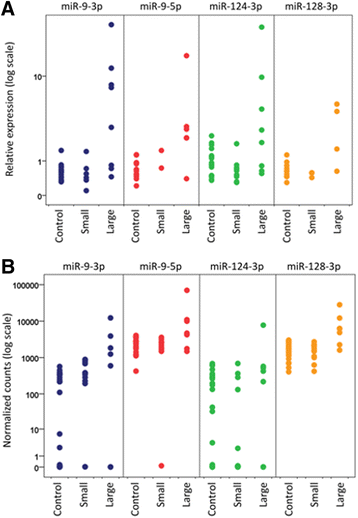
(四种脑富集miRNAs在不同分组的表达)。
3.4 诊断模型构建与验证
实验目的是评估miRNAs组合的诊断价值。方法细节:基于NGS Top15差异miRNAs,logistic lasso回归筛选最优组合,leave-one-out交叉验证。结果解读:7个miRNAs(miR-9-3p、miR-128-3p等)组合的分类准确率为0.75,ROC曲线下面积(AUC)为0.80。图片插入:
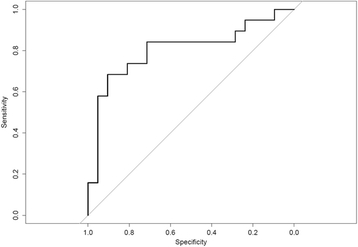
(组合模型ROC曲线)。
3.5 总miRNA表达与脑损伤关联
实验目的是探讨miRNAs总量与梗死灶大小的关系。方法细节:统计qPCR/NGS检测的miRNA总数,计算平均原始Ct值(qPCR)、总测序计数(NGS)。结果解读:大梗死灶患者的miRNA总数(qPCR/NGS)均高于小梗死灶及对照组,提示脑损伤导致CSF中miRNAs释放增加。图片插入:
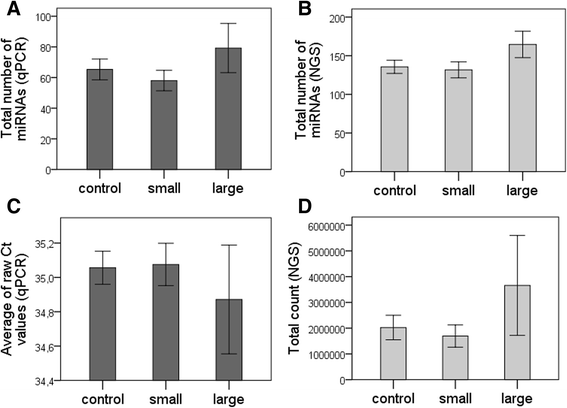
(不同分组miRNA总数及总量差异)。
4. Biomarker 研究及发现成果解析
Biomarker定位:本研究聚焦脑富集miRNAs(miR-9-5p、miR-9-3p、miR-124-3p、miR-128-3p),筛选逻辑为“NGS筛选高频miRNAs→梗死灶大小分层找差异→两种qPCR验证”,确保结果可靠性。
研究过程详述:Biomarker来源于急性缺血性脑卒中患者发病3天的CSF样本(细胞-free,避免细胞污染);验证方法包括NGS(全基因组测序)、Exiqon qPCR(372个miRNApanel)、Applied Biosystems qPCR(24个候选验证);特异性表现为仅在大梗死灶患者中升高,与脑损伤程度相关;敏感性方面,7个miRNAs组合AUC=0.80,准确率0.75,但单miRNA的显著性因技术而异(如miR-128-3p在NGS中显著,qPCR中无统计学意义)。
核心成果:1)脑富集miRNAs在大梗死灶患者CSF中一致升高,反映脑缺血导致的神经元损伤;2)现有方法下,仅能区分大梗死灶患者,无法作为通用诊断标志物;3)技术差异影响结果显著性,需标准化检测流程。
统计学结果:NGS中miR-128-3p在脑卒中组显著升高(BH校正P<0.05);大梗死灶 vs 小梗死灶患者中,miR-9-5p(n=7 vs n=12,P<0.05)、miR-128-3p(P<0.05)显著升高;组合模型AUC=0.80,准确率0.75(leave-one-out交叉验证)。
