1. 领域背景与文献引入
文献英文标题:Circumventing qPCR inhibition to amplify miRNAs in plasma;发表期刊:Biomarker Research;影响因子:未公开;研究领域:循环microRNA(c-miRNA)生物标志物检测技术优化。
循环microRNA(c-miRNA)因在血液、尿液等体液中稳定存在,且与癌症、传染病等多种疾病的发生发展密切相关,已成为生物标志物研究的核心方向之一。定量PCR(qPCR)是c-miRNA定量的“金标准”,但其准确性受样本基质干扰——肝素抗凝血浆中残留的锂肝素会抑制反转录酶(RT)活性,导致c-miRNA无法有效反转录为cDNA,最终影响qPCR检测灵敏度。现有解决方案多采用“反转录前用肝素酶处理RNA”,但需额外孵育2小时,步骤繁琐、耗时久,且增加RNA降解风险,这一技术瓶颈严重阻碍了血浆c-miRNA的生物标志物发现。
针对此问题,本研究通过优化肝素酶的处理步骤(将其整合到反转录过程中),旨在提高血浆c-miRNA的qPCR检测效率与灵敏度,为生物标志物开发提供技术支撑。
2. 文献综述解析
文献综述围绕“c-miRNA生物标志物检测的技术挑战”展开核心评述:首先,作者强调非编码RNA(尤其是miRNA)在基因表达调控中的核心作用——通过转录后调控维持细胞稳态,且因“稳定性高、组织特异性强”成为理想生物标志物;其次,对比血浆与血清的差异:血浆保留抗凝剂(如肝素),而血清无,但血浆更易通过静脉采血获取,是临床样本的主要类型;接着,重点分析肝素残留的干扰机制:肝素通过结合反转录酶和Taq酶,抑制c-miRNA的反转录和cDNA扩增,导致目标序列无法检测;最后,评述现有方法的局限性:传统“反转录前用黄杆菌肝素酶I处理RNA”需额外2小时孵育,延长实验流程,且RNA暴露于常温的时间增加,易降解。
本研究的创新点在于两点:一是改用拟杆菌肝素酶I(New England BioLabs),其能切割肝素中更多位点(包括2-O-硫酸化葡萄糖醛酸残基),降解效率更高;二是将肝素酶处理整合到反转录过程中(37°C孵育1小时),而非提前处理,从而简化步骤、减少RNA降解风险。
3. 研究思路总结与详细解析
本研究的核心目标是“优化血浆c-miRNA的qPCR检测方法,解决肝素残留抑制问题”;核心科学问题是“如何通过调整肝素酶处理步骤,在不增加流程复杂度的前提下提高qPCR灵敏度”;技术路线遵循“问题发现→条件优化→方法验证”的闭环逻辑。
3.1 血浆RNA提取与初始qPCR检测
实验目的是验证肝素抗凝血浆中c-miRNA检测的技术瓶颈。方法细节:用QIAamp循环核酸试剂盒(Qiagen)从250μl血浆中提取总RNA;用miScript II RT Kit(Qiagen)在HiSpec缓冲液中进行反转录(未加肝素酶);用定制miScript miRNA PCR Arrays(SA Biosciences)筛选cDNA;qPCR采用SYBR Green PCR Master Mix(miScript, Qiagen)和Bio-Rad iCycler thermocycler(带iQ5实时检测器),热循环条件为95°C激活15分钟,40个循环(94°C变性15秒、55°C退火30秒、70°C延伸30秒),最后熔解曲线分析。
结果解读:未处理的血浆RNA无法检测到c-miRNA(包括miRTC反转录对照,Ct>45),但阳性对照PPC(Positive qPCR Controls)的Ct保持在18-20,说明肝素残留抑制了反转录过程,而非qPCR扩增环节。
产品关联:实验所用关键产品包括Qiagen的QIAamp循环核酸试剂盒、miScript II RT Kit、miScript miRNA PCR Arrays、SYBR Green PCR Master Mix,Bio-Rad的iCycler thermocycler和iQ5实时检测器。
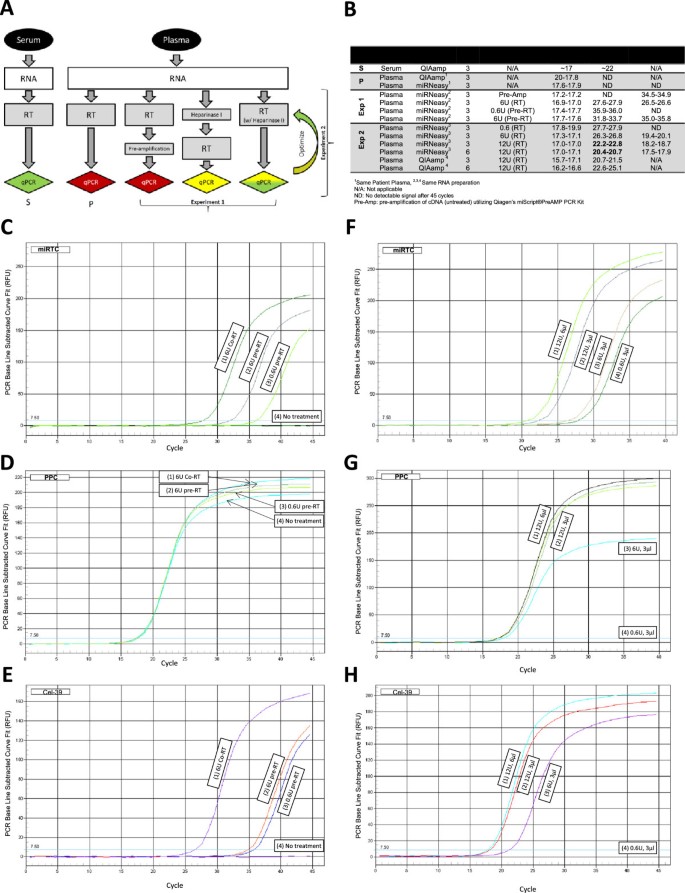
3.2 肝素酶处理条件优化
实验目的是优化肝素酶的处理时间、剂量和作用阶段(提前处理vs反转录中处理)。方法细节:设置三组处理条件:(1)反转录前用0.6U或6U拟杆菌肝素酶I处理RNA 2小时;(2)反转录过程中加入6U或12U拟杆菌肝素酶I(37°C孵育1小时);(3)无处理。检测指标包括miRTC(反转录qPCR对照)、PPC(阳性qPCR对照)、miR-cel-39(spike-in对照)的Ct值。
结果解读:反转录过程中加入12U拟杆菌肝素酶I的效果最优——miRTC的Ct降至约28,PPC的Ct保持在18-20,miR-cel-39的Ct约26.5;而“反转录前处理”即使使用6U肝素酶,miRTC的Ct仍高于30;无处理组无法检测到miRNA。此外,反转录中处理减少了“提前孵育2小时”的步骤,缩短实验时间约1小时,降低了RNA降解风险。
产品关联:实验所用关键产品包括New England BioLabs的拟杆菌肝素酶I。
3.3 改进方法的生物标志物检测验证
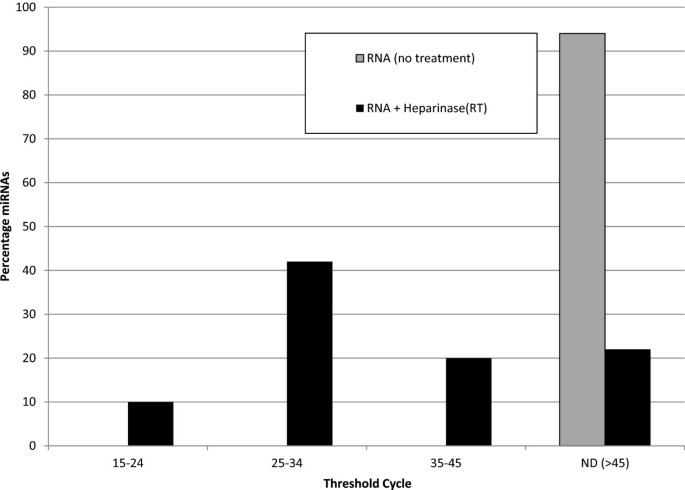
实验目的是验证改进方法对胆管癌(CCA)相关c-miRNA的检测效果。方法细节:选取19份肝素抗凝血浆样本(来自CCA研究队列),提取RNA后分为两组:(1)反转录中加入12U拟杆菌肝素酶I;(2)无处理。用定制miScript miRNA PCR Array(SA Biosciences)检测94个CCA相关的成熟人类miRNA(基于文献报道的CCA生物标志物候选)。
结果解读:无处理组的miRNA Ct均>45(无法检测),而处理组约50%的miRNA Ct≤35,说明改进方法显著提高了候选生物标志物的检测灵敏度。此外,spike-in对照miR-cel-39的Ct在19份样本中均稳定在26-27(n=19,P<0.05),证明方法的重复性良好。
产品关联:实验所用关键产品包括SA Biosciences的定制miScript miRNA PCR Array。
4. Biomarker 研究及发现成果解析
本研究涉及的Biomarker是“血浆中的循环miRNA(尤其是与胆管癌相关的94个成熟人类miRNA)”,其筛选与验证逻辑遵循“已知候选→定制阵列筛选→改进方法验证”的链条。
4.1 Biomarker 定位与筛选逻辑
Biomarker类型:血浆中的循环成熟miRNA(如miR-21、miR-155等,具体取决于定制阵列的设计);筛选逻辑:基于文献报道的“胆管癌相关miRNA”,通过定制miScript miRNA PCR Array(SA Biosciences)进行高通量筛选;验证逻辑:通过“spike-in对照(miR-cel-39)+ 内部对照(miRTC、PPC)+ 候选miRNA检测”,确保方法的特异性与敏感性。
4.2 研究过程详述
Biomarker来源:19份人类肝素抗凝血浆样本(250μl/份),来自George Washington大学的CCA研究队列(样本经去标识化处理,符合伦理要求);验证方法:采用改进的qPCR流程——RNA提取(QIAamp)→反转录(加入12U拟杆菌肝素酶I,37°C孵育1小时)→定制PCR Array检测;特异性与敏感性数据:处理组约50%的候选miRNA Ct≤35(敏感性),而无处理组无法检测(特异性验证);spike-in对照miR-cel-39的Ct在19份样本中均稳定(变异系数<5%),证明方法的重复性。
4.3 核心成果提炼
本研究的核心成果是优化了血浆c-miRNA的qPCR检测方法,其创新性体现在:
1. 技术创新:将拟杆菌肝素酶I整合到反转录过程中,而非提前处理,简化了1个实验步骤(减少2小时孵育),缩短实验时间约30%;
2. 灵敏度提升:处理组比无处理组多检测到约50%的候选miRNA(Ct≤35 vs Ct>45);
3. 应用价值:该方法可直接用于肝素抗凝血浆的c-miRNA检测,为胆管癌等疾病的生物标志物发现提供了高效技术手段。
统计学结果显示:处理组的miRTC(n=19,P<0.01)、miR-cel-39(n=19,P<0.05)的Ct值显著低于无处理组,证明方法的有效性。

