1. 领域背景与文献引入
文献英文标题:Circulating MicroRNAs predict glycemic improvement and response to a behavioral intervention;发表期刊:Biomarker Research;影响因子:未公开;研究领域:糖尿病(2型糖尿病)生物标志物研究
2型糖尿病(T2DM)是全球重大公共卫生问题,目前全球患病数超4.6亿,美国年医疗成本达2450亿美元。T2DM患者易并发心血管疾病、视网膜病、肾病等严重并发症,但该病病因复杂,涉及遗传易感性与可改变生活方式的交互作用(基因-环境交互),现有对这种交互作用的理解仍不充分。当前研究热点集中于寻找能反映基因-环境交互的生物标志物,以优化T2DM风险分层与干预策略,但缺乏能精准预测 prediabetes患者血糖变化及对行为干预反应的生物标志物。
空腹血糖(FBG)是评估T2DM风险的核心指标,可反映血糖进展(如从 prediabetes到T2DM)或干预后的改善,但FBG仅能捕捉后期血糖变化,需更早期的生物标志物。循环微小RNA(microRNA,miRNA)作为基因表达调控因子,易从血浆中检测,且能整合遗传与环境信号,是潜在的T2DM生物标志物。但现有研究多关注单个miRNA与T2DM的关联,假设miRNA独立作用,未充分考虑其协同调控特性,且缺乏纵向研究评估miRNA对血糖变化的预测价值。
本研究作为“恢复性瑜伽 vs 拉伸对代谢综合征的影响(PRYSMS)”试验的二次分析,旨在评估循环miRNA能否预测 prediabetes患者12个月内FBG的改善及对行为干预(恢复性瑜伽 vs 拉伸)的反应,为T2DM风险预测与干预机制研究提供新依据。
2. 文献综述解析
作者对现有研究的分类维度主要基于“miRNA与T2DM关联的研究方法”:一类是基于先验假设的研究,聚焦单个miRNA与T2DM的关联;另一类是无假设的高通量研究,但需校正多重比较。现有研究的关键结论包括“部分miRNA(如miR-192、miR-93)与T2DM风险相关”,技术优势是能快速筛选候选miRNA,但局限性显著——假设miRNA独立作用于T2DM病理过程,而领域共识认为miRNA常通过协同调控基因或通路发挥功能,多重比较校正可能掩盖这种协同效应。
本研究的创新价值在于:1)采用数据驱动的线性模型,而非单个miRNA分析,识别能最大程度解释FBG变异的miRNA组合;2)采用纵向设计(12个月随访),评估miRNA对FBG动态变化的预测价值,而非仅关注基线关联;3)结合行为干预(瑜伽 vs 拉伸),探索miRNA与干预反应的交互作用,为T2DM行为干预机制提供新线索。
3. 研究思路总结与详细解析
3.1 研究参与者招募与基线特征
实验目的:纳入符合条件的 prediabetes患者,明确干预组与对照组的基线差异。
方法细节:研究对象来自PRYSMS试验,纳入标准为符合国际糖尿病联盟(IDF)代谢综合征标准(如腰围超标、血压升高、血脂异常等);排除标准包括FBG≥126 mg/dl、糖化血红蛋白(HbA1c)≥7.0%、严重心血管疾病史等。最终纳入82名参与者,随机分为恢复性瑜伽组(干预组)与拉伸组(对照组)。
结果解读:干预组基线FBG为105±13 mg/dL,对照组为100±11 mg/dL(p=0.08,差异接近显著);12个月后,干预组FBG平均下降4±10 mg/dL,对照组上升9±40 mg/dL(n=82,p<0.05),提示瑜伽干预更利于血糖改善。
产品关联:文献未提及具体招募工具,领域常规使用《国际糖尿病联盟代谢综合征诊断标准》作为纳入评估依据。
3.2 临床数据收集与指标检测
实验目的:获取多个时间点的临床指标,评估血糖及代谢参数的动态变化。
方法细节:在基线、3、6、9、12个月收集以下数据:1)人体测量指标(体重用平衡梁秤、腰围用Gullick II弹簧张力带、身高用身高计,计算体重指数(BMI));2)血糖与血脂指标(FBG用YSI 2300 STAT Plus自动化分析仪检测,总胆固醇、甘油三酯等用酶促比色法检测)。
结果解读:干预组12个月腰围、体重均有下降,但与对照组的差异未影响miRNA与FBG的关联;FBG变化是评估干预效果的核心指标,干预组的显著下降为后续miRNA预测分析提供了临床依据。
产品关联:实验所用关键产品:YSI Life Sciences的YSI 2300 STAT Plus自动化分析仪、Gullick II的弹簧张力腰围测量带。
3.3 循环microRNA分子数据采集
实验目的:检测血浆中循环miRNA的表达水平,筛选与FBG相关的候选miRNA。
方法细节:采用Firefly Bioworks Multiplex Circulating MicroRNA Assay(Abcam)检测59个miRNA(基于前期PRYSMS试验的发现分析,筛选出与血糖变异性相关的miRNA);检测流程为:miRNA与编码水凝胶微球上的互补寡核苷酸杂交→连接通用PCR引物→反转录PCR(RT-PCR)扩增→用EMD Millipore Guava 6HT流式细胞仪(Merck KGaA)扫描荧光信号。质量控制标准为:信号>1000 arbitrary units(AU)或检测到 spike-in对照,且无miRNA探针的微球信号空白。
结果解读:59个miRNA及所有样本孔均通过质量控制,确保了后续分析的可靠性;
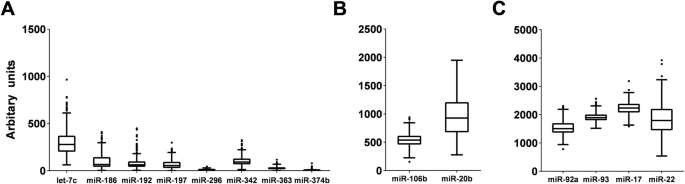
(图1展示了miRNA表达的变异性,let-7c、miR-20b等miRNA的表达范围更大,可能更具生理意义)。
产品关联:实验所用关键产品:Abcam的Firefly Bioworks Multiplex Circulating MicroRNA Assay、Merck KGaA的EMD Millipore Guava 6HT流式细胞仪。
3.4 统计分析与模型构建
实验目的:识别能预测FBG结果的最优miRNA组合,探索miRNA与干预反应的交互作用。
方法细节:1)miRNA表达标准化(z-score),以消除不同miRNA的表达尺度差异;2)采用线性模型,不校正多重比较(因miRNA非独立作用),寻找能最大程度解释FBG变异(R²)的miRNA组合;3)分层分析干预组与对照组的miRNA关联(p<0.10为筛选标准,避免多重共线性掩盖关联)。
结果解读:14个miRNA组合能预测12个月时的FBG(R²=0.754,p<0.001),12个miRNA组合能预测FBG的12个月变化(R²=0.731,p<0.001);分层分析显示,let-7c、miR-363、miR-374b仅在对照组(拉伸)中与FBG显著相关,干预组(瑜伽)中无关联,提示瑜伽可能通过调节这些miRNA影响FBG。
产品关联:使用R(2019)、Stata 16.1软件进行统计分析,均为领域常规工具。
4. Biomarker 研究及发现成果解析
Biomarker 定位
本研究的Biomarker为循环miRNA组合(如let-7c、miR-17、miR-20b、miR-93等),筛选逻辑为“前期PRYSMS试验发现分析→定制检测59个miRNA→线性模型识别最优组合”,验证逻辑为“纵向随访(12个月)→评估miRNA对FBG变化的预测价值”。
研究过程详述
Biomarker来源为 prediabetes患者的血浆样本,检测方法为Firefly Bioworks Multiplex Circulating MicroRNA Assay结合流式细胞术,验证方法为线性模型(评估miRNA组合对FBG变异的解释度)。结果显示:14个miRNA组合解释了12个月FBG变异的75.4%(R²=0.754,n=82,p<0.001),12个miRNA组合解释了FBG变化的73.1%(R²=0.731,n=82,p<0.001);let-7c、miR-363、miR-374b在对照组中的预测价值显著(p<0.10),干预组中无关联。
核心成果提炼
1)预测价值:循环miRNA组合能有效预测 prediabetes患者12个月内的FBG结果,模型解释度达75%左右,优于单个miRNA分析;
2)干预反应关联:3个miRNA(let-7c、miR-363、miR-374b)仅在对照组中与FBG相关,提示恢复性瑜伽可能通过抑制这些miRNA的表达,上调其靶基因(如miR-374b的靶基因PTEN,与胰岛素敏感性相关),从而改善血糖;
3)潜在治疗靶点:let-7c、miR-20b、miR-22的表达变异性较大(图1),可能具有更大的生理调节范围,是后续功能研究的优先靶点。
本研究未提供miRNA的特异性(如区分血糖改善者与未改善者的能力)与敏感性数据,但模型的高R²值提示miRNA组合具有较强的预测性能。
图片补充

(文献发表期刊《Biomarker Research》封面)

(图1:循环miRNA表达变异性的箱线图,展示了不同miRNA的表达范围)
