1. 领域背景与文献引入
文献英文标题:Development and validation of a predictive model based upon extracellular vesicle-derived transposable elements for non-invasive detection of pancreatic adenocarcinoma;发表期刊:Biomarker Research;影响因子:未公开;研究领域:胰腺癌无创诊断生物标志物研究。
胰腺癌(Pancreatic adenocarcinoma, PAAD)是全球范围内致死率最高的恶性肿瘤之一,约占所有癌症死亡病例的5%,且90%患者确诊时已处于晚期,5年生存率不足13%。当前临床常用的辅助诊断标志物碳水抗原19-9(Carbohydrate antigen 19-9, CA19-9)因特异性和敏感性有限,无法满足早期诊断需求,亟需开发新型无创诊断生物标志物。细胞外囊泡(Extracellular vesicles, EVs)作为细胞间通讯的重要载体,可携带亲本细胞的生物分子并在体液中保持稳定,为癌症无创诊断提供了理想的技术载体;转座元件(Transposable elements, TEs)曾被视为“垃圾DNA”,近年研究发现其参与癌症发生发展,且富集于无细胞转录组及癌症来源EVs中。然而,EV来源TEs(EV-TEs)作为胰腺癌无创诊断标志物的潜力尚未被系统探索,这一研究空白成为领域亟需解决的关键问题。本研究针对胰腺癌早期诊断标志物缺乏的痛点,首次开发基于EV-TEs的预测模型,填补了EV-TEs在胰腺癌诊断中的研究空白,为癌症诊断提供了新型“垃圾DNA”来源生物标志物。
2. 文献综述解析
文献综述按“现有生物标志物局限性→EVs的技术优势→TEs的生物学意义”三维度展开评述。现有研究结论显示:CA19-9是临床最常用的胰腺癌辅助诊断标志物,但在良性胰腺疾病(如慢性胰腺炎)中也会升高,特异性不足(75.4%-79.7%),且早期胰腺癌患者中敏感性仅73.4%-77.4%;EVs因能传递亲本细胞的遗传信息、在体液中稳定性高,被视为无创诊断的“理想载体”;TEs虽曾被忽视,但其在癌症细胞中异常激活的特性,使其成为潜在的癌症生物标志物来源。现有研究的优势在于明确了EVs的技术可行性与TEs的癌症相关性,为生物标志物开发提供了理论基础;局限性则是尚未将EVs与TEs结合,探索其在胰腺癌诊断中的价值。本研究的创新点在于首次将EV-TEs作为胰腺癌诊断标志物,通过机器学习模型验证其性能,突破了传统生物标志物的局限,为“垃圾DNA”向临床有用生物标志物的转化提供了实证。
3. 研究思路总结与详细解析
本研究的核心目标是开发基于EV-TEs的胰腺癌无创检测预测模型,核心科学问题是“EV-TEs能否作为胰腺癌的高特异性、高敏感性无创诊断标志物”,技术路线遵循“样本收集→EV-TEs定量→特征筛选→模型构建→多队列验证”的闭环逻辑。
3.1 队列样本收集与EV转录组测序
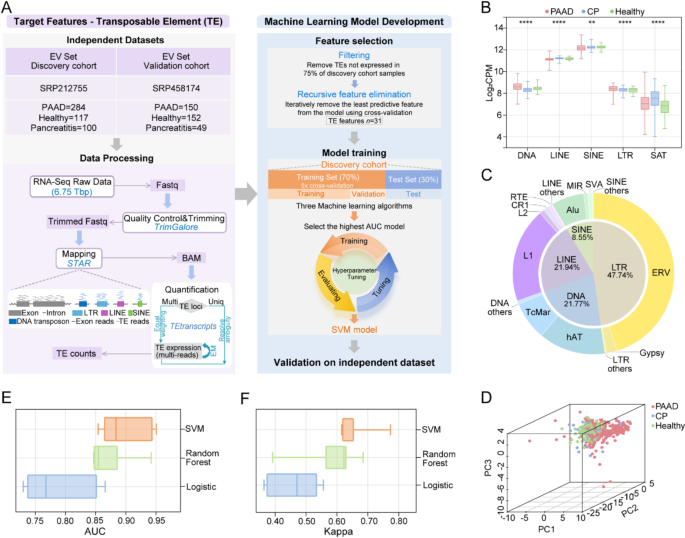
实验目的是获取胰腺癌患者、慢性胰腺炎患者及健康对照的EV来源转录组数据,为后续分析提供基础。方法细节:研究纳入“发现队列”(284例胰腺癌、100例慢性胰腺炎、117例健康对照)和“外部验证队列”(150例胰腺癌、49例慢性胰腺炎、152例健康对照),共852份EV来源转录组样本,进行总数据量6.75 Tbp的转录组测序,并对每个TE的表达水平进行定量。结果解读:不同类型重复序列(如DNA转座子、长末端重复序列)在胰腺癌与对照组中的表达水平存在显著差异(图1B);因部分TEs在多数样本中无表达,过滤后保留620种具有差异表达潜力的TEs(图1C)。
产品关联:文献未提及具体实验产品,领域常规使用EV分离试剂盒(如Thermo Fisher Total Exosome Isolation Kit)与转录组测序试剂盒(如Illumina TruSeq Stranded mRNA Library Prep Kit)完成样本处理与测序。
3.2 EV-TEs特征筛选与生物标志物 panel 构建
实验目的是从620种TEs中筛选出最优特征组合,构建胰腺癌诊断的EV-TEs panel。方法细节:在发现队列中采用递归特征消除法(Recursive Feature Elimination),逐步移除贡献较小的TEs,最终保留31个EV-TEs特征。结果解读:31个特征中,HERV1_I-int(结肠腺癌中激活)、LTR48B(多肿瘤类型中高表达)等已被报道与癌症相关;特征间相关性分析显示多数TEs无显著关联(图S1B),主成分分析进一步验证该panel能有效区分胰腺癌、慢性胰腺炎与健康对照(图1D),说明其具有良好的判别能力。
产品关联:文献未提及具体实验产品,领域常规使用R语言caret包完成特征筛选。
3.3 机器学习模型构建与多队列验证
实验目的是验证EV-TEs panel的诊断性能,筛选最优预测模型。方法细节:将发现队列按7:3比例随机分为训练集与测试集,采用支持向量机(Support Vector Machine, SVM)、逻辑回归(Logistic Regression, LR)、随机森林(Random Forest, RF)三种算法构建模型,通过ROC曲线(Area Under the Curve, AUC)、混淆矩阵评估性能,并在外部独立队列中验证。结果解读:SVM模型表现最优——训练集AUC达0.90(95%CI 0.86-0.93),胰腺癌患者真阳性率82.1%(n=39),健康对照真阴性率96.8%(n=31);测试集AUC为0.86(95%CI 0.79-0.92),真阳性率82.4%(n=85),真阴性率83.3%(n=66);外部验证集AUC进一步提升至0.88(95%CI 0.84-0.92),真阳性率83.3%(n=150),真阴性率82.6%(n=201)(图2A-I)。

产品关联:文献未提及具体实验产品,领域常规使用scikit-learn库完成模型构建。
4. Biomarker 研究及发现成果解析
Biomarker 定位与筛选逻辑
本研究的Biomarker类型为血浆EV来源转座元件(EV-TEs),筛选逻辑遵循“队列筛选→模型验证→外部验证”闭环:首先在发现队列中通过递归特征消除法筛选31个EV-TEs特征,随后在训练集、测试集验证其判别能力,最终在外部队列中确认性能。
研究过程与性能数据
Biomarker来源为血浆EV转录组,验证方法包括转录组测序定量TEs表达、机器学习模型评估诊断性能。核心性能数据显示:该EV-TEs panel在训练集的AUC为0.90(95%CI 0.86-0.93),测试集0.86(95%CI 0.79-0.92),外部验证集0.88(95%CI 0.84-0.92);混淆矩阵结果显示,三个队列中胰腺癌患者真阳性率均超过82%,健康对照真阴性率超过82%,显著优于临床常用的CA19-9(敏感性73.4%-77.4%、特异性75.4%-79.7%)。
核心成果与创新价值
本研究的核心成果是首次开发基于EV-TEs的胰腺癌无创诊断预测模型,其创新点在于:① 将“垃圾DNA”(TEs)与EVs技术结合,拓展了生物标志物的来源;② 验证了EV-TEs作为胰腺癌无创诊断标志物的潜力,解决了CA19-9特异性不足的问题;③ 多队列验证确保了模型的可靠性,为后续临床转化提供了基础。研究同时指出局限性:胰腺癌组年龄显著高于对照组(符合临床特征,但可能引入混杂因素)、缺乏患者分期数据(无法评估模型对早期胰腺癌的性能),需大样本、多中心研究进一步验证。
本研究通过系统解析EV-TEs的诊断价值,为胰腺癌无创诊断提供了新型生物标志物,也为“垃圾DNA”的临床应用提供了重要范例。
