1. 领域背景与文献引入
文献英文标题:singleCellBase: a high-quality manually curated database of cell markers for single cell annotation across multiple species;发表期刊:Biomarker Research;影响因子:未公开;研究领域:单细胞RNA测序(scRNA-seq)数据细胞注释及细胞标志物数据库构建。
单细胞RNA测序技术的普及推动了细胞异质性的深入研究,但scRNA-seq数据的细胞注释仍是研究者面临的核心挑战之一。手动细胞注释被广泛认为是细胞注释的金标准,但其劳动强度大且高度依赖对“细胞类型-基因标志物”关联的先验知识。目前,已有PanglaoDB、CellMarker v2.0、SingleR等数据库或工具为细胞注释提供支持,其中SingleR等工具基于参考数据集实现自动注释,提升了分析效率,但这些资源的共同局限性在于物种覆盖范围狭窄——主要聚焦于人类和小鼠等模式物种,无法满足非模式动物、植物等多物种scRNA-seq数据的分析需求。针对这一研究空白,本研究构建了singleCellBase数据库,旨在整合多物种的高质量细胞标志物数据,为手动细胞注释提供关键先验知识,解决现有资源“物种覆盖不足”的核心问题。
2. 文献综述解析
文献综述部分以“现有数据库的物种覆盖范围、数据质量、功能局限性”为核心维度,对领域内研究进行评述。现有研究的关键结论包括:手动细胞注释虽为金标准,但存在“耗时耗力、依赖先验知识”的局限性;现有数据库如PanglaoDB、CellMarker v2.0提供了有价值的细胞标志物资源,但物种覆盖集中于人类和小鼠;SingleR等自动注释工具依赖参考数据集,其应用范围受限于参考数据的物种限制。现有研究的技术优势在于“提升了注释效率”,但局限性也同样明显——无法支持多物种scRNA-seq数据的细胞注释。
与现有研究相比,本研究的创新价值体现在三个方面:一是物种覆盖更广泛,首次整合了31个物种的细胞标志物数据(涵盖动物界、原生生物界、植物界,包括人类、小鼠、鱼类、鸡、猴、猪、拟南芥等);二是数据质量更可靠,数据来源为10x Genomics网站的权威curated文献,经过“文献筛选-数据提取-双重核查”的严格流程,确保了数据的准确性;三是功能模块更全面,数据库整合了“浏览、搜索、可视化、下载、提交”五大功能,为研究者提供了“从数据查询到结果验证”的全流程支持。
3. 研究思路总结与详细解析
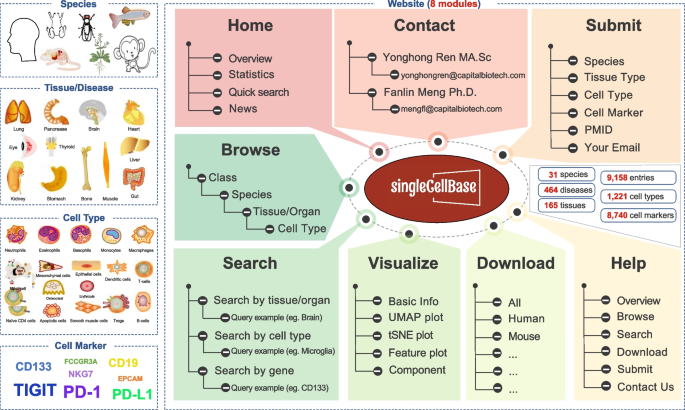
本研究的整体框架为:以“解决现有数据库物种覆盖不足”为目标,围绕“多物种细胞标志物数据整合”这一核心科学问题,采用“文献收集-数据提取-标准化处理-数据库构建-功能验证”的闭环技术路线,最终构建了singleCellBase数据库。
3.1 文献来源与数据收集
该环节的核心目标是获取高质量、多物种的细胞类型-基因标志物关联数据。研究团队选择10x Genomics网站的curated publications作为主要数据来源(原因在于10x Genomics平台的文献具有较高的质量和相关性),具体流程为:首先通过阅读文献摘要筛选出与“细胞类型-标志物关联”相关的文献,去除无关研究;随后手动阅读全文及补充表格,提取细胞类型、基因标志物、组织/器官、疾病/状态等关联信息;最后对提取的数据进行双重核查,确保信息准确。结果显示,研究共收集到9158条有效条目,覆盖31个物种、1221种细胞类型、8740个基因标志物、464种疾病/状态及165种组织类型。
文献未提及具体实验产品,领域常规使用EndNote等文献管理工具、Excel等数据表格工具进行文献筛选与数据提取。
3.2 数据标准化与条目构建
实验目的是统一不同来源数据的命名规则,解决“同一细胞类型/标志物在不同文献中表述不一致”的问题。研究团队针对细胞类型、组织/器官、疾病名称进行了标准化处理(例如将“CD8+ T细胞”“CD8 T细胞”统一为“CD8 T细胞”),并为每条条目补充了“关联置信度、PubMed ID、期刊名称、相关数据集、证据描述”等详细信息。结果构建了结构化、标准化的数据库条目,确保了不同文献数据的一致性与可用性。
文献未提及具体实验产品,领域常规使用基因本体(GO)、细胞本体(CL)等Ontology数据库进行术语标准化。
3.3 数据库功能模块开发
核心目标是为用户提供友好的交互界面,满足“数据查询、结果验证、贡献数据”的需求。研究开发了五大功能模块:“浏览”模块支持按“物种-组织-细胞类型”的分类系统浏览数据;“搜索”模块允许用户按“组织类型、细胞类型、基因标志物”检索(例如检索“人类CD8A”可得到“CD8 T细胞”的关联);“可视化”模块可展示基因在scRNA-seq数据中的表达模式(如UMAP/t-SNE图、Feature plot、细胞组成柱状图);“下载”模块提供文本格式的数据下载;“提交”模块支持用户贡献新的细胞类型-标志物关联数据。结果显示,各模块功能稳定,例如“搜索”模块能快速返回高置信度的关联结果,“可视化”模块能清晰展示基因的细胞特异性表达。
文献未提及具体实验产品,领域常规使用MySQL数据库管理系统、Python Flask框架等工具构建web界面。

3.4 数据库功能验证
该环节旨在验证数据库的实用性与数据准确性。研究团队以Zheng等的胰腺癌细胞研究(该研究用流式细胞术分选了“CD45-EPCAM+上皮细胞”)为例,利用“可视化”模块验证EPCAM的表达模式:选择Zheng等的scRNA-seq数据集,输入“EPCAM”基因,生成UMAP/t-SNE图、Feature plot及细胞组成柱状图。结果显示,EPCAM在“CD45-EPCAM+上皮细胞”中特异性高表达,与Zheng等的实验结果一致,验证了数据库中“EPCAM为上皮细胞标志物”的准确性。
文献未提及具体实验产品,领域常规使用Seurat、Scanpy等scRNA-seq分析工具进行可视化。
4. Biomarker研究及发现成果解析
本研究中的生物标志物(Biomarker)为“不同物种、不同细胞类型的基因标志物”,其筛选与验证逻辑为:从10x Genomics网站的curated文献中提取经过实验验证的细胞类型-基因标志物关联,通过“双重核查”确保数据准确性,并对术语进行标准化处理。
研究过程中,Biomarker的来源为10x Genomics网站curated文献中的实验数据(如scRNA-seq鉴定的标志物、流式细胞术验证的标志物);验证方法包括“文献中的实验验证”(如Zheng等用流式分选验证EPCAM)及“本数据库可视化模块的再次验证”(如EPCAM在胰腺癌细胞上皮细胞中的表达)。以人类“CD8A”为例,其特异性关联“CD8 T细胞”(文献中高频报道,数据库中频率分析显示高置信度);以“EPCAM”为例,其在胰腺癌细胞上皮细胞中的敏感性与特异性通过“流式分选”(Zheng等研究)及“可视化模块”(本研究)得到确认。
核心成果方面,数据库共包含9158条Biomarker-细胞类型关联数据,覆盖31个物种。其中,人类“CD8A”与“CD8 T细胞”、“EPCAM”与“上皮细胞”,小鼠“CD4”与“辅助T细胞”等关联均经过文献验证,具有较高的置信度。研究的创新性在于:首次整合了多物种(包括非模式动物、植物)的Biomarker数据,解决了现有数据库“物种覆盖不足”的问题;数据经过严格的人工审核(双重核查),确保了准确性;数据库的“可视化”模块为Biomarker的功能验证提供了便捷工具。例如,数据库中拟南芥的细胞标志物数据为植物scRNA-seq分析提供了关键先验知识,这是现有数据库未覆盖的领域。
综上,singleCellBase数据库为多物种scRNA-seq数据的细胞注释提供了高质量的Biomarker资源,有望推动“跨物种细胞类型比较”“非模式生物细胞异质性研究”等领域的进展。
