1. 领域背景与文献引入
文献英文标题:Redefining prostate cancer risk stratification: a pioneering strategy to estimate outcome based on Ki67 immunoscoring;发表期刊:Biomarker Research;影响因子:未公开;研究领域:前列腺癌预后生物标志物与风险分层。
前列腺癌(PCa)是男性最常见的恶性肿瘤之一,约90%患者初诊为局限性病变。对于低危(Grade Group, GG1)和有利中危(GG2)患者,因疾病惰性常推荐主动监测(Active Surveillance, AS)以避免过度治疗。然而,临床决策仍面临两大挑战:一是约20-30%的AS患者会出现生化复发(Biochemical Recurrence, BCR)或疾病进展,而传统风险评估(如Gleason评分、PSA、临床分期)无法精准识别这部分高风险人群;二是现有预后工具的局限性——Decipher®、Oncotype DX®等多基因panel对AS患者的进展预测效果有限(AUC约0.65-0.68),Prolaris®虽能预测复发(AUC0.825)但成本高昂(约3000美元/次),难以在基层医院推广。因此,亟需开发低成本、易实施、针对AS患者核心需求(预测BCR)的预后工具,整合临床参数与可及的生物标志物,改善风险分层准确性。本研究旨在探索Ki67免疫评分(增殖标志物)、GSTP1/KLF8启动子甲基化与临床参数的联合预后价值,构建可视化风险计算器(nomogram),为AS患者的治疗决策提供依据。
2. 文献综述解析
作者对现有研究的评述围绕“前列腺癌风险分层的挑战”“生物标志物的应用局限”“现有工具的不足”三类展开。首先,临床层面:低危/有利中危患者的AS决策是临床难点,因部分患者虽初始分级低但仍会进展,而传统风险评估无法精准识别这部分人群;其次,生物标志物研究:Ki67作为细胞增殖的金标准标记物,在前列腺癌中的预后价值已被多项meta分析证实(如关联更高Gleason评分、更差BCR-free生存),但未纳入常规临床实践;GSTP1启动子甲基化是前列腺癌常用的诊断标志物(敏感度约90%),但预后价值有限(仅在Ⅲ期与Ⅱ期患者中有差异);KLF8甲基化与肿瘤分化的关系研究较少,其预后意义尚不明确;最后,现有 genomic 工具的局限:Decipher®主要预测不良病理特征,对AS患者的进展预测效果有限;Oncotype DX®的AUC仅0.68,无法有效区分AS患者的风险;Prolaris®依赖多基因表达分析,成本高且需特定检测平台,难以在基层医院推广。
本研究的创新在于摒弃多基因panel,选择临床可及的Ki67免疫评分,结合常规临床参数(年龄、PSA、临床分期)构建风险计算器,解决了现有工具“高成本、低可及性”的痛点;同时针对AS患者的核心需求(预测BCR)优化模型,而非泛泛预测“不良病理”,提升了临床实用性。
3. 研究思路总结与详细解析
整体研究目标是构建并验证基于Ki67免疫评分与临床参数的前列腺癌预后风险计算器,核心科学问题是“Ki67联合临床参数是否能更准确预测AS患者的生化复发风险”。技术路线遵循“生物标志物筛选→临床关联分析→模型构建→独立验证”的闭环:首先在前列腺癌切除标本中检测Ki67免疫评分、GSTP1me、KLF8me水平,关联总生存(OS)与BCR-free生存终点;随后纳入年龄、临床分期、PSA等临床参数,构建死亡风险与BCR风险的nomogram;最后在独立前列腺活检队列中验证模型的预后价值。
3.1 生物标志物检测与临床终点关联分析
实验目的是评估Ki67、GSTP1me、KLF8me的预后价值。方法细节:在前列腺癌组织标本中,通过免疫组化(IHC)检测Ki67的表达(阳性细胞比例评分:<5%为0分,5-10%为1分,>10%为2分),通过甲基化特异性PCR(文献未明确具体方法)检测GSTP1与KLF8启动子甲基化水平;临床终点为OS(总生存)与BCR-free生存(定义为血清PSA≥0.2ng/mL且持续升高)。结果解读:Ki67评分越高,BCR-free生存越差(Ki67≥5%患者的BCR风险是<5%患者的1.69倍,P<0.05);KLF8me水平越高,OS越好(低KLF8me患者的死亡风险是高KLF8me患者的1.76倍,P<0.05),尤其在低级别肿瘤(GG1/2)中更显著;GSTP1me水平与OS或BCR-free生存无显著关联。实验所用关键产品:文献未提及具体实验产品,领域常规使用Ki67抗体(如Dako的MIB-1抗体)、甲基化检测试剂盒(如Zymo Research的EpiTect Bisulfite Kit)。
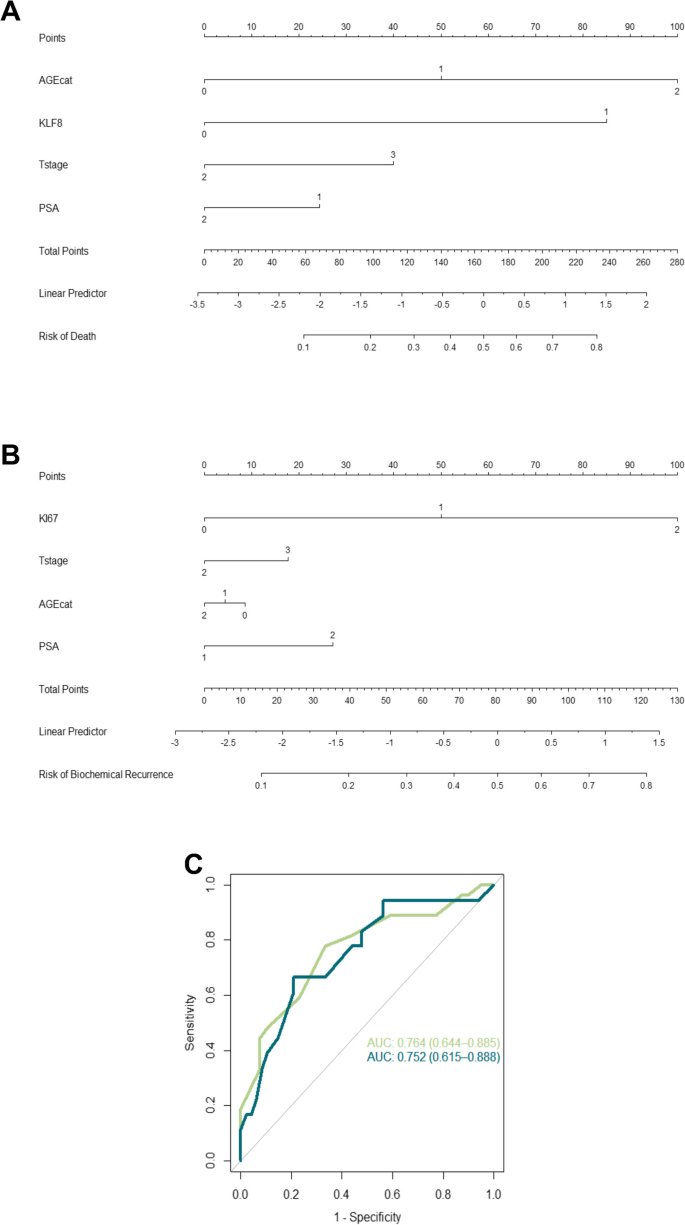
3.2 风险计算器构建与独立队列验证
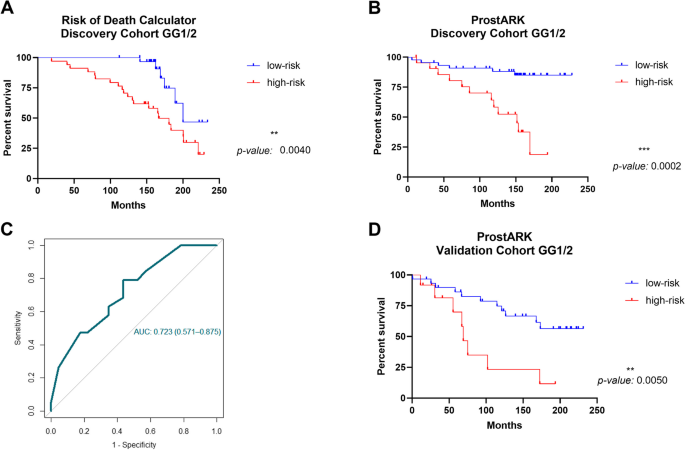
实验目的是整合生物标志物与临床参数,构建可实用的预后模型。方法细节:基于发现队列(120例前列腺癌切除标本),纳入Ki67评分、年龄(分层为≤55岁、55-65岁、>65岁)、临床分期(Tstage)、PSA水平(<10ng/mL vs >10ng/mL),通过Cox比例风险模型构建2个nomogram:①死亡风险计算器(包含KLF8me、年龄、Tstage、PSA);②BCR风险计算器(命名为ProstARK,包含Ki67、年龄、Tstage、PSA)。随后在独立验证队列(50例前列腺活检标本)中评估模型性能,主要指标为ROC曲线下面积(AUC)、敏感性、特异性、阳性预测值(PPV)、阴性预测值(NPV)。结果解读:ProstARK在发现队列中表现最优:特异性79.17%、敏感性66.67%、PPV55%、NPV86%、准确率75.76%(n=120,P<0.05);在验证队列中,ProstARK的AUC为0.723(95%CI 0.571-0.875,n=50,P<0.05),且能显著区分低风险与高风险患者的BCR-free生存(高风险组的BCR风险是低风险组的2.3倍,P<0.05)。实验所用关键产品:文献未提及具体实验产品,领域常规使用nomogram构建软件(如R语言的rms包)。
4. Biomarker 研究及发现成果解析
本研究涉及的Biomarker包括Ki67免疫评分(蛋白水平)、GSTP1启动子甲基化(GSTP1me)、KLF8启动子甲基化(KLF8me),其中Ki67是核心预后Biomarker。筛选逻辑遵循“临床相关性→预后价值”:首先选择与前列腺癌增殖(Ki67)、肿瘤发生(GSTP1甲基化沉默)、分化(KLF8甲基化)相关的标志物,再通过临床队列验证其与生存终点的关联。
4.1 检测与验证过程
- 来源:前列腺癌切除标本(发现队列,n=120)与活检标本(验证队列,n=50);
- 方法:Ki67采用免疫组化定量评分(阳性细胞比例:<5%=0分,5-10%=1分,>10%=2分);GSTP1me/KLF8me采用甲基化特异性PCR检测(甲基化水平分层为“高”“低”);
- 性能数据:Ki67评分的BCR预测敏感性66.67%、特异性79.17%(发现队列);KLF8me的OS预测HR=1.76(低vs高甲基化,P<0.05);GSTP1me无显著预后价值。
4.2 核心成果
- Ki67是前列腺癌BCR的独立预后因素:Ki67评分每升高1分(如从0分到1分),BCR风险增加69%(HR=1.69,P<0.05);Ki67≥5%患者的BCR-free生存时间较<5%患者缩短约2年(中位生存:5.2年vs 7.4年,P<0.05)。
- ProstARK的高NPV是关键优势:ProstARK的NPV达86%,意味着低风险患者的BCR概率仅14%,可安全选择AS;而高风险患者(ProstARK评分> -0.82)的BCR概率达31%,需考虑主动治疗(如前列腺癌根治术)。
- KLF8me是低级别肿瘤的OS预后因素:在GG1/2患者中,低KLF8me患者的OS时间较髙KLF8me患者缩短约3年(中位生存:8.1年vs 11.3年,HR=1.75,P<0.05),但对BCR无预测价值。
本研究的创新性在于首次将Ki67免疫评分整合到针对AS患者的nomogram中,解决了现有工具“高成本、低可及性”的问题——ProstARK仅需常规免疫组化设备(病理科标配),检测成本约50美元/次,远低于genomic测试;且其NPV(86%)优于部分genomic测试(如Decipher®的AUC0.65),更适合AS患者的风险分层。
总结
本研究通过整合Ki67免疫评分与临床参数,构建了针对AS患者的BCR风险计算器(ProstARK),具有低成本、高可及性、高NPV的优势,为临床医生提供了“可视化、易操作”的预后工具。未来需在更大样本的多中心活检队列中验证其性能,同时可探索将KLF8me整合到OS风险模型中,进一步完善前列腺癌的全程管理。
