1. 领域背景与文献引入
文献英文标题:Epigenetic signatures on plasma cell-free DNA to detect kidney allograft rejection in a non-invasive way: development of a 10-plex digital PCR assay;发表期刊:Biomarker Research;影响因子:未公开;研究领域:肾移植排斥反应的无创诊断生物标志物研究。
肾移植是终末期肾病的最佳治疗方式,但随访管理仍面临关键挑战:当前标准护理策略为“无创但非特异性的生物标志物(如血清肌酐、供体特异性抗体(DSA))+肾活检(组织学金标准)”。其中,肌酐易受脱水、药物等因素干扰,无法特异区分排斥与其他肾功能异常;DSA虽与排斥相关,但约30%~40%的阳性患者无临床排斥表现。肾活检虽能明确诊断,却存在取样偏差(仅覆盖肾组织的1/1000)、出血/感染并发症(发生率约5%~10%)及高成本(单例活检费用超万元)等局限性。因此,寻找兼具无创性与特异性的排斥诊断方法,是肾移植领域未解决的核心问题。
文献的研究初衷即针对这一痛点:作者假设血浆游离DNA(cfDNA)的表观遗传特征(甲基化模式)可反映肾组织细胞的损伤——cfDNA来自死亡/受损细胞的释放,其甲基化模式与来源细胞一致;若能筛选出肾组织特异性的甲基化标志物,即可通过检测血浆cfDNA中的这些标志物,无创且特异识别肾移植排斥导致的肾细胞死亡,从而替代或补充活检。
2. 文献综述解析
作者的评述逻辑围绕“现有策略局限性→新技术潜力→肾移植领域空白”展开:首先,系统梳理了肾移植随访的现有困境——无创标志物非特异、活检 invasiveness 高;随后,拓展了cfDNA甲基化的研究背景:该技术在癌症中已用于检测循环肿瘤DNA(通过肿瘤细胞的甲基化特征),但肾移植领域尚无研究;最后,归纳现有研究的核心结论:①肌酐、DSA等无创标志物无法单独诊断排斥;②活检是金标准但受限;③cfDNA甲基化具有无创、细胞特异性的潜力。
现有研究的技术优势在于cfDNA分析的无创性,可反复采样;局限性则是缺乏肾组织特异性标志物,无法直接应用于肾移植。文献的创新价值在于首次将cfDNA甲基化技术引入肾移植领域:针对肾组织的关键细胞类型(肾小球/肾小管的上皮、内皮细胞)筛选特异性甲基化标志物,开发10重数字PCR(dPCR) assay,实现排斥反应的无创诊断——既解决了现有无创标志物的特异性问题,又避免了活检的风险。
3. 研究思路总结与详细解析
整体框架:研究目标是开发“无创+特异”的肾移植排斥诊断工具;核心科学问题是“血浆cfDNA中的肾特异性甲基化标志物能否准确预测排斥反应”;技术路线为“in silico筛选标志物→assay开发验证→临床队列验证”的闭环。
3.1 肾特异性甲基化标志物的in silico筛选
实验目的:识别肾小球与肾小管中上皮/内皮细胞的特异性甲基化特征,确保标志物仅来源于肾组织(而非血细胞等cfDNA污染物)。
方法细节:重新分析GEO数据库(GSE186458)中21种人类组织/细胞的甲基化组数据(每种样本重复3次),从1567万余个CpG岛中,筛选“肾组织高甲基化、血细胞低甲基化”的标志物。
结果解读:最终筛选出9种肾特异性标志物,按细胞类型分为三类:①全肾标志物(CTDP1,覆盖上皮与内皮细胞);②肾内皮标志物(ARID3A、GATA2、LOC124903692等7种);③肾上皮标志物(PAX2、ACSL5)。这些标志物在血细胞中的甲基化率极低(避免干扰),在肾组织中的甲基化率显著高于肝、肺等其他组织。
产品关联:文献未提及具体实验产品,领域常规使用R语言的methylKit、minfi包分析甲基化组数据。
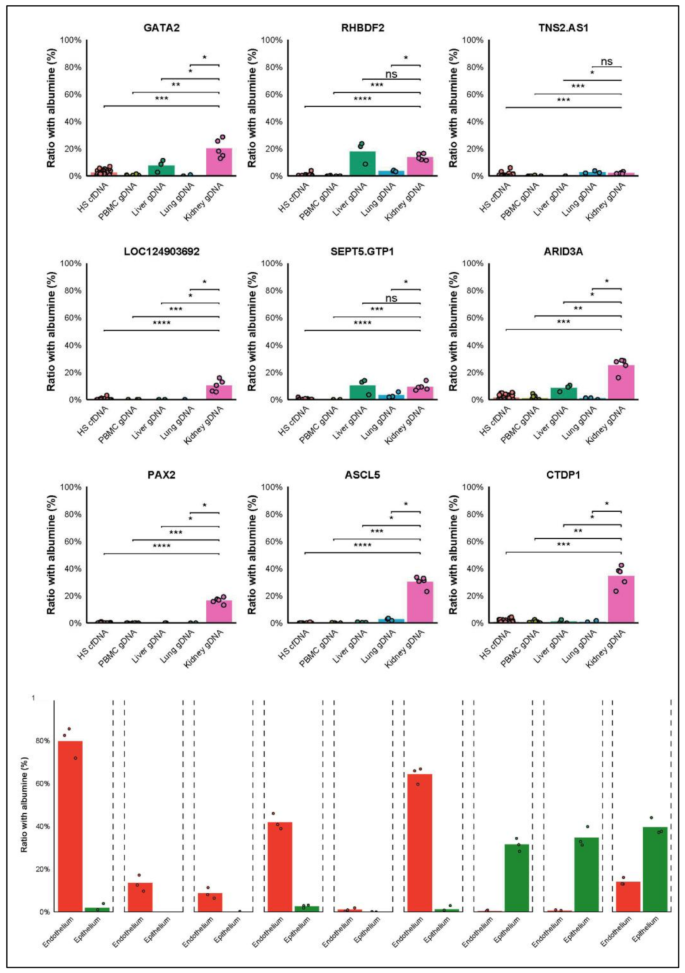
(图1展示了各标志物在不同组织/细胞中的相对定量结果,验证了肾特异性)
3.2 甲基化特异性数字PCR assay的开发与验证
实验目的:设计9种标志物的dPCR assay,验证其特异性、重复性与灵敏度,构建10重dPCR panel(含白蛋白内参,校正cfDNA输入量)。
方法细节:针对每种标志物设计甲基化特异性引物/探针(仅结合甲基化DNA);优化浓度与耦合条件,确保多重检测无交叉干扰;用甲基化/非甲基化对照验证特异性(甲基化对照检测率≥58%,非甲基化对照无信号);通过梯度稀释实验评估灵敏度(检测限)与重复性(标准差)。
结果解读:所有assay均符合要求:甲基化对照检测率58%~98%,非甲基化对照无信号(特异性良好);重复性实验标准差≤0.05%(稳定);灵敏度达1~4拷贝/标志物(经亚硫酸氢盐转化后,最小输入0.03 ng DNA,约9~10个拷贝)。白蛋白内参有效校正了样本间的cfDNA输入差异。
产品关联:文献未提及具体实验产品,领域常规使用Bio-Rad QX200数字PCR仪,引物/探针由IDT等公司合成。
3.3 回顾性临床队列的诊断性能评估
实验目的:在肾移植患者中,评估10重dPCR panel对排斥反应的诊断价值,比较其与现有指标(eGFR、DSA)的性能差异。
方法细节:收集170例肾移植受者的血浆样本(活检前采集,EDTA/PAXgene管),按Banff 2022标准分为“排斥组”(44例,含抗体/ T细胞介导排斥)与“无排斥组”(126例)。通过单变量/多变量分析筛选与排斥相关的指标,构建预测模型,用ROC曲线评估AUC(曲线下面积)。
结果解读:单变量分析显示,eGFR、DSA及5种甲基化标志物(CTDP1、LOC124903692等)与排斥显著相关(p<0.05);多变量分析中,eGFR(p=0.0013)、DSA(p=0.0075)、LOC124903692(p=0.00106)是独立预测因子。ROC曲线显示,结合eGFR+DSA+甲基化标志物的模型AUC=0.884,显著高于eGFR+DSA模型(0.776,p=0.0005)或单独甲基化模型(0.745);预测“任何Banff病变”时,甲基化模型AUC=0.754,显著优于DSA(0.596,p=0.004)。
产品关联:文献未提及具体实验产品,领域常规使用Qiagen试剂盒提取cfDNA,Zymo试剂盒进行亚硫酸氢盐转化。
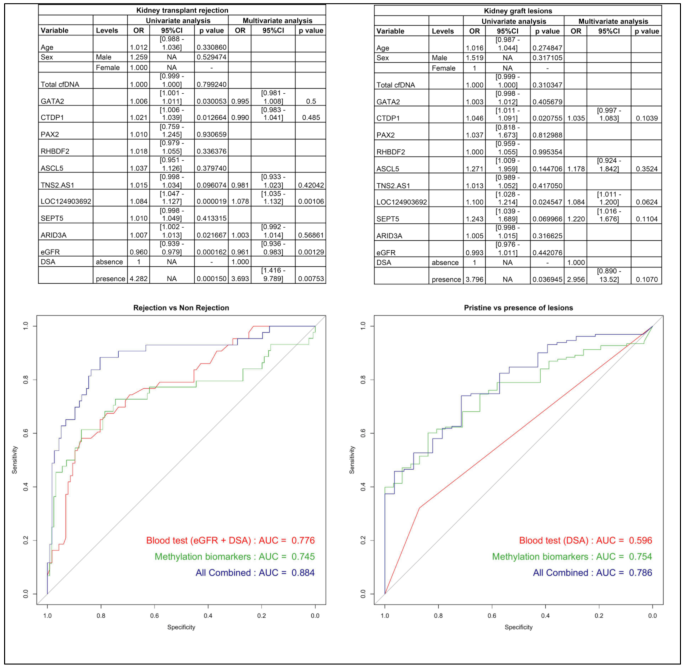
(图2展示了多变量分析结果与ROC曲线,验证了结合模型的优势)
4. Biomarker研究及发现成果解析
Biomarker定位:本研究的Biomarker为9种肾组织细胞特异性甲基化DNA标志物+1种白蛋白内参。筛选逻辑为“in silico数据库筛选→肾组织细胞验证→临床队列验证”:先通过GEO数据筛选肾高甲基化、血细胞低甲基化的标志物,再通过肾上皮/内皮细胞验证细胞特异性,最后在临床队列中验证与排斥的关联。
研究过程详述:Biomarker来源为肾移植受者的血浆cfDNA(170例);验证方法为甲基化特异性dPCR(仅检测甲基化的DNA片段);特异性方面,标志物在血细胞中甲基化率极低(避免干扰),在肾组织中显著高于肝、肺;敏感性方面,可检测1~4拷贝的甲基化DNA(满足临床样本低浓度需求)。
核心成果提炼:①功能关联:LOC124903692(肾内皮标志物)是排斥的独立预测因子(p=0.00106);结合模型对排斥的AUC达0.884,显著高于现有标准;②创新性:首次将cfDNA甲基化技术应用于肾移植领域,开发出针对肾细胞类型的10重dPCR assay;③统计学结果:结合模型AUC(0.884)显著高于eGFR+DSA(0.776,p=0.0005),预测任何Banff病变的AUC(0.754)显著优于DSA(0.596,p=0.004)。
综上,本研究开发的甲基化cfDNA标志物 panel,为肾移植排斥的无创诊断提供了新工具,有望减少活检的使用,提高随访的安全性与效率。
