1. 领域背景与文献引入
文献英文标题:The use of haplotype-specific transcripts improves sample annotation consistency;发表期刊:Biomark Res;影响因子:未公开;研究领域:生物标志物与微阵列数据质量控制(样本注释一致性的转录组标记物研究)
临床分子研究与生物标志物开发依赖高质量数据,而样本注释的准确性是数据质量的核心支撑——错误的样本注释(如供体ID混淆、性别标注错误)会导致生物标志物发现偏差、患者分层错误,甚至影响临床决策的安全性。领域共识:临床数据集的样本注释错误率可达18%,现有解决方案如REDKX性别标记物(基于异染色体基因如RPS4Y1、XIST的性别依赖性表达)可有效检测性别注释错误,但无法区分同性别供体的样本归属,仍存在关键局限性。此外,现有策略多在数据分析后期通过统计方法(如数据扰动、标签错误识别算法)处理注释错误,缺乏早期、直接的转录组水平检测工具。为填补这一空白,本研究聚焦单倍型特异性转录本的双模态表达特征,旨在开发可早期检测供体ID注释错误的转录组标记物,补充REDKX的不足,从性别和供体ID两个维度提高微阵列数据的样本注释一致性。
2. 文献综述解析
作者在综述部分围绕“样本注释准确性”的核心逻辑展开评述:首先强调样本注释错误对临床研究的危害性(如错误的生物标志物结论、患者护理风险),接着回顾现有解决方案的进展与局限——REDKX标记物解决了性别注释问题,但无法区分同性别供体;然后引入双模态表达模式的概念:部分基因(如人类白细胞抗原HLA基因)因等位基因选择性表达,在不同供体中呈现“开/关”式双模态信号分布,具有供体“分子指纹”的特征;最后说明Hartigans’ dip test的原理:通过计算经验p值识别偏离单模态分布的探针集,为筛选双模态转录本提供方法学基础。
现有研究的关键结论包括:REDKX是有效的性别注释工具,但无法覆盖同性别供体的注释错误;HLA基因的等位基因选择性表达使其具备供体特异性;双模态表达的探针集可通过dip test从微阵列数据中识别。现有研究的局限性则在于:缺乏针对供体ID注释错误的早期转录组标记物;单性别标记物无法解决同性别供体的识别问题。本研究的创新点在于:通过无监督分析从公共数据集中识别HLA-DQA1和HLA-DRB4的探针集作为供体ID标记物,结合REDKX标记物提高样本注释一致性,并在多个公共数据集上验证了性能,填补了早期检测供体ID注释错误的工具空白。
3. 研究思路总结与详细解析
本研究的整体框架为:“筛选双模态探针集→验证等位基因选择性→多数据集性能评估→结合REDKX优化注释”,核心目标是开发基于单倍型特异性转录本的标记物,解决微阵列数据中供体ID注释错误的早期检测问题;核心科学问题是“如何利用双模态表达的探针集区分不同供体的样本”。
3.1 训练数据集的探针集筛选
实验目的是从公共微阵列数据集筛选具有双模态表达特征的探针集,作为供体ID标记物的候选。方法细节:使用GSE7753数据集(47个Affymetrix HG-U133_Plus_2微阵列样本,来自系统性幼年特发性关节炎研究),首先通过MAS5算法归一化数据,再过滤强度(要求90th百分位>log₂(6)),最终得到21044个探针集;对每个探针集进行Hartigans’ dip test,模拟1×10⁶次计算经验p值(筛选p<0.001的探针集),同时要求次要等位基因频率<50%(确保标记物的供体区分能力)。结果解读:筛选出多个双模态探针集,其中HLA-DQA1(203290_at、213831_at)和HLA-DRB4(209728_at)的探针集表现突出——图1(Quantile-quantile plot)显示,模拟的单模态数据沿Identity线分布,而候选探针集的dip test结果明显偏离,验证了其双模态特征。产品关联:文献未提及具体实验产品,领域常规使用Affymetrix HG-U133_Plus_2微阵列、R软件(dip test包)进行数据分析。
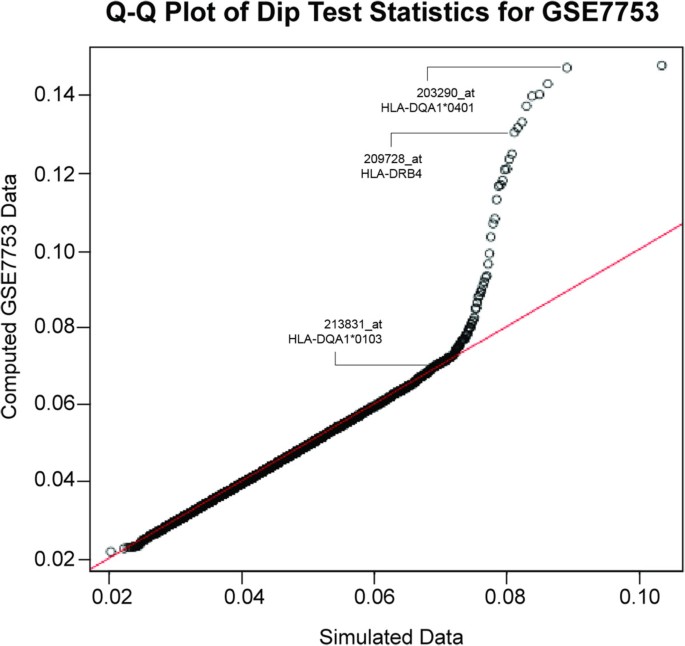
3.2 标记物的验证与性能评估
实验目的是验证候选标记物在多组织、多供体数据集上的供体ID注释检测性能,并结合REDKX标记物优化注释一致性。方法细节:选择5个公共数据集(共188个样本,53个供体,涵盖全血、外周血单个核细胞PBMC、肺活检等组织),计算每个样本的HLA-score(基于3个探针集的强度阈值:超过阈值记1,否则记0);同时使用REDKX标记物评估性别注释,结合两者结果判断供体ID注释的一致性。结果解读:6个样本被标记为注释可疑(约3%),其中5个样本的错误无法通过公共数据解决(需溯源分析);例如供体35的4个样本中,时间点1的HLA-score与其他样本差异显著,且REDKX显示该样本为女性(而其他样本为男性),提示性别和供体ID均错误;时间点4的样本虽REDKX显示为男性,但HLA-score与其他男性样本差异显著(两个探针集的强度差异达10-46倍)。图2显示候选探针集在GSE7753数据集中的双模态强度分布及经验阈值(log₂(7)),进一步验证了标记物的双模态特征。产品关联:文献未提及具体实验产品,领域常规使用GEO数据库的公共数据集、R软件进行数据分析。
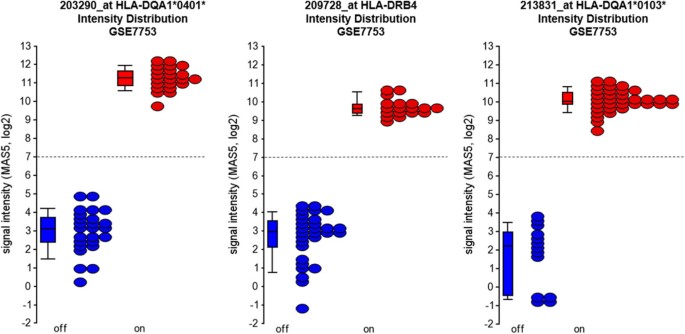
4. Biomarker 研究及发现成果解析
Biomarker 定位与筛选逻辑
本研究的Biomarker为HLA-DQA1和HLA-DRB4的转录组探针集,具体包括:203290_at(对应HLA-DQA10401等位基因)、213831_at(对应HLA-DQA10103等位基因)、209728_at(对应HLA-DRB4)。筛选与验证逻辑为“三步法”:(1)通过Hartigans’ dip test从GSE7753数据集筛选双模态探针集(p<0.001);(2)通过序列比对验证等位基因选择性(表2显示203290_at与HLA-DQA10401序列一致,与其他等位基因存在SNP差异;213831_at与HLA-DQA10103一致);(3)在5个公共数据集(188个样本)中验证其供体ID注释的检测性能,并结合REDKX标记物评估一致性。
研究过程与核心数据
Biomarker的来源为公共微阵列数据集的转录组数据(GSE7753及其他5个数据集的样本)。验证方法包括:(1)双模态验证:dip test显示候选探针集的经验p<0.001,偏离单模态分布;(2)等位基因验证:序列比对显示探针集与特定HLA等位基因完全匹配,与其他等位基因存在SNP差异;(3)性能验证:计算HLA-score并结合REDKX,在188个样本中检测到6个注释可疑样本(约3%)。特异性与敏感性数据:供体35的时间点1样本,HLA-score和REDKX均显示性别错误(应为男性但标记为女性),特异性较高;供体45的时间点1样本,209728_at的强度超过阈值(187 vs 阈值128),导致HLA-score与其他样本差异,敏感性良好。
成果与创新价值
核心成果:该Biomarker作为供体ID注释的早期质量控制工具,结合REDKX标记物可从“性别+供体ID”两个维度提高样本注释一致性——HLA-score通过等位基因选择性表达区分不同供体,补充了REDKX无法区分同性别供体的不足。创新性体现在:(1)方法创新:首次通过无监督分析(dip test)从微阵列数据中识别HLA基因的双模态探针集,作为供体ID的分子标记;(2)性能创新:标记物具有组织独立性(覆盖全血、PBMC、肺活检等),适用范围广;(3)应用创新:将HLA基因的“分子指纹”特征转化为样本注释的质量控制工具,填补了早期检测供体ID错误的空白。
统计学结果:188个样本中6个可疑(约3%),5个样本的错误无法通过公共数据解决(需溯源分析);供体35的时间点1样本,REDKX显示性别错误(n=4,信号差异显著),HLA-score也与其他样本差异显著,验证了标记物的有效性。
综上,本研究开发的HLA-DQA1/HLA-DRB4探针集,为微阵列数据的样本注释提供了“性别+供体ID”的双重质量控制工具,对提高生物标志物研究的可靠性具有重要意义。
