1. 领域背景与文献引入
文献英文标题:Diagnosing and monitoring pancreatic cancer through cell-free DNA methylation: progress and prospects;发表期刊:Biomarker Research;影响因子:未公开;研究领域:胰腺癌液体活检与表观遗传生物标志物。
胰腺癌是发达国家死亡率第三的恶性肿瘤,5年生存率仅9%(引用1、2)。欧洲每年约9.5万人死于该病(引用3),发病率呈上升趋势,患者多为70-80岁(引用4)。其核心挑战在于早期诊断困难:约80%患者确诊时已处于局部晚期或转移性阶段,仅15%-20%可行手术切除,术后复发率高(中位生存期15-20个月);转移性患者中位生存期仅4.6个月(引用7、8)。胰腺导管腺癌(PDAC)占比80%以上,KRAS突变率超90%(引用9、10),但基因检测难以用于早期诊断。
现有临床常用生物标志物CA19-9局限性明显:敏感性79%、特异性82%(引用17),且在慢性胰腺炎等良性疾病中也会升高,无法满足早期诊断需求。液体活检作为非侵入性检测手段,通过分析循环肿瘤细胞(CTCs)、游离DNA(cfDNA)等成分,能实时反映肿瘤动态变化,成为研究热点。其中,cfDNA甲基化因具有肿瘤特异性(甲基化是癌症的表观遗传标志,涉及肿瘤抑制基因沉默和癌基因激活)、可重复采样、能监测肿瘤进化等优势,成为胰腺癌生物标志物研究的重点方向(引用21、25)。
2. 文献综述解析
核心评述逻辑
作者围绕“cfDNA在胰腺癌的应用价值→DNA甲基化在胰腺癌的生物学意义→cfDNA甲基化在胰腺癌诊断/预后/监测的研究进展→现存挑战与未来方向”的脉络,系统综合近20年相关研究,重点阐述cfDNA甲基化作为胰腺癌生物标志物的潜力与局限。
现有研究总结
早期研究(2007年)采用甲基化特异性PCR(MSP)检测血浆中ppENK和p16基因甲基化,PDAC患者中阳性率仅24.6%-29.3%,敏感性极低且缺乏健康对照(引用48)。2009年Melnikov等用MethDet56技术(分析56个基因),发现5个低甲基化(hypomethylated)基因(CCND2、PLAU、SOCS1、THBS、VHL),能区分PDAC患者与健康人(敏感性76%、特异性59%)(引用68)。2013年Yi等通过转录组芯片筛选出BNC1和ADAMTS1,在123例PDAC组织中甲基化率分别为91%和67%,血清中验证显示BNC1敏感性79%、特异性89%,ADAMTS1敏感性48%、特异性92%,组合后早期PDAC敏感性达90%(引用74)。
近年大样本研究中,Majumder等(2021年)用13基因panel(GRIN2D、CD1D等)结合CA19-9,在170例PDAC、170例健康人中验证,能检测79%(I期)、82%(II期)、94%(III期)、99%(IV期)的患者,敏感性82%、特异性94%(引用87)。Garcia-Ortiz等(2023年)发现NPTX2甲基化能预测转移性PDAC患者预后,且其变化早于CT影像学提示的疾病进展(引用90)。
现有研究的共性局限:①样本量小(多为数十例);②缺乏慢性胰腺炎等良性疾病对照(如Shinjo等未纳入良性群体);③技术方法未标准化(MSP、qMSP、MOB等结果可比性差);④纵向监测研究少(仅少数关注治疗过程中的甲基化变化)。
文献创新价值
作者首次系统整合了cfDNA甲基化在胰腺癌诊断、预后、监测全流程的研究,强调“多基因panel+现有标志物(如CA19-9)”的组合策略能显著提高准确性;同时指出纵向监测(如NPTX2甲基化)在治疗反应评估中的独特优势,为临床应用提供了新方向。
3. 研究思路总结与详细解析
整体框架
本文为系统性综述,研究目标是总结cfDNA甲基化在胰腺癌诊断与监测中的研究进展;核心科学问题是“cfDNA甲基化能否作为胰腺癌的有效生物标志物”;技术路线为“文献检索→数据提取→质量评估→综合分析→结论总结”。
分步骤解析
3.1 文献检索与筛选
实验目的:获取所有关于cfDNA甲基化与胰腺癌的相关研究。
方法细节:2022年10月在PubMed数据库检索,使用MeSH术语(如“Pancreatic cancer”“cfDNA methylation”)及自由文本(如“liquid biopsy”“epigenetics”);排除仅摘要可用或非英文的文献,补充纳入参考文献中的相关研究(引用原文方法部分)。
结果解读:共纳入近50篇研究,涵盖cfDNA甲基化的检测方法、基因panel、临床应用等方面,为后续分析提供了全面数据基础。
3.2 cfDNA在胰腺癌的应用分析
实验目的:阐述cfDNA作为胰腺癌生物标志物的基础与局限。
方法细节:综合现有研究,分析cfDNA的来源(正常细胞更新、凋亡/坏死细胞、肿瘤细胞)、特点(片段大小40-200bp,肿瘤患者cfDNA片段更小)、检测方法(PCR-based技术如BEAMing、数字PCR等,需超灵敏方法因循环肿瘤DNA(ctDNA)占比低)(引用22-31)。
结果解读:cfDNA能检测KRAS、TP53等突变,转移性PDAC患者中突变检出率超80%,但早期患者仅48%(引用32);KRAS突变与不良预后相关,但部分研究未验证其监测价值(引用35-41)。cfDNA的核心局限是浓度低(血浆中约10-15ng/mL)、ctDNA占比小(早期癌症中占比<1%),需超灵敏技术才能检测(引用26、27)。
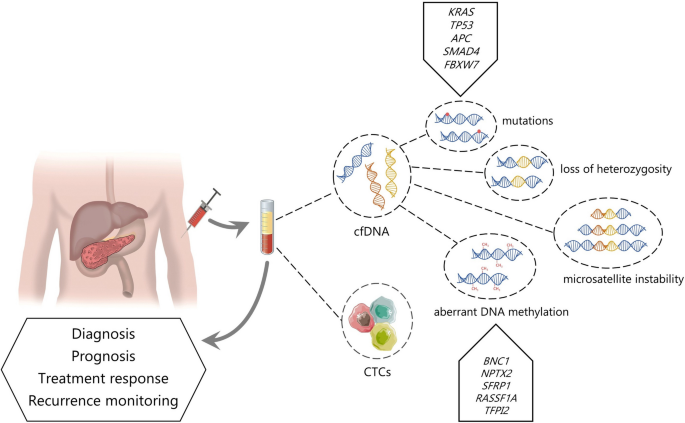
(图1:cfDNA在胰腺癌诊断与管理中的应用示意图,展示了cfDNA甲基化能反映肿瘤基因突变、甲基化模式等信息)
3.3 DNA甲基化在胰腺癌的生物学意义分析
实验目的:说明DNA甲基化与胰腺癌发生发展的关系。
方法细节:综述DNA甲基化的机制(DNMT1维持甲基化,DNMT3A/B从头甲基化)、胰腺癌中的甲基化异常(肿瘤抑制基因高甲基化(hypermethylation)如p16、PCDH10,癌基因低甲基化(hypomethylation)如SERPINB5)(引用42-59)。
结果解读:胰腺癌中约80%患者DNMT1表达上调,导致p16(细胞周期抑制基因)、PCDH10(肿瘤抑制基因)等高甲基化,进而沉默其表达(引用47、53);SERPINB5等基因低甲基化导致过表达,促进肿瘤进展(引用57、58)。此外,5-羟甲基胞嘧啶(5hmC)水平在胰腺癌中显著降低,可能作为诊断标志物(引用64)。
3.4 cfDNA甲基化在胰腺癌的临床研究分析
实验目的:评估cfDNA甲基化作为胰腺癌生物标志物的临床价值。
方法细节:按“单个基因→小基因panel→大基因panel”的顺序,分析不同研究的检测方法(MSP、qMSP、MOB、ddPCR等)、样本量、敏感性/特异性(引用48、68、74、78、87等)。
结果解读:①单个基因:NPTX2甲基化在PDAC患者中占84%,慢性胰腺炎中33%,良性胆石症中0%(n=104+60+5,P=0.016),敏感性80%、特异性76%(引用72);②小基因panel:BNC1+ADAMTS1组合在PDAC中占97.4%,但在慢性胰腺炎中占87.5%,无法区分良恶性(引用78);4基因panel(BNC1+ADAMTS1+LRFN5+PXDN)在血浆中敏感性100%、特异性90%(引用85);③大基因panel:13基因panel结合CA19-9,敏感性82%、特异性94%,能检测各期PDAC(引用87)。
3.5 挑战与结论分析
实验目的:总结现有研究的局限与未来方向。
方法细节:综合分析研究中的共性问题(技术标准化不足、样本量小、缺乏慢性胰腺炎对照、纵向研究少)(引用作者结论部分)。
结果解读:现有研究存在技术方法差异(如MSP与qMSP的结果可比性差)、样本异质性(cfDNA浓度低导致检测难度大)、缺乏多中心大样本验证等问题。未来需标准化检测流程(如样本处理、甲基化分析方法)、开展大样本临床研究(纳入健康人、慢性胰腺炎患者)、探索多标志物组合(如cfDNA甲基化+CA19-9+CTCs)、加强纵向监测研究(如治疗过程中的甲基化变化)。
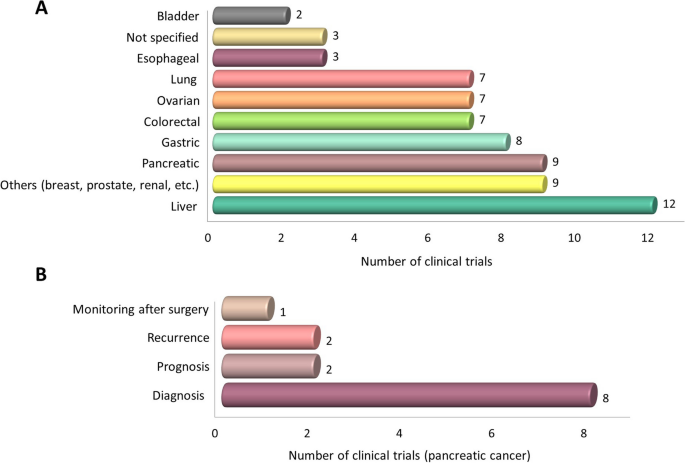
(图2:cfDNA甲基化在癌症临床trial的分布示意图,其中胰腺癌trial主要关注早期诊断)
4. Biomarker研究及发现成果解析
Biomarker定位
本文涉及的Biomarker为cfDNA中的甲基化基因/基因panel,类型包括:①单个基因(ppENK、p16、BNC1、ADAMTS1、NPTX2、ZNF154);②小基因panel(BNC1+ADAMTS1、4基因panel);③大基因panel(13基因panel)。筛选逻辑为“组织研究(发现差异甲基化基因)→血清/血浆验证(评估敏感性/特异性)→临床队列验证(确定诊断/预后价值)”;验证逻辑为“病例对照研究(PDAC vs 健康人/良性疾病)→纵向研究(监测治疗反应)”。
研究过程详述
- 单个基因:NPTX2甲基化通过MSP在104例PDAC、60例慢性胰腺炎、5例良性胆石症中验证,PDAC中占84%,慢性胰腺炎33%,良性0%(n=104+60+5,P=0.016),敏感性80%、特异性76%(引用72);通过ddPCR在44例转移性PDAC患者中验证,其甲基化水平与疾病进展相关,且变化早于CT影像学(引用90)。
- 小基因panel:BNC1+ADAMTS1通过MOB技术在42例PDAC、26例健康人中验证,BNC1敏感性79%、特异性89%,ADAMTS1敏感性48%、特异性92%,组合后敏感性90%(引用74);9年后在39例PDAC、95例健康人、8例慢性胰腺炎中验证,组合后PDAC中占97.4%,但慢性胰腺炎中占87.5%(引用78)。
- 大基因panel:13基因panel(GRIN2D、CD1D等)通过TELQAS技术在170例PDAC、170例健康人中验证,结合CA19-9后,敏感性82%、特异性94%,I期PDAC检出率79%(引用87)。
核心成果提炼
- 诊断价值:大基因panel(13基因+CA19-9)能有效区分PDAC与健康人/良性疾病,敏感性82%、特异性94%,I期检出率79%(引用87);4基因panel(BNC1+ADAMTS1+LRFN5+PXDN)在血浆中敏感性100%、特异性90%(引用85)。
- 预后价值:NPTX2甲基化水平越高,转移性PDAC患者预后越差(引用90);SST基因高甲基化与PDAC患者生存期缩短相关(引用83)。
- 监测价值:NPTX2甲基化变化能早于CT提示疾病进展,优于CA19-9(引用90);Vrba等用10基因甲基化签名监测9例转移性PDAC患者,治疗后甲基化水平显著降低(引用89)。
创新性:首次系统总结了cfDNA甲基化在胰腺癌监测中的研究(如NPTX2甲基化的纵向研究),强调“甲基化panel+现有标志物”的组合策略能提高准确性,为临床应用提供了依据。
注:文献未提及具体实验产品,领域常规使用甲基化特异性PCR试剂盒、数字PCR仪、MOB试剂盒等试剂/仪器。
