1. 领域背景与文献引入
文献英文标题:Targeted proteomics-derived biomarker profile develops a multi-protein classifier in liquid biopsies for early detection of esophageal squamous cell carcinoma from a population-based case-control study;发表期刊:Biomarker Research;影响因子:5.1;研究领域:食管鳞状细胞癌早期诊断生物标志物研究
食管鳞状细胞癌(ESCC)是全球高发恶性肿瘤,2018年全球新发病例57.2万、死亡50.9万,死亡率与发病率比值超0.8,5年生存率仅15%-25%——核心原因是多数患者确诊时已达晚期。现有内镜筛查虽能识别早期病变,但侵入性强、成本高,在高发区(如中国泰兴)导致人群参与率低,造成沉重公共卫生负担。因此,开发非侵入性、高性价比的血液生物标志物(液体活检),用于ESCC风险分层以筛选高风险人群接受内镜检查,成为领域关键需求。
当前研究热点集中于血液生物标志物(如cfDNA突变、非编码RNA、蛋白质),但多数蛋白标志物的曲线下面积(AUC)低于0.8,缺乏高敏感性与特异性;靶向蛋白质组学技术(如邻近延伸分析,PEA)可同时定量检测多蛋白,为高效筛选标志物提供工具,但尚未应用于ESCC早期诊断的大样本人群研究。
本研究针对“ESCC早期诊断缺乏有效血液标志物”的核心问题,利用靶向蛋白质组学技术在大样本人群中筛选差异蛋白,构建多蛋白分类器并验证其价值,旨在为ESCC三级筛查策略(环境暴露+血液活检+内镜)提供关键工具。
2. 文献综述解析
文献综述以“生物标志物类型+检测技术”为分类维度,系统评述现有研究的结论、优势与局限性:
现有研究总结
- 非靶向蛋白质组学:通过质谱技术发现ApoA-I、HSP70等ESCC相关蛋白,但样本量小(多为几十例),缺乏人群验证;
- 血液-based单一标志物:蛋白芯片、ELISA等技术发现FAPα、IGFBP7、自身抗体panel(p53、MMP-7等),但AUC多低于0.8(如FAPα联合传统标志物AUC=0.745,IGFBP7 AUC=0.725),特异性不足;
- 靶向蛋白质组学:PEA等技术可同时检测多蛋白,敏感性与特异性高,但尚未应用于ESCC早期诊断的大样本人群研究。
现有研究局限性
样本量小、标志物特异性不足、缺乏大样本人群验证、未结合剂量-反应关系分析(即标志物水平与疾病进展的相关性)。
本研究创新价值
首次在ESCC高发区的大样本人群(30例癌前病变、60例I期、70例II期、70例III/IV期ESCC患者及70例健康对照)中,使用Olink Oncology II靶向蛋白质组学panel(92种癌相关蛋白)筛选早期诊断标志物;通过剂量-反应关系验证确保标志物与疾病进展的相关性,最终构建高准确性多蛋白分类器(AUC=0.936),弥补了现有研究的不足。
3. 研究思路总结与详细解析
整体框架概括
研究目标:开发ESCC早期检测的血清多蛋白分类器;
核心科学问题:筛选能区分早期ESCC与健康对照的差异蛋白,构建高准确性分类模型;
技术路线:人群样本选择→靶向蛋白检测(Olink PEA)→生物信息学分析(差异筛选+剂量-反应验证)→多蛋白模型构建与验证。
3.1 研究人群选择与样本处理
实验目的:选择符合条件的研究对象,确保组间性别、年龄分布均匀,排除干扰因素(如溶血、治疗后样本)。
方法细节:从中国ESCC高发区(泰兴市)的人群病例对照研究中筛选样本,纳入标准:健康对照(无食管疾病)、癌前病变(高级别上皮内瘤变)、I期(早期)、II期、III/IV期(晚期)ESCC患者;匹配性别和5年年龄组;排除溶血样本和治疗后采集的样本。最终纳入70例健康对照、30例癌前病变、60例I期、70例II期、70例III/IV期ESCC患者。
结果解读:各组性别、年龄分布均匀(Table S2),确保组间可比性;排除干扰样本后,样本质量符合检测要求。
产品关联:文献未提及具体样本处理产品,领域常规使用血清分离管(如BD Vacutainer)、-80℃超低温冰箱(如Thermo Fisher)等。

3.2 靶向蛋白质组学检测
实验目的:定量检测血清中92种癌相关蛋白的表达水平,获取差异蛋白谱。
方法细节:使用Olink Multiplex Oncology II panel,基于邻近延伸分析(PEA)技术(结合抗体特异性与PCR敏感性),将血清样本随机分配到4个96孔板,每板包含3个板间对照(用于归一化)和3个阴性对照(建立背景);数据归一化为Normalized Protein eXpression(NPX)值(log scale相对定量)。
结果解读: intra-assay变异系数(CV)为5%,inter-assay CV为23%,表明检测重复性良好;92种蛋白的表达水平在各组间存在显著差异。
产品关联:实验所用关键产品:Olink Multiplex Oncology II panel(未提及货号)。
3.3 生物信息学分析与标志物筛选
实验目的:筛选能区分早期ESCC与健康对照的差异蛋白,验证其与疾病进展的相关性,并分析蛋白功能。
方法细节:1)主成分分析(PCA)评估组间聚类;2) unconditional logistic回归分析蛋白NPX值与早期ESCC的关联(FDR<0.01控制假阳性);3)Spearman相关验证蛋白水平与ESCC分期的剂量-反应关系(FDR<0.01);4)STRING数据库分析蛋白互作网络,ConsensusPathDB做基因本体(GO)与通路富集分析。
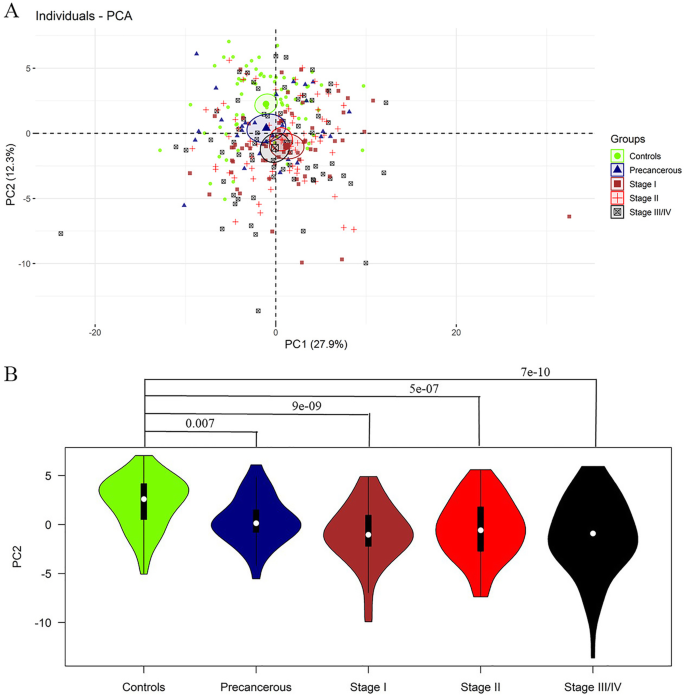
结果解读:1)PCA显示健康对照与ESCC组在主成分2(PC2)上显著分离(图2);2)筛选出23种差异蛋白(10种上调、13种下调),均与ESCC分期呈剂量-反应关系(如ANXA1的NPX值随分期升高而增加,hK8随分期升高而降低,图4);3)蛋白互作网络显示13种蛋白(ANXA1、hK8、VIM等)存在相互作用;4)GO富集显示分子功能(信号受体结合、催化活性)、细胞组分(细胞外空间)、生物过程(刺激反应负调控);通路富集显示TP53网络和Glypican 1网络(Table S3、S4)。
产品关联:文献未提及具体生物信息学工具,领域常规使用R软件(version 3.6.2)、STRING数据库、ConsensusPathDB数据库。

3.4 多蛋白分类器构建与验证
实验目的:构建简洁高效的多蛋白分类器,验证其早期诊断价值。
方法细节:1)最小绝对收缩和选择算子(LASSO)回归从23种蛋白中筛选11种最优蛋白(降维避免过拟合);2)向后消除法进一步缩小到5种核心蛋白(ANXA1、hK8、hK14、VIM、RSPO3);3)构建logistic回归模型,ROC曲线评估AUC,五折交叉验证评估稳定性。
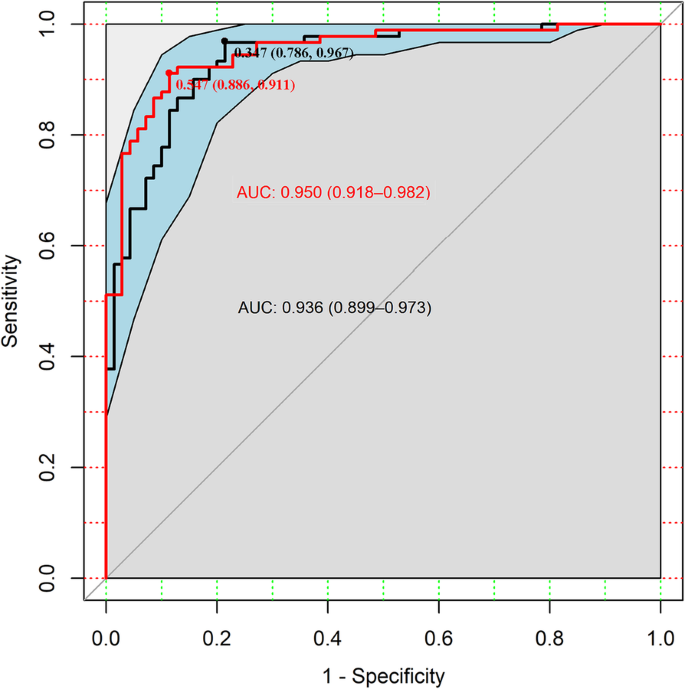
结果解读:5蛋白分类器区分早期ESCC与健康对照的AUC为0.936(95%置信区间:0.899-0.973),敏感性96.7%、特异性78.6%(图5);五折交叉验证显示训练集准确率0.861、测试集0.825,模型稳定性良好。
产品关联:文献未提及具体统计软件,领域常规使用R软件的glmnet包(LASSO回归)、pROC包(ROC分析)等。
4. Biomarker研究及发现成果解析
Biomarker定位
类型:血清蛋白生物标志物(ANXA1、hK8、hK14、VIM、RSPO3);
筛选逻辑:Olink panel检测92种蛋白→logistic回归筛选差异蛋白(FDR<0.01)→Spearman相关验证剂量-反应关系(FDR<0.01)→LASSO+向后消除筛选核心蛋白。
研究过程详述
Biomarker来源:ESCC患者与健康对照的治疗前血清样本(排除溶血干扰);
验证方法:1)Olink PEA检测(定量蛋白水平);2)logistic回归(关联分析);3)Spearman相关(剂量-反应关系);4)ROC曲线(诊断价值);
特异性与敏感性:5蛋白分类器的特异性78.6%、敏感性96.7%,AUC=0.936(95%CI 0.899-0.973)。
核心成果提炼
- 功能关联:ANXA1、VIM在ESCC患者血清中上调(与肿瘤增殖、转移相关),hK8、hK14、RSPO3下调(与肿瘤抑制、信号通路调控相关);
- 诊断价值:5蛋白分类器的AUC(0.936)显著高于现有单一标志物(如IGFBP7的AUC=0.725),且与ESCC分期呈剂量-反应关系(确保标志物与疾病进展的相关性);
- 创新性:首次在大样本人群中验证这些蛋白作为ESCC早期诊断标志物的价值,为ESCC三级筛查策略提供了高敏感性的血液检测工具。
本研究通过靶向蛋白质组学与大样本人群验证,解决了ESCC早期诊断缺乏有效血液标志物的问题,为高发区ESCC防控提供了关键技术支撑。未来需在多中心队列中进一步验证分类器的通用性,同时探索标志物与其他癌种的特异性。
