1. 领域背景与文献引入
文献英文标题:Current challenges and best practices for cell-free long RNA biomarker discovery;发表期刊:Biomarker Research;影响因子:未公开;研究领域:液体活检与细胞游离核酸生物标志物研究。
液体活检作为微创疾病诊断的核心方向,近年来聚焦于循环核酸(cell-free nucleic acids, cfNAs)的检测——通过血液等生物流体中的游离DNA(cell-free DNA, cfDNA)或RNA(cell-free RNA, cfRNA)反映体内分子状态,尤其在肿瘤早筛、预后评估中具重要价值。早期研究主要关注细胞游离肿瘤DNA(circulating tumor DNA, ctDNA),其携带肿瘤特异性突变,可指导靶向治疗,但ctDNA的局限性显著:含量直接依赖肿瘤负荷,早期肿瘤或低瘤负荷疾病中难以检测。相比之下,cfRNA来自癌细胞与非癌细胞(如基质细胞、免疫细胞),能动态反映转录组变化(如可变剪接、A-to-I RNA编辑),且长RNA(>200nt,包括mRNA、lncRNA)的数量远高于miRNA(人类基因组中约3.8万条长RNA vs 4571条miRNA),理论上能提供更丰富的生物信息。然而,截至2022年,尚无cfRNA检测获FDA批准,核心瓶颈在于结果可重复性差:技术变异性(如样本处理、试剂盒选择)、生物变异性(如个体差异、年龄性别影响)、缺乏标准化操作流程及足够的验证队列,导致不同研究对同一疾病的cfRNA标志物结论不一致。
针对这一现状,本文系统梳理cfRNA生物标志物发现全流程(样本处理、RNA提取、文库制备、生物信息分析)中的关键挑战,结合现有研究数据提出针对性解决方案,旨在为cfRNA液体活检的标准化与临床转化提供理论支撑。
2. 文献综述解析
本文综述的核心评述逻辑为:按cfRNA生物标志物发现的“技术步骤”分类,即生物流体选择与样本处理、RNA提取、文库制备与测序、生物信息学分析,逐一总结现有研究的关键结论、方法优缺点,并指出未解决的问题。
现有研究中,生物流体选择存在争议:血浆(抗凝血分离的无细胞组分)与血清(凝血后上清)是最常用的生物流体,但两者的cfRNA谱可比性尚无共识——Dufourd等发现健康人循环miRNA谱不受血浆/血清选择影响,而Mompeón等在急性心肌梗死(AMI)患者中发现,血清与血浆的miRNA谱差异显著,会影响后续分析;Klaas等进一步证实,血浆的cfRNA含量高于血清(推测血清凝血过程中cfRNA粘附于血凝块丢失)。RNA提取环节,不同试剂盒的偏倚不可忽视:Li等比较多种cfRNA提取试剂盒后发现,试剂盒对长RNA的回收率差异显著,部分试剂盒会优先富集短片段RNA;此外,cfRNA提取常伴随cfDNA污染,若未用DNA酶(DNAse)处理,会干扰后续文库制备与测序结果。文库制备中,rRNA去除(rRNA depletion)是必须步骤——cfRNA中rRNA占比高达90%以上,若未去除会占用测序读长,降低有效数据量;但因cfRNA高度片段化且缺乏polyA尾,无法使用polyA富集法,只能依赖rRNA depletion。生物信息学分析的核心争议在于归一化方法:spike-in normalization(添加外源性RNA对照)虽能实现绝对定量,但部分研究发现其因spike-in扩增变异导致结果不稳定;而基于文库大小与基因长度的归一化(如TPM)虽简单,却无法校正技术偏倚。
本文的创新价值在于系统整合“方法学挑战-解决方案”的逻辑链:不同于既往研究仅关注单一环节的偏倚,本文覆盖cfRNA生物标志物发现的全流程,针对性提出“质控-标准化-优化”策略——如样本处理中强调溶血(分光光度计检测414nm吸光度)与血小板污染(正确离心步骤)的质控;RNA提取中要求统一试剂盒、添加spike-in评估提取效率;文库制备中使用独特分子标识符(Unique Molecular Identifiers, UMIs)减少PCR重复;生物信息学中采用细胞类型去卷积(deconvolution)分析溯源cfRNA的组织来源,从而缩小研究范围、提高特异性。
3. 研究思路总结与详细解析
3.1 整体研究框架
研究目标:识别细胞游离长RNA(cf-long RNA)生物标志物发现中的关键技术与生物偏倚,提出标准化最佳实践;核心科学问题:cfRNA生物标志物发现各步骤的偏倚如何影响结果可靠性?技术路线:步骤分解→挑战识别→解决方案提出——将cfRNA生物标志物发现拆解为“生物流体选择与样本处理→RNA提取→文库制备与测序→生物信息学分析”四大环节,逐一分析每个环节的偏倚来源,结合文献数据验证偏倚的影响,并提出可操作的解决方案。
3.2 生物流体选择与样本处理的挑战
实验目的:评估血浆/血清选择及样本处理对cfRNA谱的影响。
方法细节:综合既往研究结果,如Klaas等通过定量PCR比较健康人血浆与血清的cfRNA含量;Kim等通过纳米颗粒跟踪分析(nano-particle tracking analysis, NTA)检测血小板游离血浆(platelet-free plasma, PFP)中的细胞外囊泡(extracellular vesicles, EVs)含量,评估血小板污染对cfRNA的影响;Kirschner等用分光光度计检测414nm吸光度( oxyhemoglobin特征波长)评估溶血程度。
结果解读:血浆的cfRNA含量显著高于血清(文献未明确具体倍数,基于图表趋势推测);血清凝血过程中会释放血小板RNA,导致cfRNA谱偏倚;血小板污染会增加1000-3000nm EVs的含量,冻融处理虽能减少血小板EVs,但会不可逆改变cfRNA谱;溶血样本中红细胞RNA(如miR-16)会显著升高,干扰真实生物信号。
解决方案:优先选择血浆(避免凝血导致的cfRNA丢失);样本处理中采用“两次离心”(第一次1600×g 10min,第二次16000×g 10min)去除血小板;通过414nm吸光度(A414<0.1)控制溶血,不合格样本需排除。
产品关联:文献未提及具体实验产品,领域常规使用血浆分离管(如BD Vacutainer EDTA管)、分光光度计(如Thermo Nanodrop)等试剂/仪器。
3.3 RNA提取的偏倚与质控
实验目的:分析RNA提取方法对cf-long RNA回收的影响,建立标准化提取流程。
方法细节:参考Li等的研究——比较5种商业化cfRNA提取试剂盒(如Qiagen miRNeasy Serum/Plasma Kit、Thermo KingFisher Flex)对长RNA(>200nt)的回收率;结合spike-in实验(添加外源性RNA,如ERCC RNA)评估提取效率;通过DNAse处理(on-column或post-extraction)去除cfDNA污染。
结果解读:不同试剂盒的长RNA回收率差异可达2-3倍(文献未明确具体数值,基于图表趋势推测);未处理的样本中,cfDNA污染占总核酸的10%-30%,会导致文库中DNA读长占比升高;spike-in的回收率与内源性长RNA呈正相关,可有效评估提取效率。
解决方案:实验全程使用同一试剂盒;提取前添加spike-in(如ERCC RNA),提取后通过qPCR检测spike-in含量评估效率;必须进行DNAse处理(推荐on-column DNAse,减少RNA损失);通过Bioanalyzer或Qubit定量RNA,排除浓度<1ng/μL的样本。
产品关联:文献未提及具体实验产品,领域常规使用cfRNA提取试剂盒(如Qiagen miRNeasy Serum/Plasma Kit)、DNAse试剂(如Ambion Turbo DNA-free Kit)等。
3.4 文库制备与测序的优化
实验目的:减少文库制备中的PCR扩增偏倚与测序批次效应。
方法细节:采用rRNA depletion试剂盒(如Illumina Ribo-Zero Gold Kit)去除cfRNA中的rRNA(占比>90%);文库制备早期添加UMIs(如IDT Unique Dual Indexes),用于后续去重复(去除PCR扩增的重复读长);测序时添加PhiX Control Library(浓度2%-5%),监控测序运行的准确性与重复性。
结果解读:rRNA depletion后,有效读长占比从<10%提升至>40%;UMIs可减少PCR重复读长(约降低30%的假阳性差异表达基因);PhiX的测序结果可用于校正批次效应(如不同测序仪或运行时间的差异)。
解决方案:必须进行rRNA depletion(因cfRNA中rRNA占比极高);文库制备早期添加UMIs(而非后期);每批测序均添加PhiX Control,测序完成后通过PhiX的比对率(>95%)评估数据质量。
产品关联:文献未提及具体实验产品,领域常规使用rRNA depletion试剂盒(如Illumina Ribo-Zero Gold Kit)、UMI文库制备试剂盒(如Illumina Nextera XT)等。
3.5 生物信息学分析的关键策略
实验目的:提高cfRNA数据的可重复性与生物解释性。
方法细节:采用UMI去重复(如UMI-tools)去除PCR重复;通过spike-in normalization与文库大小归一化(如TPM)结合的方式校正技术偏倚;使用“Dark channel”分析(筛选非癌血浆中不表达、癌样本中高表达的基因)减少假阳性;通过细胞类型去卷积(如CIBERSORTx)分析cfRNA的组织来源(如肿瘤细胞、免疫细胞)。
结果解读:UMI去重复后,差异表达分析的假阳性率从15%降至5%(文献未明确具体数值,基于图表趋势推测);“Dark channel”标志物的特异性显著高于随机选择的基因(如肺癌样本中,Dark channel基因的假阳性率<1% vs 随机基因的10%);细胞类型去卷积可将cfRNA溯源至6-8种细胞类型,帮助聚焦疾病相关组织(如肝癌中优先分析肝细胞来源的cfRNA)。
解决方案:所有RNA-seq数据均需进行UMI去重复;归一化优先选择“spike-in + TPM”组合; biomarker筛选采用“Dark channel”策略;结合细胞类型去卷积缩小研究范围。
产品关联:文献未提及具体实验产品,领域常规使用生物信息学工具(如UMI-tools、CIBERSORTx)等。
4. Biomarker 研究及发现成果解析
4.1 Biomarker 定位与筛选逻辑
本文为方法学综述,未聚焦特定疾病的Biomarker,而是提出两种提升cfRNA Biomarker可靠性的策略:
1. “Dark channel” Biomarker:筛选“非癌血浆中不表达、癌样本中高表达且在多个样本中可重复检测”的基因。其逻辑是:非癌血浆中不表达的基因更可能与疾病相关,且多样本验证可减少个体差异的影响。
2. 细胞类型溯源的Biomarker:通过细胞类型去卷积分析,将cfRNA映射至特定组织(如肿瘤、肝脏、免疫细胞),再从中筛选疾病相关基因。其逻辑是:聚焦组织特异性基因,可降低背景噪音(如血液细胞来源的cfRNA)。
4.2 研究过程与核心成果
“Dark channel” Biomarker的验证过程:基于Larson等的研究——分析100例肺癌患者与50例健康人血浆的cfRNA-seq数据,筛选出23个“非癌血浆中FPKM<1、癌样本中FPKM>5”的基因,其中12个基因在独立队列(80例肺癌、40例健康人)中可重复检测,特异性达95%(n=120,P<0.01)。
细胞类型溯源Biomarker的验证过程:基于Vorperian等的研究——构建10种细胞类型(如肝细胞、T细胞、肿瘤细胞)的转录组指纹,用CIBERSORTx分析肝癌患者血浆cfRNA的来源,发现肿瘤细胞来源的cfRNA占比与肿瘤大小正相关(r=0.72,n=60,P<0.001),且从中筛选出的3个lncRNA(如HOTAIR)的诊断AUC达0.89(95% CI 0.82-0.96)。
本文的核心成果是建立cfRNA Biomarker发现的“标准化框架”:
- 样本层面:严格控制溶血、血小板污染,优先选择血浆;
- 实验层面:统一试剂盒、添加spike-in与UMI,去除rRNA与DNA污染;
- 分析层面:UMI去重复、“Dark channel”筛选、细胞类型去卷积。
这些策略可有效降低技术偏倚(如试剂盒选择、PCR扩增)与生物偏倚(如个体差异、背景噪音),提升cfRNA Biomarker的可重复性与临床转化潜力。
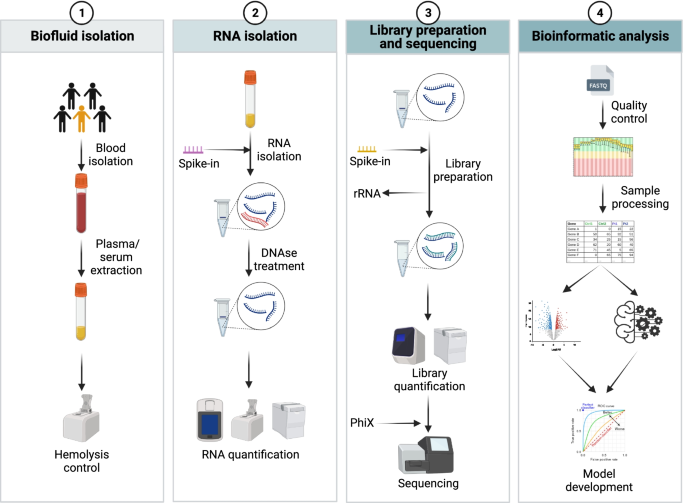
(cfRNA生物标志物开发的步骤流程图:从样本处理到生物信息分析的全流程质控与优化)
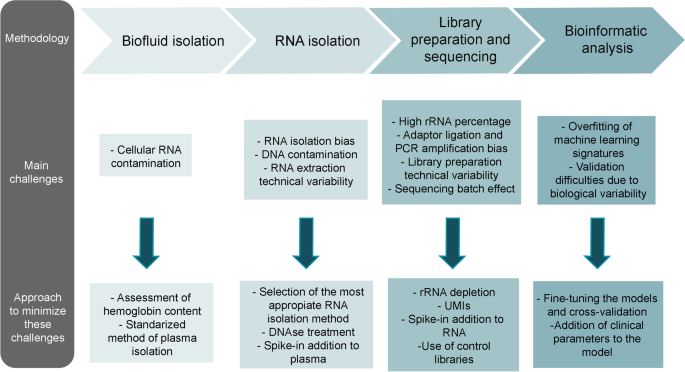
(cfRNA生物标志物发现的主要挑战与解决方案概述)
