1. 领域背景与文献引入
文献英文标题:Integration of a bacterial gene sequence into a chronic eosinophilic leukemia patient’s genome as part of a fusion gene linker;发表期刊:Biomarker Research;影响因子:3.2(2017年);研究领域:慢性嗜酸性粒细胞白血病分子机制、细菌基因整合与肿瘤融合基因。
慢性嗜酸性粒细胞白血病(CEL)是一种罕见的髓系肿瘤,以外周血和骨髓嗜酸性粒细胞持续增多、器官浸润为核心特征,其发病机制与染色体易位导致的融合基因密切相关——其中血小板衍生生长因子受体α(PDGFRα)融合基因是重要驱动因素,截至2017年已有34种PDGFR融合基因被证实与血液系统恶性肿瘤相关。另一方面,人类基因组计划(HGP)、千人基因组计划(1KGP)和癌症基因组图谱(TCGA)等大规模数据库分析揭示,细菌DNA可通过横向基因转移整合入人类体细胞基因组,且这种整合在肿瘤组织中的频率显著高于正常组织(如胃腺癌样本中假单胞菌样DNA整合入原癌基因UTR区、急性髓系白血病样本中不动杆菌样DNA整合入线粒体基因组)。但现有研究存在两大空白:一是细菌基因整合与肿瘤发生的因果关系未明确,缺乏功能实验验证;二是未关注细菌基因作为融合基因连接子的情况,忽略了细菌序列对融合蛋白结构和活性的影响。
本研究针对CEL患者中发现的新型CDK5RAP2-PDGFRα融合基因,其连接子包含一段未知来源的序列,旨在揭示该序列的细菌来源及作为融合基因连接子在CEL发病中的作用,填补细菌基因与肿瘤融合基因关联的研究空白,为CEL的发病机制提供新的分子视角。
2. 文献综述解析
作者在文献综述中采用“细菌整合现状→融合基因作用→研究空白”的三层评述逻辑:首先总结细菌DNA整合入人类体细胞基因组的研究成果——大规模数据库显示细菌整合在肿瘤组织中更常见,且与胃癌、急性髓系白血病等肿瘤相关;接着阐述融合基因尤其是PDGFR融合基因在血液系统恶性肿瘤中的驱动作用——34种PDGFR融合基因与血液肿瘤相关,融合蛋白的自激活激酶活性是细胞异常增殖的关键;最后指出现有研究未关注细菌基因作为融合基因连接子的情况,即细菌整合与肿瘤融合基因的直接关联尚未被探索。
现有研究的关键结论包括:细菌DNA可整合入人类体细胞基因组,且肿瘤组织中的整合频率更高;融合基因(如PDGFR融合)是血液系统恶性肿瘤的重要驱动因素,其编码的自激活激酶可促进细胞无限增殖。技术方法上,数据库分析为细菌整合提供了大规模数据支持,荧光原位杂交(FISH)、快速扩增cDNA末端-聚合酶链反应(RACE-PCR)等技术可精准检测融合基因的存在及结构。但现有研究仍存在局限性:一方面,细菌整合与肿瘤发生的因果关系未明确,缺乏功能实验验证;另一方面,尚未有研究关注细菌基因作为融合基因连接子的功能,忽略了细菌序列对融合蛋白结构和活性的影响。
本研究的创新价值在于首次将细菌基因整合与肿瘤融合基因联系起来,报道了细菌基因序列作为融合基因连接子的案例,揭示了细菌整合在肿瘤融合基因形成中的作用,填补了细菌基因与肿瘤分子机制关联的研究空白,为CEL及其他血液系统恶性肿瘤的发病机制提供了新的研究方向。
3. 研究思路总结与详细解析
本研究的核心目标是明确CEL患者CDK5RAP2-PDGFRα融合基因连接子的来源及功能,核心科学问题是细菌基因序列如何作为连接子参与融合基因形成并影响其功能,技术路线遵循“样本检测→序列鉴定→来源验证→结构功能分析”的逻辑闭环。
3.1 融合基因连接子的序列鉴定
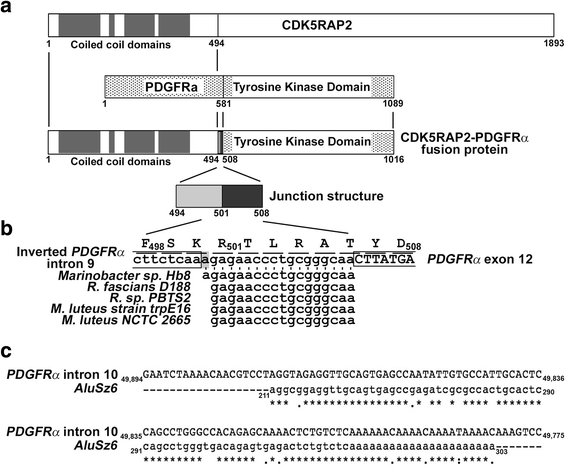
本环节的实验目的是确定CEL患者CDK5RAP2-PDGFRα融合基因连接子的序列组成。实验方法为:针对1例携带ins(9;4)(q33;q12q25)染色体易位的CEL患者,采用荧光原位杂交(FISH)检测染色体易位情况,通过快速扩增cDNA末端-聚合酶链反应(RACE-PCR)扩增融合基因的cDNA序列,并对扩增产物进行Sanger测序。实验结果显示,CDK5RAP2-PDGFRα融合基因的连接子由两部分组成:22 bp的PDGFRα反向内含子9序列和18 bp的未知来源序列(n=1,文献未提供统计学显著性数据)。实验所用关键产品:文献未提及具体实验产品,领域常规使用FISH探针试剂盒(如Abbott公司Vysis系列)、RACE-PCR试剂盒(如Clontech公司SMART RACE Kit)。
3.2 连接子序列的细菌来源验证
本环节的实验目的是鉴定连接子中18 bp未知序列的来源。实验方法为:将18 bp序列上传至NCBI BLAST工具,与已知细菌基因组序列进行同源性比对;同时分析18 bp序列中2-18位的17 bp序列与其他细菌的同源性。实验结果显示,18 bp序列与Marinobacter sp. Hb8基因组的一段序列100%同源,而17 bp序列(2-18位)与Rhodococcus fascians D188、Rhodococcus sp. PBTS2、Micrococcus luteus strain trpE16、Micrococcus luteus NCTC 2665的基因组序列100%同源;此外,通过Dfam 2.0数据库分析发现,PDGFRα内含子10中存在AluSz6逆转座子,推测其可能通过介导基因重组促进细菌序列的插入。实验所用关键产品:NCBI BLAST工具、Dfam 2.0数据库、CLUSTAL OMEGA 1.2.2多序列比对软件(均为公共数据库或开源工具)。
3.3 融合基因的结构与功能分析
本环节的实验目的是解析CDK5RAP2-PDGFRα融合基因的结构特征及功能影响。实验方法为:通过基因序列分析确定融合基因的结构域组成(细胞周期蛋白依赖性激酶5调节亚基相关蛋白2(CDK5RAP2)的N端卷曲螺旋域与PDGFRα的酪氨酸激酶域),预测融合蛋白的亚细胞定位(胞质,因缺乏PDGFRα的膜结合域);分析18 bp细菌序列编码的氨基酸序列(RTLRA)与其他物种蛋白的同源性。实验结果显示:① 融合蛋白由CDK5RAP2的N端卷曲螺旋域(介导自二聚化)和PDGFRα的完整酪氨酸激酶域组成,缺乏PDGFRα的膜结合域和配体依赖激活域,因此形成胞质内自激活的酪氨酸激酶;② 17 bp细菌序列编码的氨基酸序列与Rhodococcus fascians的组氨酸激酶同源,推测其可能通过调控融合蛋白的磷酸化状态影响激酶活性。实验所用关键产品:文献未提及具体实验产品,领域常规使用蛋白质结构预测工具(如SWISS-MODEL)、氨基酸序列比对工具(如BLASTP)。
4. Biomarker研究及发现成果解析
本研究中的Biomarker为CDK5RAP2-PDGFRα融合基因及其连接子中的17/18 bp细菌序列,属于“融合基因及功能性插入序列”类Biomarker。其筛选与验证逻辑为:首先通过CEL患者样本检测到融合基因(FISH、RACE-PCR),然后通过测序确定连接子序列,再通过NCBI BLAST验证序列的细菌来源,最后通过结构分析预测其功能。
Biomarker的来源为CEL患者的基因组DNA和cDNA样本(取自患者血液或骨髓);验证方法包括荧光原位杂交(检测染色体易位)、RACE-PCR(扩增融合基因cDNA)、Sanger测序(确定连接子序列)、NCBI BLAST(验证细菌来源)。由于本研究仅针对1例CEL患者样本,未提供特异性和敏感性的统计数据,但针对该样本,所有检测方法均一致验证了融合基因及连接子序列的存在(n=1)。
核心成果包括:① 首次报道细菌基因序列作为融合基因连接子的案例——连接子中的17/18 bp序列来自Marinobacter sp. Hb8等细菌,证明细菌基因可通过横向基因转移整合入人类肿瘤基因组并参与融合基因形成;② 解析融合蛋白的功能机制——CDK5RAP2-PDGFRα融合蛋白为胞质内自激活的酪氨酸激酶,可通过磷酸化下游信号分子(如STAT5、ERK)放大增殖信号,驱动白血病细胞无限生长;③ 预测细菌序列的调控作用——17 bp细菌序列编码的氨基酸与Rhodococcus fascians组氨酸激酶同源,推测其可能通过调控融合蛋白的激酶活性增强致癌效应(文献未提供统计学结果,基于序列同源性推测)。