1. 领域背景与文献引入
文献英文标题:Exploring non-invasive precision treatment in non-small cell lung cancer patients through deep learning radiomics across imaging features and molecular phenotypes;发表期刊:Biomarker Research;影响因子:未公开;研究领域:非小细胞肺癌(NSCLC)的深度学习放射组学与无创精准治疗。
NSCLC占肺癌病例的85%,是全球死亡率最高的恶性肿瘤之一,5年生存率不足20%。靶向治疗(如EGFR、ALK抑制剂)和免疫治疗(如PD-1/PD-L1抑制剂)显著改善了患者预后,但治疗决策依赖肿瘤分子特征检测(如基因突变、免疫表型)。传统组织活检存在有创性、样本异质性、动态变化捕捉困难等局限,无法满足临床对实时、全面分子信息的需求。
放射组学作为无创分子表型检测的关键技术,通过提取医学图像的定量特征(如形状、纹理)预测肿瘤异质性和分子特征,已在NSCLC诊断、预后评估中显示价值。但传统放射组学存在明显不足:单一肿瘤区域分析(忽略瘤周微环境)、手工特征的鲁棒性与泛化性差(易受分割方式影响)、聚焦单一分子特征(无法覆盖多维度临床需求)。近年来,深度学习与放射组学的结合(如深度网络提取自动特征)虽提升了特征表征能力,但多数研究仍局限于单一分子靶点(如仅预测EGFR突变),缺乏多区域、多特征类型的融合,难以支撑临床精准治疗决策。
针对上述空白,本文旨在开发多源融合的深度学习放射组学框架,整合肿瘤+瘤周区域的手工放射组学特征与深度网络特征,同时预测NSCLC患者的预后风险因子(淋巴血管浸润LVI、胸膜浸润PI、T分期)、基因突变(EGFR、KRAS等)、免疫表型(PD-1/PD-L1表达),为无创精准治疗提供全面影像学依据。
2. 文献综述解析
现有NSCLC放射组学研究可分为两类:
第一类是传统放射组学研究:聚焦肿瘤区域的手工特征提取(如形状、直方图、纹理),已成功预测肿瘤异质性、治疗反应及预后(如LVI、EGFR突变)。但此类研究依赖人工设计特征,存在鲁棒性差、泛化性不足的问题(如不同分割方式导致特征差异),且多关注单一分子特征,无法满足临床对多维度分子信息的需求。
第二类是深度学习与放射组学结合的研究:通过深度网络(如ResNet、DenseNet)自动提取图像特征,提升了特征的语义表征能力,在肿瘤分期、疗效预测中显示优势。但现有研究仍存在局限:① 多关注单一肿瘤区域(忽略瘤周微环境的分子信息);② 聚焦单一分子靶点(如仅预测PD-L1表达),缺乏多分子特征的同时预测;③ 模型可解释性差,难以获得临床信任。
本文的创新点在于突破传统研究的“单区域、单特征、单分子”局限:① 纳入多中心队列(3个机构508例患者),提升模型泛化性;② 融合肿瘤+瘤周区域的影像学信息(瘤周区域通过算法自动生成);③ 整合手工放射组学特征(Radiomics-Score)与深度网络特征(Deep-CT),捕捉互补的肿瘤异质性信息;④ 同时预测10种临床关键分子特征(3种预后因子、6种基因突变、1种免疫表型),覆盖靶向与免疫治疗的核心决策需求。
3. 研究思路总结与详细解析
3.1 患者队列与数据收集
实验目的:构建多中心NSCLC患者队列,获取CT图像、临床病理及分子特征数据。
方法细节:回顾性纳入508例NSCLC患者(Cohort I:370例,来自赣南医学院第一附属医院;Cohort II:138例,来自美国退伍军人事务医疗系统及斯坦福大学),合并后按7:3比例分为训练集(n=356)与独立测试集(n=152)。收集数据包括:① CT图像(平扫/增强);② 临床病理特征(LVI、PI、T分期);③ 基因突变状态(EGFR、KRAS、ALK、TP53、PIK3CA、ROS1);④ PD-1/PD-L1表达(Cohort I通过免疫组化(IHC)检测,Cohort II通过CD8+T细胞浸润替代)。
结果解读:队列人口学与临床特征均衡(见表1),确保模型训练的代表性。
3.2 CT图像预处理与ROI分割
实验目的:获取准确的肿瘤及瘤周区域感兴趣区(ROI),为特征提取奠定基础。
方法细节:① Cohort I的CT图像采用3D Slicer手动分割:由5年经验的临床医生完成初分割,10年经验的放射科医生审核修正,分歧通过共识解决;② Cohort II的CT图像与ROI来自公共数据库(如TCIA);③ 瘤周区域通过内部算法生成:以肿瘤边界为基准,向外扩展3mm形成环形区域(兼顾肿瘤浸润特性与图像分辨率);④ 深度ROI构建:选取肿瘤最大截面,生成160×160像素的方形斑块(聚焦肿瘤核心特征)。
结果解读:成功获取三类ROI(肿瘤、瘤周、深度),为多区域特征提取提供了数据基础。
3.3 深度学习放射组学模型构建
3.3.1 Deep-CT深度网络特征提取
实验目的:选择最优深度网络,自动提取肿瘤的语义特征。
方法细节:比较5种经典深度网络(ResNet50、ResNet101、DenseNet121、VGG19、Inception V3)在LVI预测中的性能,以曲线下面积(AUC)为核心指标。网络结构调整:将DenseNet121的全连接层(原1000节点)替换为2层Xavier初始化的全连接层,倒数第二层输出作为深度网络特征(保留高维语义信息)。
结果解读:DenseNet121在测试集的AUC最高(0.792),显著优于其他模型(如ResNet50为0.676、VGG19为0.679),因此选作Backbone网络,用于后续所有分子特征预测。
3.3.2 Radiomics-Score手工特征提取
实验目的:提取肿瘤与瘤周区域的手工放射组学特征,构建传统放射组学模型。
方法细节:对每个ROI提取851个定量特征(包括形状、直方图、纹理、滤波器特征),通过Z-Score归一化消除量纲差异。采用LASSO回归进行特征选择(保留与分子特征强相关的关键特征),计算Radiomics-Score(关键特征的线性组合,权重由模型训练获得)。
结果解读:肿瘤区域Radiomics-Score预测LVI的AUC为0.842(95%CI 0.832-0.852),瘤周区域Radiomics-Score预测PI的AUC为0.853(95%CI 0.844-0.862),显示手工特征对分子表型的预测价值。
3.3.3 Deep-RadScore多特征融合模型
实验目的:融合手工特征与深度特征,提升模型预测性能。
方法细节:将肿瘤+瘤周的Radiomics-Score与Deep-CT的深度特征输入融合层(全连接层 concatenate 后输入分类器),比较7种分类算法(支持向量机SVM、随机森林RF、逻辑回归LR等)的性能。采用5折交叉验证(10次重复)优化模型参数,选择AUC最高的分类器作为最终模型。
结果解读:融合后的Deep-RadScore性能显著优于单一模型(Radiomics-Score或Deep-CT)。例如,预测LVI的AUC从Radiomics-Score的0.842提升至0.889(P<0.05,DeLong检验),预测PD-1/PD-L1表达的AUC从Deep-CT的0.839提升至0.893(见表3)。
3.4 模型性能评估与验证
实验目的:验证模型在独立测试集的泛化能力。
方法细节:采用AUC、准确率(ACC)、召回率、F1 score评估模型性能,比较Deep-RadScore与Radiomics-Score、Deep-CT的差异。
结果解读:Deep-RadScore在所有分子特征预测中表现最优(见图2、图3):
- 预后风险因子:LVI(AUC=0.889)、PI(AUC=0.903)、T分期(AUC=0.894);
- 基因突变:EGFR(AUC=0.884)、KRAS(AUC=0.896)、ALK(AUC=0.884)、TP53(AUC=0.889)、PIK3CA(AUC=0.896)、ROS1(AUC=0.895);
- 免疫表型:PD-1/PD-L1(AUC=0.893)。
所有结果均通过独立测试集验证,证明模型的鲁棒性与泛化性。
3.5 相关性分析与模型可解释性
实验目的:分析手工特征与深度特征的相关性,提升模型可解释性。
方法细节:① 用Pearson相关系数分析Radiomics-Score与Deep-CT特征的相关性;② 用SHAP算法可视化特征对预测结果的贡献;③ 用Grad-CAM生成热图,展示模型关注的图像区域。
结果解读:
- 相关性弱:手工特征与深度特征的平均绝对相关系数仅0.113(PD-1/PD-L1),说明两者捕捉了互补的肿瘤异质性(见图4a);
- 特征贡献:Deep-CT特征对基因突变(如ROS1)的贡献更大,Radiomics-Score对预后因子(如LVI)的贡献更显著(见图4b);
- 区域关注:Grad-CAM热图显示,模型主要关注肿瘤核心区域及瘤周浸润带(与临床病理表现一致),提升了模型的临床可信度。
4. Biomarker研究及发现成果解析
Biomarker定位与筛选逻辑
本文的Biomarker是多源融合的影像学特征,包括三类:
1. Radiomics-Score:肿瘤与瘤周区域的手工放射组学特征(形状、纹理等),通过LASSO回归筛选关键特征;
2. Deep-CT特征:DenseNet121提取的深度语义特征(高维、非线性);
3. Deep-RadScore:Radiomics-Score与Deep-CT特征的融合结果(多模态、多区域)。
筛选逻辑:多中心训练+交叉验证+独立测试——在训练集通过5折交叉验证优化特征权重,独立测试集验证泛化能力,比较7种分类器选择最优模型。
研究过程详述
- Biomarker来源:CT图像的肿瘤、瘤周及深度ROI;
- 验证方法:① 分类性能评估(AUC、ACC等);② 相关性分析(Pearson系数);③ 可解释性可视化(SHAP、Grad-CAM);
- 性能数据:Deep-RadScore在独立测试集的核心性能(n=152):
- 预后因子:LVI(AUC=0.889,95%CI 0.881-0.897)、PI(AUC=0.903,95%CI 0.896-0.910);
- 基因突变:EGFR(AUC=0.884,95%CI 0.873-0.895)、KRAS(AUC=0.896,95%CI 0.887-0.905);
- 免疫表型:PD-1/PD-L1(AUC=0.893,95%CI 0.882-0.905)。
核心成果提炼
- 多特征融合的优势:Deep-RadScore的AUC较单一特征模型提升0.039-0.070(如LVI预测从0.842提升至0.889),证明手工特征与深度特征的互补性;
- 多分子特征覆盖:首次实现NSCLC患者10种临床关键分子特征的同时预测(覆盖靶向/免疫治疗的核心决策点);
- 临床转化价值:模型基于常规CT图像,无需额外检查,可无创、实时提供分子信息,解决了传统活检的局限性;
- 可解释性提升:通过相关性分析与可视化工具,明确了特征的生物学意义(如深度特征关注肿瘤语义信息,手工特征关注形态异质性),为临床信任提供了依据。
关键图片展示
图1:研究设计框架
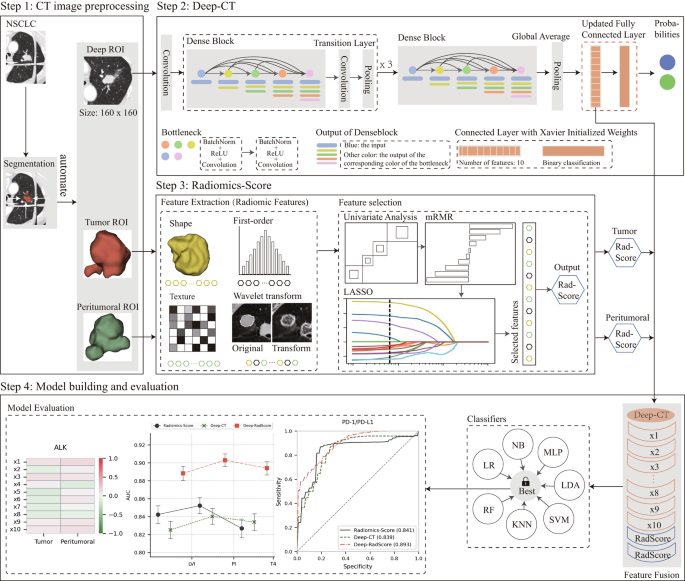
图2:模型预测预后因子与基因突变的ROC曲线
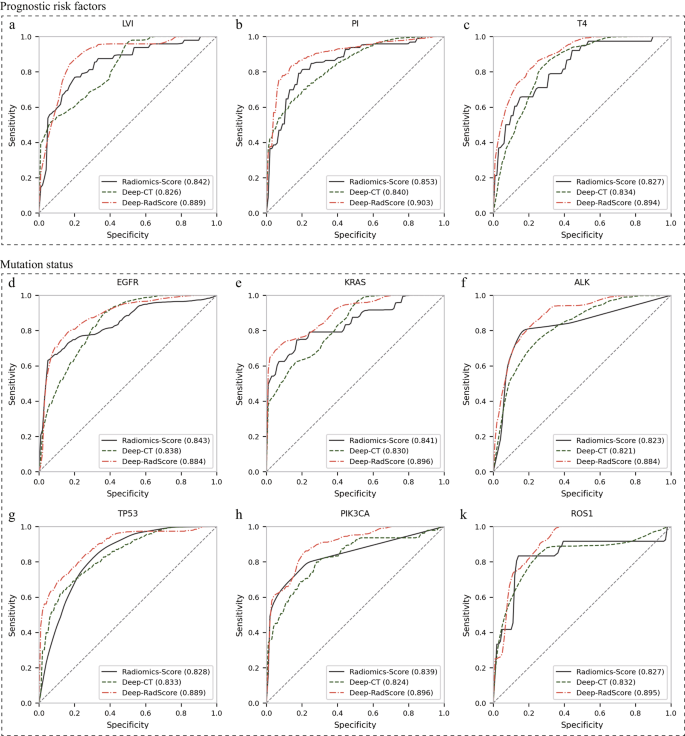
图3:模型预测性能的AUC分布
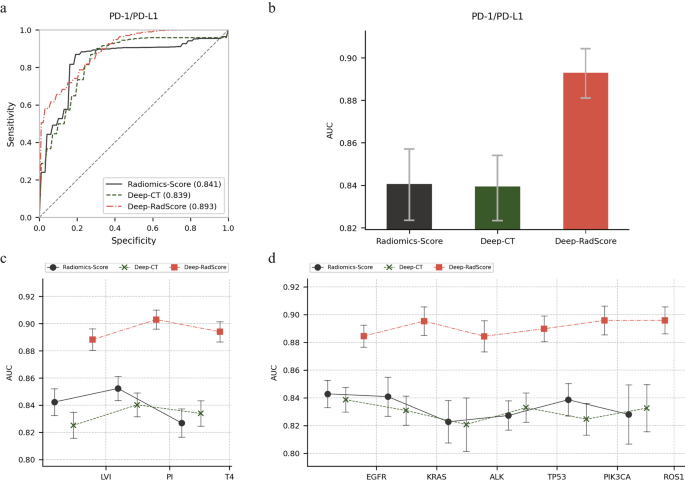
图4:特征相关性与贡献分析
结论
本文开发的Deep-RadScore模型,通过多区域、多特征融合,实现了NSCLC患者多分子特征的无创预测,为精准治疗决策提供了重要工具。研究突破了传统放射组学的局限,证明了深度学习与多源融合在肿瘤分子表型检测中的价值,为NSCLC的无创精准医疗提供了新范式。未来需通过前瞻性研究进一步验证模型的临床转化价值。
