1. 领域背景与文献引入
文献英文标题:Bridging PCR and partially overlapping primers for novel allergen gene cloning and expression insert decoration;发表期刊:World Journal of Gastroenterology;影响因子:未公开;研究领域:变态反应学(过敏原基因克隆与分子表达技术细分领域)。
变态反应性疾病(如哮喘、花粉过敏、食物过敏)是工业化国家近20年发病率快速上升的公共健康问题,但其有效治疗受限于花粉与食物过敏的交叉反应机制未完全阐明。重组过敏原是过敏诊断与免疫治疗的核心工具,但生产重组过敏原需首先获取完整的过敏原基因开放阅读框(ORF),而传统技术路径存在两大瓶颈:一是全长基因获取困难——通过cDNA文库筛选或回到原始cDNA池的方法,易产生多态性克隆,耗时且效率低;二是表达插入片段修饰的非特异性扩增——为将基因插入载体需设计含酶切位点或载体序列的长引物,其高Tm值(常>72℃)会导致PCR非特异性扩增,增加后续筛选成本。针对这些问题,本研究旨在开发bridging PCR(BPCR)与部分重叠引物PCR(POP-PCR)的组合技术,解决全长过敏原基因克隆及表达插入片段修饰的关键问题,为重组过敏原的高效生产提供技术支撑。
2. 文献综述解析
作者围绕“全长基因获取”与“表达插入片段修饰”两个核心技术问题,对现有研究进行分类评述:
现有研究中,全长基因获取的主流方法(如cDNA文库筛选、原始cDNA池PCR)虽能得到基因序列,但易产生多态性克隆,需大量筛选工作,导致时间与成本过高;表达插入片段修饰环节,长引物(含载体或酶切位点序列)的高Tm值会引发非特异性扩增,增加错误插入的风险。本研究的创新在于针对性解决两大技术痛点:BPCR利用基因片段的同源区域直接拼接全长基因,避免多态性克隆;POP-PCR通过“长短引物部分重叠”的设计,降低长引物的非特异性扩增,从而建立“全长基因克隆-表达插入片段修饰”的高效技术体系,弥补了现有方法的不足。
3. 研究思路总结与详细解析
本研究以“建立高效全长过敏原基因克隆及表达插入片段修饰方法”为目标,核心科学问题是解决传统方法中“全长基因获取困难”与“非特异性扩增”的问题,技术路线遵循“基因片段获取→BPCR拼接全长→POP-PCR修饰表达插入片段→功能验证”的闭环逻辑。
3.1 基因片段获取与验证
实验目的是获取短豚草(Ambrosia artemisiifolia L.)花粉过敏原基因的5’端与3’端片段。方法为:采集短豚草花粉,液氮研磨后用RNeasy Maxi kit(Qiagen)提取总RNA,通过ProtoScript first strand cDNA synthesis kit(New England Biolabs)将2μg总RNA反转录为cDNA;利用泛简并引物PCR(引物Sg1P5/S3D10)扩增5’端片段A,通过快速cDNA末端扩增(RACE)技术(引物S5D106/RAC3)获取3’端片段B。结果显示,片段A长度为363 bp,片段B为431 bp,两者存在126 bp的同源区域,为后续BPCR拼接全长基因提供了关键序列基础。实验所用关键产品:RNeasy Maxi kit(Qiagen)、ProtoScript first strand cDNA synthesis kit(New England Biolabs)。
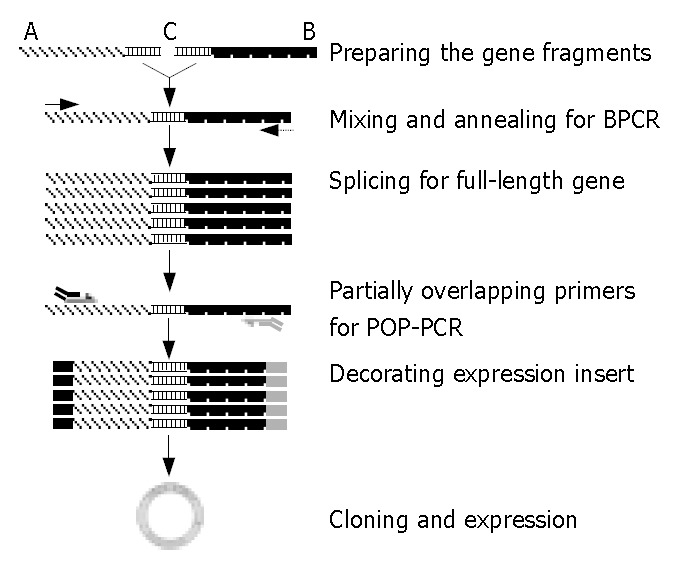
3.2 BPCR拼接全长过敏原基因
实验目的是利用片段A与B的同源区域拼接全长cDNA。方法为:将片段A与B等体积混合并稀释15倍作为模板,使用HotstarTaq DNA polymerase(Qiagen)进行BPCR,引物为Sg1P5/RAC3;反应条件为95℃15 min激活酶,随后以-0.1℃/s降至66℃并延伸5 min,进行7个touchdown循环(94℃45 s、68℃30 s、72℃30 s,退火温度每循环降1℃),再进行30个常规循环(94℃45 s、66℃30 s、72℃2 min);产物经12 g/L琼脂糖凝胶电泳后,用QIAquick gel extraction kit(Qiagen)回收,克隆至pGEM-T easy vector(Promega)并转化JM109感受态细胞,测序后通过GenBank BLAST分析同源性。结果显示,拼接得到的全长cDNA序列(GenBank登录号AY268426)编码131个氨基酸的多肽,与花粉来源肌动蛋白抑制蛋白(profilin)的同源性为54%-89%,与食物来源profilin的同源性为79%-89%,证明该基因为profilin家族成员,命名为Amb a 8(D106)。实验所用关键产品:HotstarTaq DNA polymerase(Qiagen)、pGEM-T easy vector(Promega)、QIAquick gel extraction kit(Qiagen)。

3.3 POP-PCR修饰表达插入片段
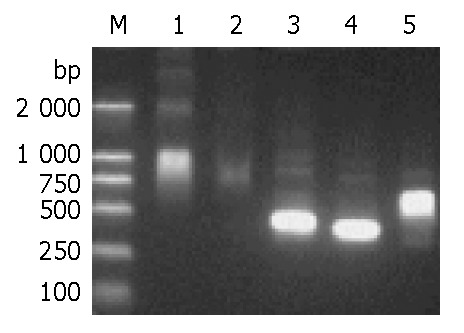
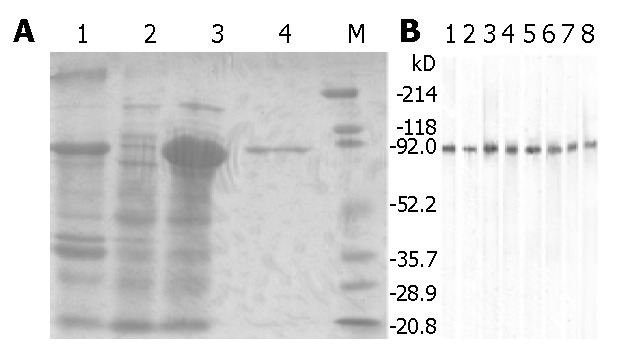
实验目的是解决长引物导致的非特异性扩增问题,为表达载体插入修饰基因片段。方法为:根据Amb a 8(D106)的ORF序列与pET-44 EK/LIC载体的要求,设计长引物(如POP51/POP31,Tm>72℃)及与其5’端序列一致的短引物(如POP52/POP32),按不同摩尔比(1:1、1:10等)混合形成部分重叠引物;以BPCR产物为模板进行POP-PCR,反应条件与BPCR类似,但调整退火温度与循环参数;产物经凝胶回收后克隆至pET-44 EK/LIC载体,转化NovaBlue Singles competent cells(Novagen)并测序验证。结果显示,当长短引物摩尔比为1:10(正向)与1:1(反向)时,POP-PCR产生清晰的预期大小条带(图5),而常规长引物PCR出现smear;测序确认插入片段读框正确,且两端含载体特异性序列,成功构建表达载体。实验所用关键产品:pET-44 EK/LIC载体(Novagen)、NovaBlue Singles competent cells(Novagen)。
3.4 基因功能与过敏原性验证
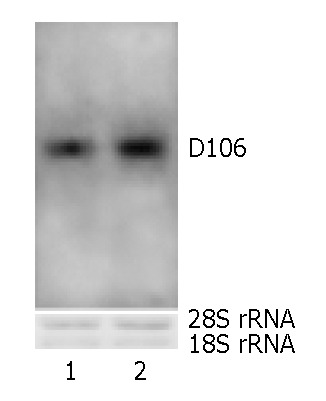
实验目的是验证Amb a 8(D106)的RNA来源及重组蛋白的过敏原性。方法包括:(1)Northern blot验证RNA来源:提取花萼/花梗与花粉的总RNA,经甲醛凝胶电泳后转移至尼龙膜,用Psoralen-biotin标记的D106探针杂交,通过Phototope star检测 kit(Ambion)显影;(2)重组蛋白表达:将表达载体转化BL21(DE3)感受态细胞,用0.5 mmol/L IPTG诱导2 h(30℃),通过150 g/L SDS-PAGE分析表达情况;(3)免疫印迹(Western blot)验证过敏原性:用S·Tag thrombin purification kit(Novagen)纯化重组蛋白,经SDS-PAGE转移至PVDF膜,用过敏患者血清(n=8,IgE>3.5 kU_A/L)孵育后,加入辣根过氧化物酶标记的羊抗人IgE(Sigma-Aldrich),DAB显色底物(Amersco)显影。结果显示:Northern blot中花粉与花萼/花梗RNA均出现杂交条带(图4),证明D106的RNA真实来源;重组蛋白分子量约70 kDa(与预期一致),且能与过敏患者血清IgE结合(图6),而正常对照血清(n=13)无反应,证明其过敏原性。实验所用关键产品:S·Tag thrombin purification kit(Novagen)、peroxidase conjugated goat anti-human IgE(Sigma-Aldrich)、DAB chromogenic substrate solution(Amersco)。
4. Biomarker研究及发现成果解析
本研究中的Biomarker为短豚草花粉过敏原基因Amb a 8(D106)编码的profilin家族蛋白,其筛选与验证逻辑遵循“基因片段获取→BPCR拼接全长→同源性分析→RNA来源验证→过敏原性验证”的完整链条。
Biomarker定位与验证
Biomarker来源为短豚草花粉的cDNA,验证方法包括:(1)同源性分析:通过GenBank BLAST显示,D106与花粉来源profilin的同源性为54%-89%,与食物来源profilin的同源性为79%-89%,符合“泛过敏原(panallergen)”的特征(可介导花粉与食物过敏的交叉反应);(2)RNA来源验证:Northern blot显示花粉与花萼/花梗均有D106的RNA表达,排除PCR假阳性;(3)过敏原性验证:免疫印迹中,8例短豚草过敏患者血清(n=8)均能与重组蛋白结合,而13例正常对照无反应,证明其能诱导IgE介导的过敏反应。
核心成果提炼
Amb a 8(D106)是新的短豚草花粉过敏原,属于profilin家族,可作为花粉与食物过敏交叉反应研究的Biomarker;同时,本研究建立的BPCR与POP-PCR组合方法,解决了全长过敏原基因克隆与表达插入片段修饰的关键技术问题,为重组过敏原的高效生产提供了技术参考。其中,重组蛋白的过敏原性数据显示:对过敏患者血清的敏感性为100%(8/8),对正常对照的特异性为100%(13/13)(文献未明确提供统计学显著性P值,但样本量n=8(患者)与n=13(对照))。
综上,本研究不仅发现了新的花粉过敏原Biomarker,更提供了“全长基因克隆-表达修饰”的高效技术方案,为过敏诊断与免疫治疗的基础研究提供了重要支撑。
