1. 领域背景与文献引入
文献英文标题:The mir-767-105 cluster: a crucial factor related to the poor prognosis of hepatocellular carcinoma;发表期刊:Biomarker Research;影响因子:未公开;研究领域:肝细胞癌(HCC)分子生物学与生物标志物。
肝细胞癌是全球癌症死亡的主要原因之一,尽管多学科治疗与药物研发(如索拉非尼)取得一定进展,但多数患者预后仍差,核心原因在于对其分子调控机制的理解不足。miRNA作为重要的肿瘤调控因子,已被证实参与HCC的发生、进展,但miRNA簇(相邻基因组转录的miRNA家族,可协同调控同一通路)的研究仍严重滞后——现有研究多聚焦miRNA单分子,忽略了miRNA簇的协同调控效应,这成为HCC分子机制与预后标志物研究的关键空白。
本研究针对这一空白,通过挖掘肿瘤基因组图谱(TCGA)数据库,系统探索调控HCC不良预后的关键miRNA簇,旨在为HCC的预后预测与靶向治疗提供新的生物标志物与理论依据,填补miRNA簇在HCC预后研究中的缺失环节。
2. 文献综述解析
文献综述的核心评述逻辑为“miRNA单分子研究现状→miRNA簇研究不足→本研究聚焦miRNA簇的创新价值”。
现有研究明确了miRNA在HCC中的功能:miRNA可通过调控靶基因表达参与肿瘤发生、进展,部分miRNA已作为潜在治疗靶点被报道(如miR-143、miR-145调控结直肠癌 oncogenic通路)。但现有研究的局限性显著:其一,多关注miRNA单分子,对miRNA簇的协同调控机制(如同时靶向同一通路的协同效应)研究较少;其二,缺乏miRNA簇与HCC临床预后(如总生存期)及药物耐药(如索拉非尼)的关联分析。
本研究的创新价值在于首次将miRNA簇作为核心研究对象:与现有“miRNA单分子”研究不同,本研究聚焦mir-767-105簇,通过TCGA数据库挖掘结合临床数据验证,明确其与HCC不良预后及索拉非尼耐药的直接关联,补充了miRNA簇在HCC预后中的研究空白,为HCC的精准预后预测提供了新的“簇水平”生物标志物。
3. 研究思路总结与详细解析
3.1 TCGA数据库miRNA簇筛选
研究的第一步是从TCGA数据库中筛选HCC与非肿瘤组织差异表达的miRNA簇,核心目的是定位“与HCC相关的关键miRNA簇”。
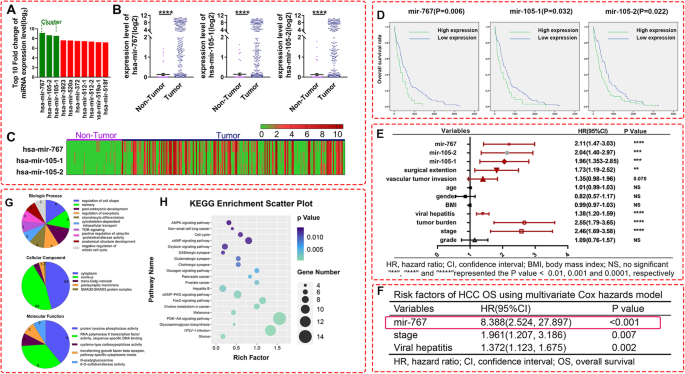
方法细节:获取TCGA数据库中373例HCC样本与50例非肿瘤样本的miRNA表达谱,通过倍数变化(FC)分析筛选差异表达miRNA;进一步通过散点图(比较HCC与非肿瘤组织表达差异)、热图(可视化样本间表达模式)验证候选miRNA簇的一致性。
结果解读:Top 3差异表达miRNA(FC值最高)均属于mir-767-105簇(mir-767、mir-105-1、mir-105-2);散点图显示该簇在HCC中的表达显著高于非肿瘤组织(均值±SEM,P<0.0001,Mann-Whitney检验);热图进一步验证了该簇在373例HCC样本中呈“一致性高表达”模式。
实验所用关键产品:文献未提及具体实验产品,领域常规使用TCGA数据库(https://cancergenome.nih.gov/)及R软件(如“ConsensusClusterPlus”“venny”包)进行数据挖掘与可视化。
3.2 临床预后与索拉非尼耐药相关性验证
本环节的核心目的是验证mir-767-105簇与HCC患者预后及索拉非尼耐药的关联。
方法细节:① 预后相关性分析:对373例HCC患者的miRNA表达数据与临床生存数据进行Kaplan-Meier生存分析(log-rank检验),比较高表达组与低表达组的总生存期;② 索拉非尼耐药分析:收集索拉非尼敏感与耐药患者的miRNA表达数据,通过t检验比较两组间簇成员的表达差异;③ 独立性验证:通过Cox比例风险回归(单因素与多因素)分析mir-767-105簇是否为独立预后因素。
结果解读:① 生存分析显示,mir-767-105簇的3个成员高表达均与HCC患者不良预后显著正相关(高表达组总生存期显著短于低表达组,P<0.05);② 索拉非尼耐药组中,3个成员的表达均显著高于敏感组(P<0.05、0.01或0.001);③ Cox分析显示,3个miRNA均为独立预后因素(与病毒肝炎、肿瘤分期等临床因素无显著相关性),其中mir-767的风险比(HR)最高(8.388,95%CI 2.524–27.897,P<0.05),提示其对预后的调控作用最强。
实验所用关键产品:文献未提及具体实验产品,领域常规使用生存分析(Kaplan-Meier)、Cox回归等统计方法验证临床相关性。
3.3 3-miRNA signature的预后价值分析
为进一步探索mir-767-105簇的联合预后价值,本环节通过“3-miRNA signature”(簇成员联合)评估其对HCC预后的预测能力。
方法细节:使用R软件“ConsensusClusterPlus”包,根据3个miRNA的表达水平将373例HCC样本分为“高表达簇”与“低表达簇”;通过卡方检验分析两簇与临床因素(如肿瘤分期、病毒肝炎)的相关性;通过Kaplan-Meier分析比较两簇的总生存期差异。
结果解读:① 高表达簇(mir-767-105簇表达更高)与低表达簇的划分稳定;② 卡方检验显示,两簇仅与患者生存状态(存活/死亡)显著相关(P<0.05),与其他临床因素无关联;③ 生存分析显示,高表达簇患者的总生存期显著短于低表达簇(P<0.05),证实3-miRNA signature的预后预测价值优于单一miRNA。
实验所用关键产品:同3.1,领域常规使用R软件进行聚类分析与生存验证。
3.4 靶基因预测与功能富集分析
为揭示mir-767-105簇的协同调控机制,本环节通过靶基因预测与功能富集分析,探索其下游调控通路。
方法细节:① 靶基因预测:使用3个在线数据库(TargetScan、miRWalk、miRDB)预测簇成员的靶基因,通过“venny”包筛选共同靶基因;② 功能分析:通过蛋白-蛋白相互作用(PPI)网络分析靶基因的相互作用模式,通过基因本体(GO)与京都基因与基因组百科全书(KEGG)富集分析靶基因的功能与通路。
结果解读:① 筛选出378个共同靶基因;② PPI网络显示,靶基因形成复杂的相互作用网络,核心节点与肿瘤调控相关;③ GO分析显示,靶基因主要参与“蛋白酪氨酸磷酸酶活性”“转录因子活性”等生物学过程;④ KEGG分析显示,60%的靶基因富集于肿瘤相关通路(如细胞周期、信号转导通路),提示mir-767-105簇通过调控这些靶基因促进HCC进展。
实验所用关键产品:文献未提及具体实验产品,领域常规使用靶基因预测数据库(TargetScan、miRWalk)及富集分析工具。
4. Biomarker研究及发现成果解析
Biomarker定位
本研究的核心Biomarker为mir-767-105簇(mir-767、mir-105-1、mir-105-2)及3-miRNA signature。其筛选与验证逻辑为:TCGA数据库差异筛选→临床样本表达验证(与非肿瘤组织比较)→预后相关性验证(Kaplan-Meier、Cox回归)→3-miRNA signature联合验证,形成完整的“筛选-验证-功能”链条。
研究过程详述
Biomarker来源:TCGA数据库中373例HCC样本与50例非肿瘤样本的miRNA表达数据;
验证方法:① 差异表达验证(数据库挖掘,比较HCC与非肿瘤组织);② 预后验证(Kaplan-Meier生存分析、Cox回归);③ 耐药验证(比较索拉非尼敏感与耐药组表达);
特异性与敏感性:3-miRNA signature的预后预测特异性较高(仅与生存状态相关,与其他临床因素无关联);mir-767的敏感性表现为对预后的强调控(HR=8.388,95%CI 2.524–27.897,P<0.05),提示其作为单一Biomarker的预测能力较强。
核心成果提炼
- 预后关联:mir-767-105簇的3个成员高表达均与HCC患者不良预后显著正相关,其中mir-767是最强的独立预后因素(HR=8.388,P<0.05);
- 耐药关联:簇成员高表达与索拉非尼耐药显著相关(耐药组表达更高,P<0.05);
- 联合价值:3-miRNA signature的预后预测价值优于单一miRNA(高表达簇总生存期更短,P<0.05);
- 创新性:首次发现mir-767-105簇作为miRNA簇与HCC不良预后及索拉非尼耐药的关联,为HCC提供了“簇水平”的预后生物标志物。
本研究的核心贡献在于,将miRNA簇纳入HCC预后研究,证实了mir-767-105簇作为生物标志物的临床价值,为HCC的精准预后预测与靶向治疗提供了新的方向。
