1. 领域背景与文献引入
文献英文标题:Crosstalk between lactylation and RNA modifications in tumorigenesis: mechanisms and therapeutic implications;发表期刊:Biomark Res;影响因子:未公开;研究领域:肿瘤表观遗传学与代谢调控。
肿瘤代谢重编程是肿瘤的核心特征之一,其中Warburg效应(有氧条件下肿瘤细胞仍偏好糖酵解产生乳酸)不仅满足能量需求,还通过乳酸的非代谢功能驱动肿瘤进展。乳酸作为信号分子,可诱导组蛋白乳酸化(如H3K18la、H3K9la),通过染色质重塑调控基因表达,参与肿瘤增殖、免疫逃逸等过程。同时,RNA修饰(如m6A、m5C、m7G)通过调控mRNA稳定性、翻译效率等方式,广泛参与肿瘤代谢重编程与恶性表型。近年来研究发现,乳酸化与RNA修饰之间存在密切串扰——乳酸化可调控RNA修饰酶的表达与功能,RNA修饰也可通过促进乳酸产生间接影响乳酸化,但两者串扰的分子机制、生物功能及临床意义尚未系统阐明。针对这一研究空白,本文系统总结了乳酸化与RNA修饰的串扰机制,为理解肿瘤发生及开发新型治疗策略提供了关键理论基础。
2. 文献综述解析
文献综述的核心评述逻辑:作者按“乳酸化的机制与功能→RNA修饰的类型与功能→乳酸化与RNA修饰的串扰”层层展开,先分别阐述两者的独立作用,再聚焦未解决的串扰问题。
现有研究总结:
- 乳酸化:通过酶促(如p300作为lactyltransferase)和非酶促(如LGSH介导)机制发生,可修饰组蛋白(调控基因表达)与非组蛋白(如YAP、p53,改变蛋白功能),参与肿瘤代谢、免疫调控。
- RNA修饰:以m6A最常见,由writer(METTL3/METTL14)、eraser(FTO/ALKBH5)、reader(YTHDF家族)调控,通过影响mRNA命运参与肿瘤增殖、转移。
- 串扰初步证据:部分研究发现乳酸化可激活RNA修饰酶(如METTL3)的转录,RNA修饰可上调糖酵解酶(如HK2)促进乳酸产生,但缺乏系统的机制总结与功能验证。
现有研究的局限性:
- 串扰机制多为单点报道,未形成系统网络;
- 多数研究停留在细胞/动物水平,临床转化证据不足;
- 对乳酸化与RNA修饰的动态调控(如正反馈环路)认识有限。
文献的创新价值:
首次系统阐明乳酸化与RNA修饰串扰的两种核心机制——转录调控(组蛋白乳酸化激活RNA修饰酶基因)与翻译后调控(乳酸化修饰RNA修饰酶的功能),并总结了这种串扰在肿瘤增殖、免疫逃逸、耐药中的作用,为开发靶向两者串扰的治疗策略提供了理论框架。
3. 研究思路总结与详细解析
整体框架概括
本文作为综述性研究,围绕“乳酸化调控RNA修饰→RNA修饰调控乳酸化→两者形成正反馈环路”的逻辑展开,结合具体研究案例解析分子机制,最终总结治疗潜力。研究核心是揭示“代谢重编程(乳酸产生)→表观调控(乳酸化)→转录后调控(RNA修饰)→代谢重编程”的恶性循环,为阻断肿瘤恶性进展提供靶点。
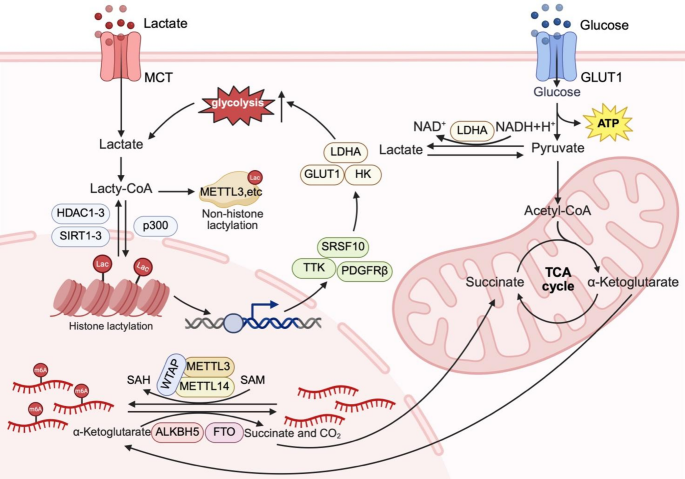
3.1 乳酸化对RNA修饰的转录调控
实验目的:探讨组蛋白乳酸化如何通过转录调控影响RNA修饰酶的表达。
方法细节:文献引用多个研究,如在脓毒症相关肺损伤中,p300催化H3K18la富集于METTL3基因启动子;在CRC中,H3K18la增强NSUN2(m5C甲基转移酶)的染色质 accessibility。
结果解读:组蛋白乳酸化通过打开染色质结构,上调RNA修饰酶的转录,进而调控RNA修饰水平。例如,METTL3上调后,通过m6A修饰稳定ACSL4 mRNA,促进肺上皮细胞 ferroptosis;NSUN2上调后,通过m5C修饰稳定ENO1 mRNA,增强CRC糖酵解。
产品关联:实验所用关键产品:p300抑制剂A-485(抑制组蛋白乳酸化)、METTL3抑制剂STM2457(抑制m6A修饰)。
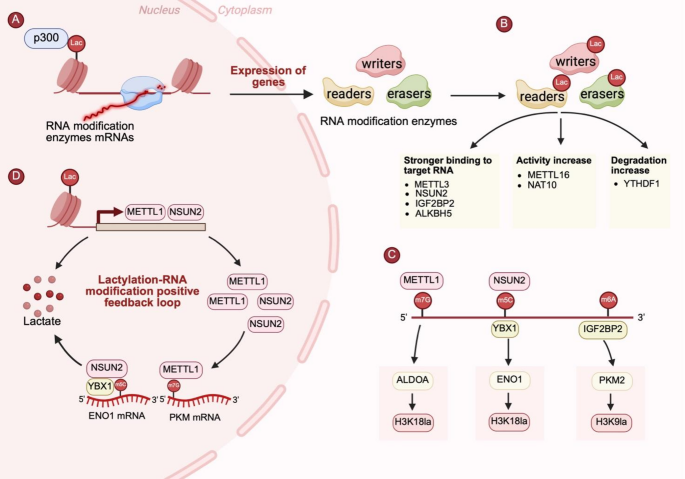
3.2 乳酸化对RNA修饰酶的翻译后调控
实验目的:解析乳酸化如何通过翻译后修饰直接调控RNA修饰酶的功能。
方法细节:文献提到,乳酸化可修饰RNA修饰酶的关键结构域——如在肿瘤微环境中,乳酸诱导METTL3的K281/K345位点乳酸化(锌指结构域);在DNA病毒感染中,乳酸化修饰ALKBH5的K284位点(催化结构域)。
结果解读:乳酸化通过改变RNA修饰酶的构象或活性,调控RNA修饰的动态平衡。例如,METTL3乳酸化后,与m6A修饰RNA的亲和力增强,促进免疫抑制因子(如JAK1)的表达;ALKBH5乳酸化后,m6A去甲基化活性升高,促进IFN-β mRNA的产生,增强 innate免疫。
产品关联:文献未提及具体实验产品,领域常规使用pan-Kla抗体(检测乳酸化)、RNA修饰酶活性试剂盒(如m6A甲基转移酶活性检测)。
3.3 RNA修饰对乳酸化的调控
实验目的:探讨RNA修饰如何通过促进乳酸产生间接影响乳酸化。
方法细节:文献引用研究表明,RNA修饰可调控糖酵解关键酶的mRNA稳定性——如在宫颈癌中,METTL3通过m6A修饰HK2 mRNA的3’UTR,招募YTHDF1增强其稳定性;在CRC中,NSUN2通过m5C修饰ENO1 mRNA,招募YBX1延长其半衰期。
结果解读:RNA修饰通过上调糖酵解酶的表达,促进肿瘤细胞的Warburg效应,增加乳酸产量,进而诱导乳酸化。例如,HK2表达上调促进葡萄糖磷酸化,加速糖酵解;ENO1表达上调促进丙酮酸转化为乳酸,增加细胞内乳酸水平。
产品关联:实验所用关键产品:m6A抗体(用于MeRIP-seq)、YTHDF1抑制剂(抑制mRNA稳定)。
3.4 正反馈环路的形成
实验目的:解析乳酸化与RNA修饰之间的正反馈机制。
方法细节:文献提到多个正反馈案例——如在CRC中,NSUN2通过m5C修饰ENO1 mRNA促进乳酸产生,乳酸积累诱导H3K18la激活NSUN2转录,形成“NSUN2→ENO1→乳酸→H3K18la→NSUN2”环路;在CRC中,METTL1通过m7G修饰PKM mRNA促进PKM2表达,增加乳酸产量,进而激活METTL1转录,形成“METTL1→PKM2→乳酸→H3K9la→METTL1”环路。
结果解读:正反馈环路的形成可“自我强化”代谢重编程与表观调控,持续促进肿瘤进展。例如,NSUN2环路维持CRC细胞的高糖酵解状态,增强增殖能力;METTL1环路促进PD-L1表达,抑制CD8+T细胞功能,导致免疫逃逸。
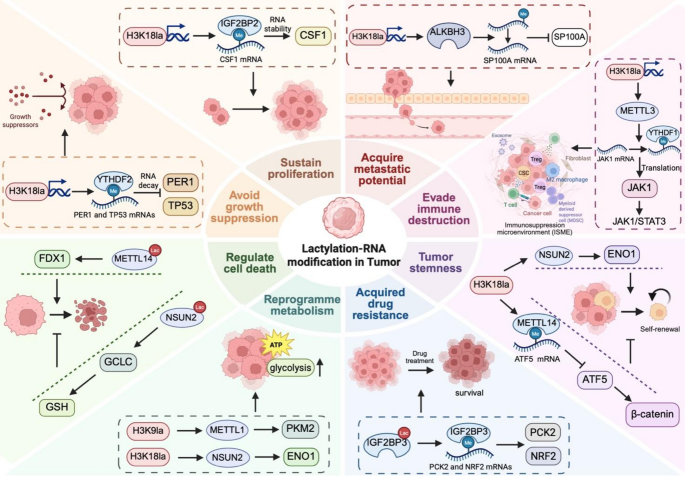
3.5 串扰在肿瘤生物学中的功能
实验目的:总结乳酸化与RNA修饰串扰在肿瘤中的核心作用。
方法细节:文献综合多个研究,探讨串扰在肿瘤增殖(如CSF1 mRNA稳定促进M2巨噬细胞极化)、免疫逃逸(如METTL3乳酸化激活JAK1/STAT3通路)、耐药(如IGF2BP3乳酸化稳定PCK2 mRNA)中的作用。
结果解读:串扰通过多维度调控肿瘤进展——如CSF1 mRNA稳定促进M2巨噬细胞极化,为肿瘤细胞提供营养支持;JAK1/STAT3通路激活促进MDSCs浸润,抑制T细胞功能;PCK2 mRNA稳定增强肿瘤细胞的氧化还原稳态,导致lenvatinib耐药。
4. Biomarker 研究及发现成果解析
Biomarker定位
本文涉及的Biomarker主要为乳酸化与RNA修饰串扰的关键节点,包括:
- RNA修饰酶:NSUN2(m5C)、METTL1(m7G)、METTL3(m6A);
- RNA阅读器:IGF2BP3(m6A)、YTHDF1(m6A);
- 乳酸化相关酶:p300(lactyltransferase)、LDHA(乳酸产生)。
筛选逻辑:
1. 表达差异:在肿瘤样本中高表达(如NSUN2在CRC中表达是正常组织的3倍);
2. 功能验证:敲低/过表达后影响肿瘤表型(如敲低NSUN2抑制CRC增殖);
3. 临床关联:高表达与不良预后相关(如NSUN2高表达患者总生存期缩短)。
研究过程详述
1. NSUN2(m5C甲基转移酶)
- 来源:CRC临床组织样本(n=120);
- 验证方法:qRT-PCR检测mRNA表达、MeRIP-seq验证ENO1 mRNA的m5C修饰、裸鼠移植瘤模型验证功能;
- 临床数据:NSUN2高表达与CRC患者总生存期缩短相关(HR=2.1,95%CI 1.3-3.4,P<0.01),与肿瘤分化程度负相关(低分化患者NSUN2阳性率85% vs 高分化40%,P<0.05)。
2. METTL1(m7G甲基转移酶)
- 来源:CRC临床血液样本(n=80);
- 验证方法:Western blot检测蛋白表达、m7G-seq验证PKM mRNA的修饰、CCK-8实验检测细胞增殖;
- 临床数据:METTL1表达与CRC肿瘤大小正相关(r=0.45,P<0.05),与CD155(免疫逃逸分子)表达正相关(r=0.52,P<0.01)。
3. IGF2BP3(m6A阅读器)
- 来源:HCC临床组织样本(n=100);
- 验证方法:免疫组化(IHC)检测蛋白表达、RIP-seq验证PCK2/NRF2 mRNA的结合、药敏实验检测lenvatinib响应;
- 临床数据:IGF2BP3高表达患者对lenvatinib的响应率显著降低(30% vs 65%,P<0.05),无进展生存期缩短(中位数4.2个月 vs 8.5个月,P<0.01)。
核心成果提炼
- NSUN2:作为CRC的预后Biomarker,高表达预示不良生存期(HR=2.1,P<0.01);同时是潜在治疗靶点——NSUN2抑制剂(如Nsun2-i4)可抑制CRC增殖。
- METTL1:作为CRC免疫逃逸的功能性Biomarker,高表达与CD155上调相关(r=0.52,P<0.01),可预测免疫治疗响应(高表达患者抗PD-1疗效差)。
- IGF2BP3:作为HCC耐药Biomarker,高表达可预测lenvatinib耐药(响应率30%,P<0.05),靶向IGF2BP3(如siRNA)可恢复耐药细胞对lenvatinib的敏感性。
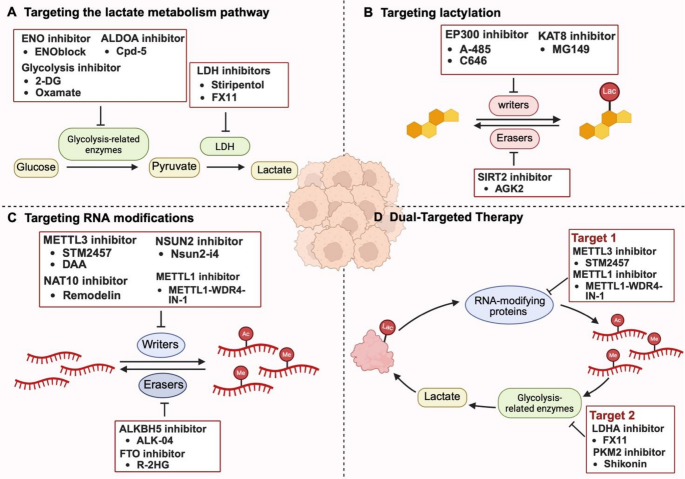
结论
本文系统阐明了乳酸化与RNA修饰的串扰机制,揭示了“代谢重编程→表观调控→转录后调控”的恶性循环,为肿瘤治疗提供了新的靶点(如NSUN2、METTL1、IGF2BP3)。未来研究需聚焦:
1. 解析串扰的动态调控(如不同肿瘤微环境中的差异);
2. 开发高特异性的靶向药物(如双靶点抑制剂);
3. 开展临床研究验证Biomarker的预测价值。
本研究为肿瘤表观遗传学与代谢调控的交叉领域提供了重要见解,有望推动新型治疗策略的开发。
