1. 领域背景与文献引入
文献英文标题:Multi-faceted attributes of salivary cell-free DNA as liquid biopsy biomarkers for gastric cancer detection;发表期刊:Biomarker Research;影响因子:未公开;研究领域:胃癌液体活检、唾液游离DNA生物标志物。
液体活检因非侵入性优势成为癌症诊断的重要方向,其中血浆游离DNA(cfDNA)的突变检测已用于多种癌症,但胃癌因肿瘤异质性高、缺乏特异性驱动突变,循环肿瘤DNA(ctDNA)检测敏感性受限。近年研究发现,cfDNA的非突变特征(如片段组学、末端基序、微生物组贡献)可辅助癌症检测,成为替代策略。唾液作为易获取的生物流体,此前已发现其蛋白、RNA成分可作为胃癌生物标志物,但唾液游离DNA(ScfDNA)的结构特征及临床价值尚未系统解析——现有研究仅关注ScfDNA的单一维度(如片段长度),未综合分析其多维度特征(片段组学、末端基序、微生物来源、功能元件)在胃癌中的差异。本研究旨在通过“Broad-Range 游离DNA测序(BRcfDNA-seq)”技术,系统分析ScfDNA的多维度特征,探索其作为胃癌液体活检生物标志物的潜力,填补ScfDNA临床应用的研究空白。
2. 文献综述解析
文献综述围绕“血浆cfDNA的应用局限→非突变特征的研究进展→唾液作为生物流体的优势→ScfDNA的研究空白”展开核心评述:
- 现有研究关键结论:血浆cfDNA的非突变特征(如片段长度分布、末端基序)可区分癌症与健康人群;唾液的蛋白(如CSTB、DMBT1)和RNA(如外泌体RNA)已被验证为胃癌生物标志物;但ScfDNA的结构特征(如多峰片段分布、末端基序多样性)及临床价值未被系统报道。
- 现有研究局限性:缺乏对ScfDNA多维度特征的综合分析,尤其未探索其在胃癌中的差异;传统文库制备方法难以捕获ScfDNA的多种构象(单链、双链、带缺口),限制了特征解析的完整性。
- 本研究创新点:首次采用BRcfDNA-seq技术(单链文库制备)整合ScfDNA的多种构象,系统分析其片段组学、末端基序、微生物组、功能元件等多维度特征;首次验证ScfDNA多维度特征对胃癌的区分能力,为其作为液体活检生物标志物提供依据。
3. 研究思路总结与详细解析
本研究以“探索ScfDNA多维度特征及胃癌区分能力”为目标,核心科学问题是“ScfDNA的多维度特征能否有效区分胃癌与非癌样本”,技术路线为“样本收集→ScfDNA提取→BRcfDNA-seq文库制备→多维度特征解析→统计验证”的闭环设计。
3.1 样本收集与ScfDNA提取
实验目的:获取合格的唾液样本及ScfDNA。
方法细节:纳入10例胃癌患者(经内镜确诊)和10例非癌志愿者(胃镜示胃炎),按标准操作流程(SOP)收集唾液,4℃ 2600×g离心15分钟取上清,再10000×g离心15分钟去除残留细胞;用Qiagen QIAmp循环核酸试剂盒提取1mL唾液中的ScfDNA,不添加载体RNA,最终洗脱体积20μL。
结果解读:电泳显示ScfDNA有两条带——~150bp(单核糖核小体cfDNA)和>1500bp(基因组DNA污染),符合细胞游离DNA的典型特征(补充图1A)。
实验所用关键产品:Qiagen的QIAmp Circulating Nucleic Acid Kit(货号55114)、Qiagen的ATL Lysis buffer(货号19076)。
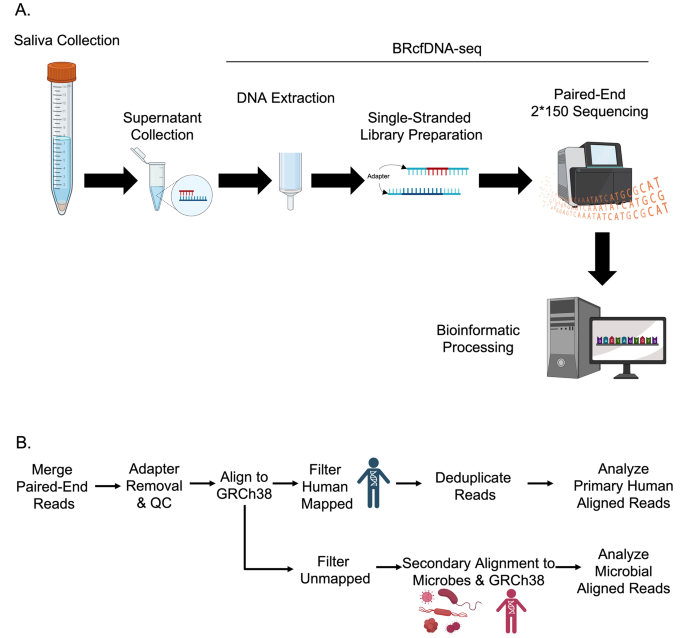
3.2 ScfDNA构象分析
实验目的:明确ScfDNA的分子构象(单链、双链、带缺口)。
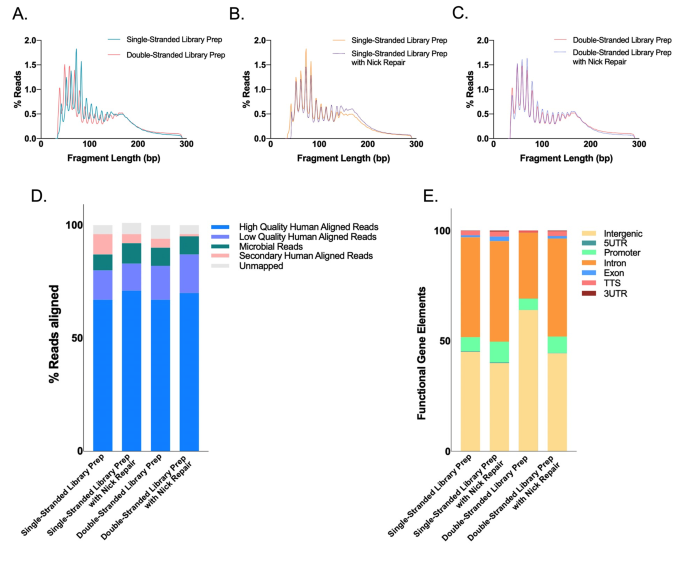
方法细节:用链特异性核酸酶处理健康人ScfDNA——Arcticzyme双链DNA酶(降解双链DNA)、Exonuclease 1(降解单链DNA)、PreCR修复酶(修复带缺口DNA),随后制备单链/双链文库,电泳分析片段分布。
结果解读:经不同酶处理后,ScfDNA电泳显示300bp和200bp条带(对应~160bp单核糖核小体cfDNA和~50bp超短cfDNA,扣除160bp文库接头),表明ScfDNA存在单链、双链、带缺口的多种构象(补充图1B)。
实验所用关键产品:ArcticZyme的dsDNase(货号70600-201)、NEB的Exonuclease 1(货号M0293S)、NEB的PreCR Repair(货号M0309S)。
3.3 BRcfDNA-seq文库制备与测序
实验目的:构建ScfDNA的单链文库,保留多构象信息。
方法细节:用Claret Bioscience的SRSLYTM PicoPlus试剂盒制备单链文库——取18μL ScfDNA进行热休克,采用低分子权重保方案保留小片段,PCR扩增11个循环;用Illumina Novaseq 6000测序仪(2×150bp),每个样本产出约4000万条读段。
结果解读:单链文库有效整合了ScfDNA的多种构象,测序数据覆盖片段组学、末端基序等多维度分析需求,避免了双链文库对单链/超短片段的遗漏。
实验所用关键产品:Claret Bioscience的SRSLYTM PicoPlus DNA NGS Library Preparation Base Kit(货号CBS-K250B-24)、Illumina Novaseq 6000测序仪。
3.4 多维度特征生物信息学分析
实验目的:解析ScfDNA的片段组学、末端基序、微生物组及功能元件特征。
方法细节:用SRSLYumi软件对测序数据去 multiplex,BBmerge合并配对读段,fastp修剪接头;用Bowtie2比对人类基因组(hg38)和Lambda噬菌体基因组,未比对读段用OneCodex比对微生物数据库;用samtools分析片段长度,HOMER识别功能元件(启动子、外显子),Prism8进行t检验和AUROC分析,ClustVis做PCA(主成分分析)和层次聚类。
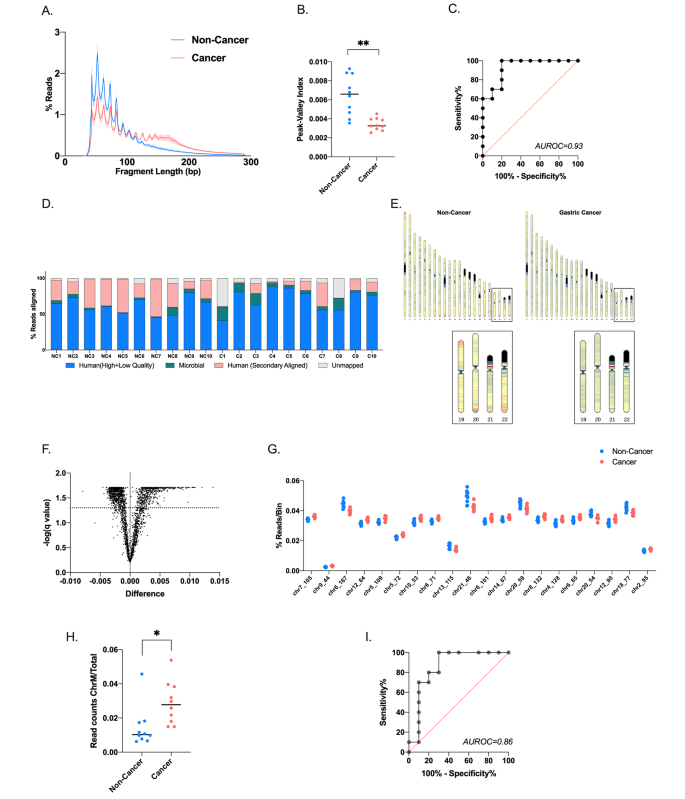
结果解读:胃癌与非癌样本的ScfDNA在片段长度(峰谷指数)、末端4-mer基序(87个差异基序)、微生物来源比例(胃癌组更高)、功能元件覆盖(非癌组启动子/外显子覆盖更高)上存在显著差异;多维度特征整合后,PCA的PC1轴可有效区分两组(p<0.0001)。
4. Biomarker研究及发现成果解析
4.1 Biomarker定位与筛选逻辑
本研究的生物标志物为ScfDNA的多维度特征组合,包括:(1)片段组学特征(峰谷指数、<100bp与>100bp片段比);(2)末端基序特征(87个差异4-mer基序);(3)微生物组特征(微生物来源比例、多样性);(4)功能元件特征(峰数量、基因组区域覆盖)。筛选逻辑为“描述基础特征→比较组间差异→整合特征验证区分能力”:先通过BRcfDNA-seq解析ScfDNA的构象和片段分布,再比较胃癌与非癌样本的差异特征,最后用多变量分析验证整合特征的区分效能。
4.2 研究过程与核心数据
Biomarker来源:唾液上清的ScfDNA。
验证方法与结果:
- 片段组学特征:胃癌样本的ScfDNA峰谷指数(反映片段分布的“锯齿状”程度)显著低于非癌组(n=10,p=0.0012),AUROC=0.93(区分胃癌与非癌);<100bp与>100bp片段比在非癌组更高(n=10,p=0.0059),AUROC=0.89。
- 末端基序特征:胃癌样本的末端4-mer基序随机性更低(Shannon熵更低,n=10,p=0.1894),共鉴定出87个差异基序(如G-rich基序在非癌组更丰富)。
- 微生物组特征:胃癌样本的ScfDNA微生物来源比例更高(n=10,p=0.0361),多样性更低(Shannon指数更低,n=10,p=0.1936)。
- 功能元件特征:非癌样本的ScfDNA在启动子、外显子区域的峰数量更多(n=10,p=0.0447),AUROC=0.67。
4.3 核心成果与创新性
功能关联:ScfDNA的多维度特征可有效区分胃癌与非癌样本,其中峰谷指数的区分能力最优(AUROC=0.93),多特征整合后的PCA-PC1轴区分效能达p<0.0001。
创新性:(1)首次系统解析ScfDNA的多维度特征(片段组学+末端基序+微生物组+功能元件),发现其“锯齿状”片段分布、差异末端基序及微生物组变化是胃癌的潜在生物标志物;(2)首次验证单链文库测序(BRcfDNA-seq)在ScfDNA分析中的优势,解决了传统方法对单链/超短片段的遗漏问题;(3)为唾液作为胃癌液体活检生物流体提供了cfDNA层面的证据,补充了现有蛋白/RNA标志物的不足。
本研究通过多维度特征分析,证实ScfDNA的非突变特征可作为胃癌液体活检的潜在生物标志物,为胃癌的非侵入性诊断提供了新方向。未来需在更大样本量中验证特征的稳定性,并探索其与临床病理参数(如肿瘤分期)的关联,进一步推动临床转化。
