1. 领域背景与文献引入
文献英文标题:Personalized circulating tumor DNA analysis for sensitive disease monitoring and detection of relapse in neuroblastoma;发表期刊:Biomarker Research;影响因子:未公开;研究领域:神经母细胞瘤液态活检与个性化 biomarker 研究。
神经母细胞瘤是儿童最常见的颅外实体瘤,起源于发育中的交感神经系统,预后差异极大——依赖肿瘤分期、遗传学特征(如MYCN扩增、ALK突变)及诊断年龄。常规疾病监测主要采用计算机断层扫描(CT)、磁共振成像(MRI)或123I-间碘苄基胍(MIBG)显像,但这些方法存在电离辐射、麻醉风险,且对低肿瘤负荷(如微小残留病)的检测敏感性有限。循环肿瘤DNA(ctDNA)作为液态活检的核心生物标志物,因非侵入性、实时反映肿瘤负荷的优势,近年来在神经母细胞瘤中显示出潜力。然而,既往研究多采用“通用ctDNA检测试剂盒”,仅靶向预定义的常见驱动变异(如MYCN扩增、ALK突变),导致两大局限性:①约50%患者无此类变异,会被排除在分析之外;②预定义变异数量有限,难以覆盖肿瘤异质性,敏感性不足。针对这一空白,本研究旨在评估患者特异性ctDNA分析的临床价值——通过设计个性化测序panel覆盖每个患者的肿瘤特异性变异,解决通用试剂盒的普适性与敏感性问题。
2. 文献综述解析
文献综述围绕“ctDNA检测策略的演变”展开核心评述,作者将现有研究分为两类:
- 通用ctDNA试剂盒:靶向预定义的常见驱动变异(如MYCN、ALK),虽能反映肿瘤负荷,但仅适用于携带特定变异的患者(约50%患者被排除),且预定义变异数量有限,难以捕捉肿瘤异质性,敏感性不足。
- 个性化ctDNA分析:针对患者肿瘤特异性变异,理论上能覆盖所有患者,但此前未在神经母细胞瘤中系统验证。
现有研究的关键结论是“通用ctDNA试剂盒可行,但局限性显著”,而本研究的创新价值在于首次在神经母细胞瘤中验证个性化ctDNA panel的临床价值:①每个患者设计10个肿瘤特异性单核苷酸变异(SNVs)的测序panel,覆盖编码与非编码区域,即使无MYCN/ALK异常也能纳入分析;②采用带独特分子标识符(UMI)的超深测序技术(SiMSen-Seq)消除PCR误差,显著提高检测敏感性。
3. 研究思路总结与详细解析
整体框架概括
本研究核心目标是评估个性化ctDNA分析在神经母细胞瘤“治疗监测(肿瘤负荷评估)”与“复发检测”中的有效性;核心科学问题是“个性化ctDNA是否比常规标志物更敏感、更适用于所有患者”;技术路线遵循“肿瘤变异筛选→个性化panel设计→纵向样本检测→临床关联分析”闭环:①通过肿瘤与白细胞全基因组测序筛选患者特异性SNVs;②设计10个SNVs/患者的个性化panel;③收集13例患者的136份纵向血浆样本,用SiMSen-Seq检测ctDNA;④关联ctDNA水平与临床参数(风险分组、治疗反应、复发)及常规标志物。
3.1 患者特异性SNV筛选与个性化panel设计
实验目的是筛选每个患者的肿瘤特异性变异,为ctDNA检测提供靶标。方法细节:纳入13例神经母细胞瘤儿童(n=13),对肿瘤组织与白细胞DNA进行全基因组测序,筛选“肿瘤中高变异等位基因频率(VAF)、白细胞中无检出”的SNVs(排除胚系变异),最终为每个患者选择10个SNVs(覆盖编码与非编码区域)构建个性化panel。结果解读:共纳入74个编码、55个非编码SNVs,其中6例患者(46%)无MYCN/ALK异常(若用通用试剂盒会被排除),说明个性化panel能覆盖更广泛患者。产品关联:文献未提及具体测序试剂盒,领域常规使用Illumina TruSeq DNA PCR-Free Kit等全基因组测序试剂。
3.2 血浆cfDNA提取与ctDNA检测(SiMSen-Seq技术)
实验目的是从血浆中提取游离DNA(cfDNA),并高敏感检测ctDNA。方法细节:收集13例患者的136份纵向血浆样本(n=136),用标准方法提取cfDNA;采用SiMSen-Seq技术——在每个cfDNA分子上添加独特分子标识符(UMI),PCR扩增后仅保留UMI家族大小≥3的分子,生成误差校正的“共识读长”(消除PCR诱导错误);通过共识读长中的肿瘤特异性SNVs计算ctDNA水平(单位:突变肿瘤分子数/毫升血浆,MTM/ml)。结果解读:测序中位深度为42180 reads(n=136),中位共识读长数21642;诊断时,cfDNA中SNVs的VAF与肿瘤组织DNA的VAF高度相关(编码与非编码SNVs的Spearman相关系数均P<0.0001),说明个性化panel能准确捕捉肿瘤变异。产品关联:实验采用SiMSen-Seq技术(文献引用方法学[12]),未明确试剂品牌,领域常规使用IDT for Illumina UMI Adapters等UMI条码试剂盒。
3.3 纵向ctDNA水平与临床参数的关联分析
实验目的是评估ctDNA与临床结局的相关性,并对比常规标志物性能。方法细节:①按高危/非高危分组分析诊断时ctDNA水平;②纵向监测治疗中ctDNA变化;③对比复发与无复发患者的ctDNA状态;④将ctDNA与5种常规标志物(血清NSE、嗜铬粒蛋白A,尿多巴胺、高香草酸(HVA)、香草扁桃酸(VMA))的敏感性比较。结果解读:①诊断时,高危患者的cfDNA/ctDNA水平显著高于非高危患者(P=0.009,Mann-Whitney检验);②有效治疗期间,ctDNA逐步下降(如高危患者诱导化疗后ctDNA明显降低);③无复发患者的23份随访样本中,ctDNA均为阴性(0假阳性);④4次复发均伴随ctDNA升高,其中1例患者的ctDNA在复发前78天转为阳性,水平升高1500倍(复发时所有SNVs均阳性);⑤ctDNA比常规标志物更敏感——诊断时ctDNA高于参考值(1 MTM/ml)的倍数(10^4-10^6倍)远高于常规标志物(仅数倍),复发时ctDNA更早升高(早于影像学78天)。
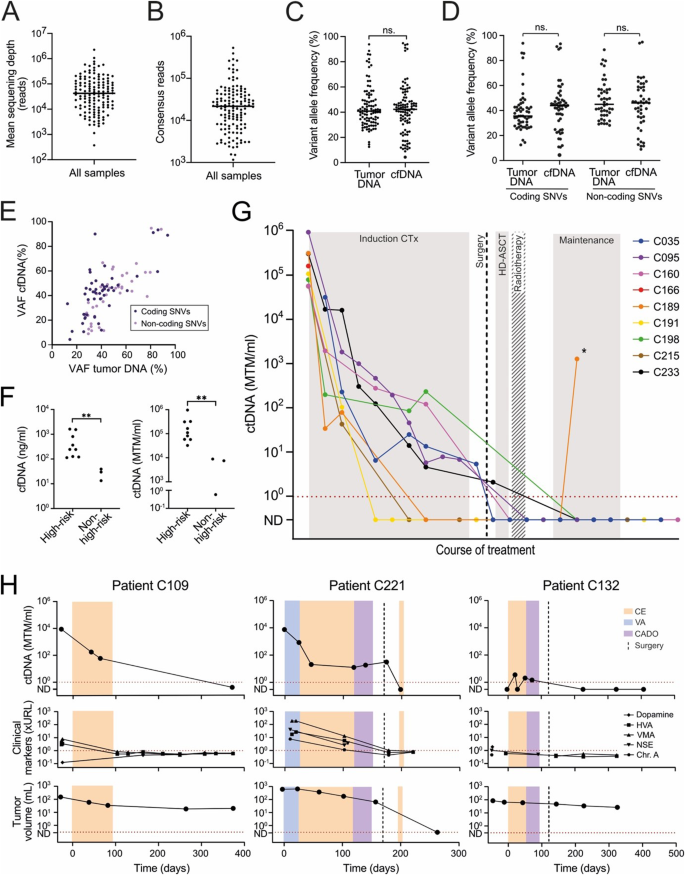
图1展示了个性化ctDNA与肿瘤DNA的相关性、高危与非高危患者的ctDNA差异,及治疗过程中ctDNA的动态变化。

图2对比了ctDNA与常规标志物在复发检测中的性能,显示ctDNA更早预警复发。
4. Biomarker 研究及发现成果解析
Biomarker 定位
本研究的核心Biomarker是患者特异性ctDNA(每个患者的10个肿瘤特异性SNVs组合)。筛选逻辑:通过肿瘤与白细胞全基因组测序,筛选高VAF的肿瘤特异性SNVs(排除胚系变异);验证逻辑:纵向血浆样本的SiMSen-Seq检测,关联临床结局(治疗反应、复发)。
研究过程详述
Biomarker来源是“患者肿瘤组织的特异性SNVs”(通过全基因组测序确定);验证方法采用SiMSen-Seq超深测序——添加UMI消除PCR误差,要求共识读长的UMI家族大小≥3,确保检测特异性;特异性数据:无复发患者的23份随访样本中,ctDNA均为阴性(0假阳性);敏感性数据:4次复发均检测到ctDNA升高(100%敏感性),其中1例患者的ctDNA在复发前78天即转为阳性,水平升高1500倍(复发时所有SNVs均阳性)。
核心成果提炼
①功能关联:诊断时ctDNA水平与风险分组正相关(高危患者更高,P=0.009);治疗期间ctDNA逐步下降,反映治疗有效性;复发时ctDNA显著升高,能提前78天预警复发(早于常规影像学)。
②创新性:首次证明个性化ctDNA分析适用于所有神经母细胞瘤患者(包括46%无MYCN/ALK异常的患者),解决了通用试剂盒的局限性;同时,ctDNA比常规标志物更敏感——诊断时ctDNA高于参考值的倍数远高于常规标志物,复发时ctDNA更早升高。
③统计学结果:诊断时ctDNA VAF与肿瘤DNA VAF显著相关(P<0.0001,Spearman相关);高危与非高危患者的cfDNA/ctDNA水平差异有统计学意义(P=0.009);4次复发的ctDNA均阳性(100%敏感性,n=4)。
综上,本研究证明个性化ctDNA分析是神经母细胞瘤的敏感监测工具,能覆盖所有患者,提前预警复发,为临床个性化管理提供了新的生物标志物策略。
