1. 领域背景与文献引入
文献英文标题:A robust prognostic gene expression signature for early stage lung adenocarcinoma;发表期刊:Biomark Res;影响因子:未公开;研究领域:早期肺腺癌预后基因表达生物标志物。
肺癌是全球发病率第三、死亡率第一的癌症类型,非小细胞肺癌(NSCLC)的5年生存率不足15%。早期(I/II期)肺腺癌患者的临床决策核心是是否在手术切除后进行辅助化疗——目前IA期患者不推荐辅助化疗(1A类证据),IB期肿瘤>4cm患者可考虑(2B类证据),但IA期患者5年生存率仅约73%,IB期约58%,仍有大量患者存在复发风险。因此,亟需可靠的预后生物标志物筛选出早期高风险患者,指导辅助治疗决策。
此前,多项研究尝试通过基因表达谱寻找肺腺癌预后生物标志物,但结果未达成共识——部分研究混合了接受辅助治疗的患者,或未校正基因芯片数据的技术偏差(如RNA降解、mRNA起始量差异),导致结果可靠性不足。针对这一空白,本研究聚焦未接受辅助治疗的I/II期肺腺癌患者,分析多队列基因表达数据,旨在筛选一致的预后基因并构建稳健的预后签名。
2. 文献综述解析
现有研究多采用“基因表达量化-差异基因筛选-验证”的策略寻找肺腺癌预后生物标志物,但存在三大局限性:① 患者选择异质性:部分研究纳入了接受辅助化疗的患者,治疗干预会混淆预后信号;② 技术偏差未校正:早期基因芯片数据常因RNA降解、样本间mRNA起始量差异引入偏差,影响结果重复性;③ 队列规模小:单队列研究的统计效能不足,难以发现一致的预后基因。
本研究针对上述问题进行创新设计:① 严格纳入未接受辅助治疗的I/II期肺腺癌患者,避免治疗干扰;② 对基因芯片数据进行偏差校正(使用bias包校正RNA降解和mRNA多样性偏差),提高数据可靠性;③ 整合七个独立队列(覆盖GEO数据库及TCGA数据),通过多队列验证确保基因的一致性。
3. 研究思路总结与详细解析
3.1 队列选择与数据收集
实验目的:获取符合“未接受辅助治疗、I/II期肺腺癌、Affymetrix芯片平台”的基因表达及临床数据。
方法细节:从GEO数据库下载6个队列(GSE8894、GSE14814、GSE30219、GSE31210、GSE37745、GSE50081),从caarraydb.nci.nih.gov下载Shedden等的队列;筛选标准:① 组织学确诊肺腺癌;② 病理分期I/II期;③ 未接受新辅助/辅助/靶向治疗;④ 使用Affymetrix HG-U133A或HG-U133 Plus 2.0芯片。
结果解读:最终纳入7个队列(共约500例患者),临床数据包括总生存期(OS)或无复发生存期(RFS)。
3.2 数据归一化与偏差校正
实验目的:消除基因芯片数据的技术偏差,提高可比性。
方法细节:① 除GSE9984(未获得原始数据,使用GEO提供的GCRMA归一化数据)外,其余队列用RMA算法归一化;② 使用bias包(v0.0.5)校正两种技术偏差:RNA降解(5’与3’探针表达差异)、mRNA起始量差异(RMA IQR偏差)。
结果解读:偏差校正后的数据更符合生物学预期,减少了技术因素对后续分析的干扰。
3.3 预后基因筛选
实验目的:筛选在多队列中一致显著的预后基因。
方法细节:对每个基因(共22277个共同探针)进行:① 按表达量中位数分为高/低表达组;② 单因素Cox回归分析总生存期(OS);③ 筛选标准:所有队列中风险比(HR)方向一致(高表达均与不良预后相关),且至少6个队列P<0.05。
结果解读:最终筛选出7个基因(代表7个独特探针),包括ADAM10、ASPM、DLGAP5、FGFR10P、KIF15、NCAPG、RAD51AP1。
3.4 ESLA-7签名构建与初步验证
实验目的:基于筛选的基因构建预后签名,并评估其在训练队列中的性能。
方法细节:将患者的ESLA-7得分定义为7个基因log₂表达量的平均值;按得分中位数将每个队列分为高风险组(>中位数)和低风险组(≤中位数),进行Kaplan-Meier生存分析及Cox回归。
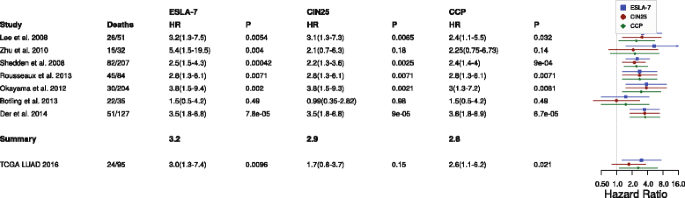
结果解读:ESLA-7签名在7个队列中的加权平均HR=3.2,且6个队列中差异有统计学意义(P<0.05);多变量分析(校正年龄、分期、性别)显示,加权平均HR=2.7,仍显著。

3.5 独立队列验证(TCGA数据集)
实验目的:在独立队列中验证ESLA-7的预后性能,尤其关注未接受化疗的患者。
方法细节:① 下载TCGA LUAD队列的RNA-seq数据(RSEM归一化)及临床信息;② 分析I/II期患者的无复发生存期(RFS),并根据治疗信息 censoring 接受化疗患者的生存时间,得到未接受化疗的亚组(n=95);③ 计算ESLA-7得分并进行Cox回归。
结果解读:① 全I/II期队列(n=~200)的HR=1.8(95%CI 1.3-2.6,P<0.001);② 未接受化疗亚组的HR=3.0(95%CI 1.3-7.4,P<0.01),显示ESLA-7在未治疗患者中具有更强的预后价值。
3.6 与现有预后签名的比较
实验目的:对比ESLA-7与已知签名(CIN25、CCP)的预后性能。
方法细节:将CIN25(染色体不稳定性签名)、CCP(细胞周期进展签名)以相同方法应用于7个训练队列,计算加权平均HR。
结果解读:ESLA-7的加权平均HR=3.2,优于CIN25(HR=2.9)和CCP(HR=2.8),提示其捕获了更丰富的预后信息。
4. Biomarker 研究及发现成果解析
Biomarker 定位与筛选逻辑
本研究的核心生物标志物是ESLA-7基因表达签名(由7个基因的平均表达量构成),筛选逻辑遵循“多队列一致验证”原则:① 从7个训练队列中筛选出“HR方向一致+6个队列显著”的基因;② 通过独立TCGA队列验证,尤其聚焦未接受化疗的早期患者,确保签名的特异性。
验证过程与性能
- 来源:训练队列来自GEO数据库的7个肺腺癌队列(未接受辅助治疗的I/II期患者),验证队列来自TCGA LUAD的RNA-seq数据。
- 验证方法:无复发生存期(RFS)分析(Kaplan-Meier曲线、Cox回归);对接受化疗的患者进行生存时间 censoring,构建未治疗亚组。
- 性能数据:① 训练队列加权平均HR=3.2;② 未接受化疗的I/II期患者亚组(n=95)HR=3.0(95%CI 1.3-7.4,P<0.01);③ 优于已有的CIN25(HR=2.9)和CCP(HR=2.8)签名。
核心成果与功能关联
- 预后价值:ESLA-7签名可有效区分早期肺腺癌高风险患者,未接受化疗的高风险组患者RFS显著缩短(HR=3.0)。
- 功能关联:7个基因均与染色体不稳定性(CIN)相关——如ASPM、KIF15参与动粒组装,NCAPG属于凝缩蛋白复合物,这些基因的高表达提示染色体分离异常,进而导致肿瘤进展。
- 创新性:① 首次在未接受辅助治疗的早期患者中筛选出一致的预后基因;② 签名性能优于经典的CIN25和CCP签名,可能因纳入了ADAM10、FGFR10P等与细胞增殖相关的基因,补充了染色体不稳定性之外的预后信息。
结论:ESLA-7签名若经前瞻性验证,有望成为早期肺腺癌患者的预后工具,指导高风险患者的辅助治疗决策,减少过度治疗。
