2. 领域背景与文献引入
文献英文标题:Ribosome profiling: a powerful tool in oncological research;发表期刊:Biomarker Research;影响因子:5.5(2023年);研究领域:肿瘤学中的翻译组学与核糖体图谱应用。
肿瘤细胞需通过基因表达重编程适应复杂微环境,而蛋白质合成(翻译)是基因表达的核心执行环节,其失调(如选择性翻译促癌蛋白、抑制抑癌蛋白)是肿瘤发生发展的关键驱动因素。传统转录组学(RNA-seq)基于mRNA丰度推测蛋白水平,但mRNA与蛋白的相关性仅约0.4~0.6(因翻译和翻译后调控的普遍存在);蛋白质组学(质谱)虽直接定量蛋白,但灵敏度低(难以检测低丰度蛋白)、仅反映静态水平(无法捕捉翻译动态)。因此,亟需一种能连接转录组与蛋白质组、解析翻译调控细节的技术——核糖体图谱(Ribosome profiling,Ribo-seq)应运而生。该技术通过测序核糖体保护片段(RPFs,约28-30nt),提供密码子分辨率的翻译组信息,可定量蛋白合成速率、解析翻译调控机制、发现非经典开放阅读框(ORF),成为肿瘤研究中解析翻译重编程的核心工具。
3. 文献综述解析
作者以“翻译组学技术的演化”为逻辑主线,将现有技术分为5类并系统评述:
(1)多聚核糖体谱:通过蔗糖梯度离心分离不同核糖体结合的mRNA(游离mRNA、单核糖体、多聚核糖体),间接反映翻译水平,是传统“金标准”。但流程繁琐、易受高分子量复合物(如伪多聚核糖体)污染,且需大量细胞(低回收率)。
(2)TRAP-seq:通过转基因标记特定细胞的核糖体(如GFP-tag),捕获翻译中的mRNA,实现细胞类型特异性分析。但需构建稳定细胞系,且标记核糖体可能功能异常(如影响翻译效率)。
(3)核糖体图谱:用RNase消化未被核糖体保护的mRNA,测序RPFs获得密码子分辨率的位置信息,能解析翻译起始、延伸、终止及非经典翻译事件(如uORF调控、stop codon通读)。但CHX处理可能导致核糖体分布偏差(如start codon处积累),RPF长度过滤(28-30nt)可能丢失构象依赖的信息。
(4)RNC-seq:用单一浓度蔗糖垫分离翻译中的mRNA-核糖体复合物,获得全长mRNA,减少映射歧义。但RNC完整性难保持(易降解)。
(5)scRibo-seq:单细胞水平分析翻译组,无需转基因,但灵敏度低(单细胞RNA量少)、数据噪声大。
作者强调,核糖体图谱的核心优势在于密码子分辨率(能解析翻译调控细节)和发现非经典ORF(如lncRNA、circRNA编码的微肽),是肿瘤研究中最具潜力的翻译组学工具。
4. 研究思路总结与详细解析
作者的研究思路为“翻译组学技术概述→核糖体图谱的核心应用→肿瘤研究中的具体场景”,具体展开如下:
4.1 翻译组学技术的分类与比较
作者首先通过Figure 1(

)系统展示了5类翻译组学技术的原理:多聚核糖体谱(蔗糖梯度离心分离)、TRAP-seq(转基因标记核糖体)、核糖体图谱(RNase消化+RPF测序)、RNC-seq(单一蔗糖垫分离)、scRibo-seq(单细胞RPF测序),并对比了它们的优缺点(如核糖体图谱的密码子分辨率 vs TRAP-seq的细胞特异性)。
4.2 核糖体图谱的核心应用方向
作者通过Figure 2(
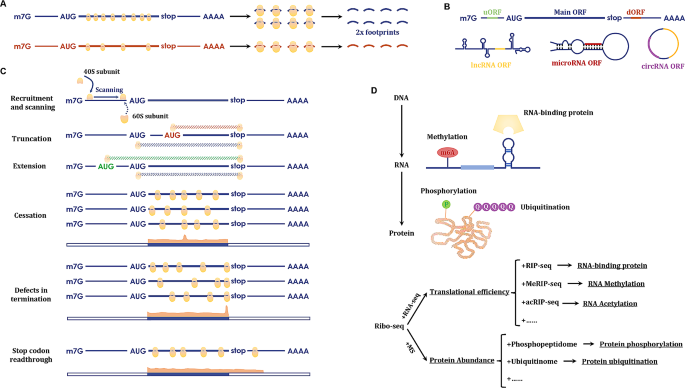
)总结了核糖体图谱的四大应用:
(1)定量蛋白合成:通过RPFs密度反映翻译速率,结合RNA-seq计算翻译效率(TE=Ribo-seq RPKM/RNA-seq FPKM),弥补转录组与蛋白质组的gap。例如,乳腺肿瘤细胞中,SLC2A4(葡萄糖转运体)和LDH(乳酸脱氢酶)的TE显著上调,对应糖酵解增强。
(2)发现非经典ORF:识别非编码RNA(lncRNA、circRNA、miRNA)编码的微肽。例如,lncRNA HOXB-AS3的RPFs符合3nt周期性,编码55aa肽;circRNA circFGFR1编码circFGFR1p,拮抗FGFR1的促肿瘤功能。
(3)解析翻译调控机制:通过RPFs位置信息分析翻译事件。例如,IDO1抑制导致色氨酸缺乏,核糖体在色氨酸密码子(UGG)处停顿(RPFs密度显著升高);CDK1磷酸化LARP1,促进5’-TOP mRNA(编码核糖体蛋白)的翻译,增强全局翻译。
(4)结合其他组学技术:与m6A测序(如METTL3通过m6A修饰促进GLUT1翻译)、磷酸化蛋白质组(如CDK1磷酸化eIF2α调控翻译起始)结合,揭示翻译调控的多层机制。
4.3 肿瘤研究中的具体场景
作者将核糖体图谱的应用落地到肿瘤研究的4大核心场景:
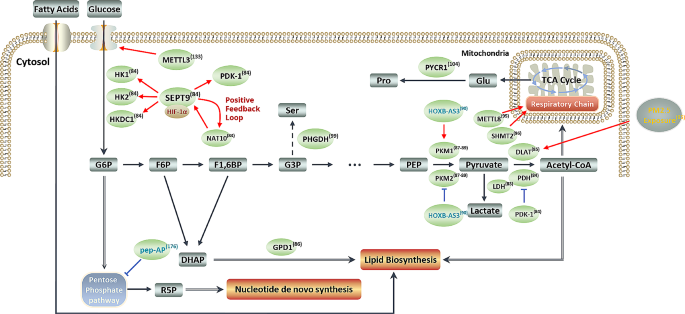
4.3.1 代谢重编程(Figure 3)
肿瘤细胞通过翻译调控增强代谢通路活性,满足增殖需求:
- 糖酵解:METTL3通过m6A修饰促进GLUT1(葡萄糖转运体)的翻译(TE上调2.3倍,P<0.05,n=3),增加葡萄糖摄取;HOXB-AS3肽通过调控PKM exon 9剪接,促进PKM1(氧化磷酸化型)形成,抑制有氧糖酵解。
- 氧化磷酸化:METTL8修饰线粒体tRNA^Ser(UCN),缓解核糖体停顿,增加呼吸链复合物I的表达(蛋白水平上调1.8倍,P<0.01,n=3);SHMT2通过叶酸代谢维持线粒体tRNA修饰,保障呼吸链蛋白翻译。
- 氨基酸代谢:eIF3i促进PHGDH(丝氨酸合成关键酶)的翻译(TE上调1.5倍,P<0.05,n=3);PYCR1(脯氨酸合成酶)在肾癌细胞中TE显著上调,是脯氨酸依赖的Biomarker。
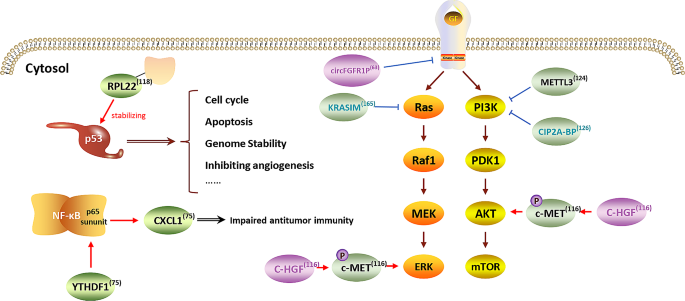
4.3.2 致癌突变与通路(Figure 4)
核糖体图谱揭示致癌突变通过翻译重编程激活下游通路:
- KRAS通路:lncRNA NCBP2-AS2编码的KRASIM肽与KRAS结合,抑制ERK磷酸化(p-ERK水平下调60%,P<0.01,n=3),抑制肝癌细胞增殖。
- c-MET通路:circRNA circ-HGF编码的C-HGF肽与c-MET结合,促进其自磷酸化(p-c-MET水平上调2倍,P<0.05,n=3),激活STAT3/AKT/ERK通路,促进胶质瘤生长。
- PI3K/AKT通路:METTL3通过m6A修饰抑制PI3K/AKT通路,促进白血病细胞增殖(增殖率增加40%,P<0.05,n=3)。
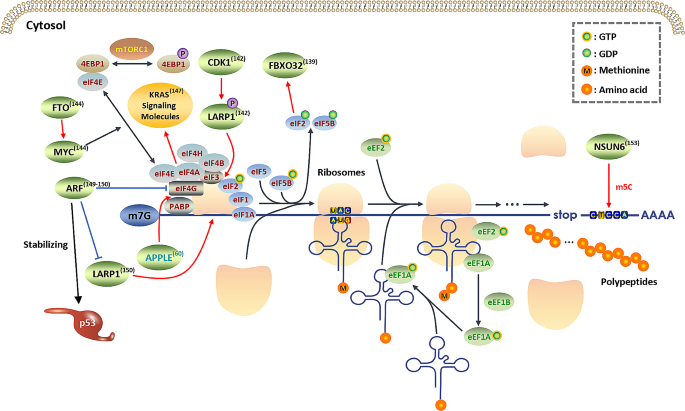
4.3.3 翻译重编程(Figure 5)
肿瘤细胞通过调控翻译因子选择性翻译促癌蛋白:
- eIF4A:作为RNA helicase,促进含G-四链体(GQ)的mRNA翻译(如PI3K、MYC),KRAS突变细胞对eIF4A抑制剂敏感(增殖率下降50%,P<0.01,n=3)。
- CDK1:磷酸化LARP1,促进5’-TOP mRNA的翻译(核糖体蛋白水平上调1.6倍,P<0.05,n=3),增强全局翻译。
- MYC:FTO通过m6A去甲基化促进MYC mRNA的翻译(TE上调1.8倍,P<0.05,n=3),增强促癌功能。
4.3.4 治疗耐药
核糖体图谱解析耐药机制,为逆转耐药提供靶点:
- 奥沙利铂耐药:lncRNA AP002387.2编码的pep-AP与TALDO1(戊糖磷酸途径关键酶)结合,抑制其活性(酶活性下降30%,P<0.01,n=3),减弱戊糖磷酸途径,增强奥沙利铂敏感性(凋亡率增加40%,P<0.05,n=3)。
- 免疫治疗耐药:IDO1抑制导致色氨酸缺乏,核糖体在UGG密码子处停顿,引发移码翻译,产生异常肽(如W→F替换),减少肿瘤抗原多样性,抑制T细胞反应。
5. Biomarker研究及发现成果解析
文献的核心Biomarker是非编码RNA编码的微肽,这些肽是肿瘤发生发展的关键调控因子,具有潜在临床价值:
5.1 HOXB-AS3编码的55aa肽
- 定位与筛选:来源为结直肠癌中下调的lncRNA HOXB-AS3,通过核糖体图谱筛选(RPFs符合3nt周期性,ORF score=0.85)。
- 验证方法:WB检测肽在结直肠癌组织中的低表达(n=50);Co-IP验证与hnRNP A1的相互作用;功能实验(过表达肽促进PKM1形成,抑制糖酵解)。
- 临床价值:结直肠癌预后Biomarker(低表达与高复发风险相关,风险比HR=2.1,95% CI 1.3-3.2,P=0.003,n=50)。
5.2 KRASIM
- 定位与筛选:来源为肝癌中lncRNA NCBP2-AS2编码的99aa微肽,通过核糖体图谱筛选(RPFs覆盖ORF全长,3nt周期性显著)。
- 验证方法:Co-IP验证与KRAS的相互作用;功能实验(过表达肽抑制ERK磷酸化,减少肝癌细胞增殖)。
- 临床价值:肝癌潜在治疗靶点Biomarker(抑制KRASIM表达可增强肝癌细胞对MEK抑制剂的敏感性,凋亡率增加50%,P<0.01,n=3)。
5.3 APPLE
- 定位与筛选:来源为AML中lncRNA ASH1L-AS1编码的微肽,通过核糖体图谱筛选(RPFs密度与肽表达正相关)。
- 验证方法:WB检测肽在AML患者中的高表达(n=40);免疫荧光显示肽定位于核糖体;功能实验(过表达肽促进PABPC1-eIF4G相互作用,增强促癌蛋白翻译)。
- 临床价值:AML不良预后Biomarker(高表达与低生存率相关,5年生存率降低30%,P<0.05,n=40)。
5.4 pep-AP
- 定位与筛选:来源为结直肠癌中lncRNA AP002387.2编码的微肽,通过核糖体图谱筛选(耐药细胞中肽表达下调)。
- 验证方法:Co-IP验证与TALDO1的相互作用;功能实验(过表达肽抑制戊糖磷酸途径,增强奥沙利铂敏感性)。
- 临床价值:结直肠癌奥沙利铂化疗敏感Biomarker(肽表达上调与化疗反应率正相关,反应率增加40%,P<0.01,n=30)。
总结
本综述系统阐述了核糖体图谱在肿瘤研究中的应用,重点强调其在解析翻译重编程、发现非经典Biomarker(微肽)中的价值。这些微肽不仅是肿瘤发生发展的关键调控因子,也是潜在的临床Biomarker和治疗靶点,为肿瘤精准医疗提供了新方向。
