1. 领域背景与文献引入
文献英文标题:Multiple ctDNA- based biomarkers predict benefit from selective RET Inhibition in non-small cell lung cancer patients: exploratory analysis of a prospective study;发表期刊:Biomark Res;影响因子:未公开;研究领域:非小细胞肺癌(NSCLC)靶向治疗生物标志物研究。
非小细胞肺癌(NSCLC)是全球发病率和死亡率最高的恶性肿瘤之一,其中1-2%的晚期患者携带RET(Rearranged during transfection)融合突变。普拉替尼等选择性RET抑制剂的问世,使RET融合阳性NSCLC患者的治疗进入靶向时代,成为该亚型患者的标准治疗方案。然而,临床实践中仍面临两大核心挑战:一是早期识别对RET抑制剂敏感的患者,二是及时监测耐药性及明确耐药机制——目前RET抑制剂的耐药机制尚未完全阐明。
液体活检技术,尤其是循环肿瘤DNA(ctDNA)分析,因具有非侵入性、可重复采样、实时反映肿瘤基因组动态的优势,已成为NSCLC靶向治疗监测的重要手段。此前研究虽证实ctDNA可监测RET变异及治疗后的ctDNA清除,但多数研究随访时间较短(缺乏长期纵向数据),且仅采用单一ctDNA指标(如等位频率),难以全面预测治疗反应和疾病进展模式。因此,亟需通过前瞻性研究、整合多维度ctDNA指标,深入解析其对RET融合NSCLC患者治疗反应及进展的预测价值——这也是本研究的核心初衷,旨在为RET融合NSCLC患者的精准治疗监测提供更全面的生物标志物策略。
2. 文献综述解析
作者的综述评述逻辑围绕“RET融合NSCLC治疗现状→临床挑战→液体活检的潜力→现有研究局限”展开:首先概述RET融合NSCLC的治疗进展(普拉替尼是标准治疗,但耐药性不可避免);接着指出临床痛点——早期识别应答者和耐药性困难;随后引入液体活检(ctDNA)的优势,强调其作为非侵入性监测手段的潜力;最后评述现有研究的局限:现有研究虽证实ctDNA可监测RET变异及清除,但存在随访时间短、指标单一等不足,难以全面关联治疗反应和进展模式。
现有研究的关键结论可概括为两点:一是RET抑制剂对RET融合NSCLC患者有效,但耐药性不可避免;二是ctDNA可作为非侵入性手段监测RET变异及治疗后的ctDNA清除。技术方法的优势在于液体活检的非侵入性和可重复性,但局限性在于现有研究多为小样本、短随访,且仅采用单一ctDNA指标,无法全面反映肿瘤负荷和恶性程度。
本研究的创新价值在于:(1)采用前瞻性队列(21例患者),提供了更长时间的随访数据(中位随访17.5个月);(2)整合三种ctDNA指标(等位频率、cfDNA定量归一化、甲基化),从不同维度评估肿瘤负荷(突变频率、肿瘤分子数量、甲基化恶性程度);(3)详细分析了ctDNA的纵向动态模式与疾病进展模式的关联,填补了现有研究在进展模式分析上的空白。
3. 研究思路总结与详细解析
整体框架概括:研究目标是评估三种ctDNA指标(maxAF、MTM/mL、MD ratio)对RET融合NSCLC患者普拉替尼治疗反应及进展的预测价值;核心科学问题是多维度ctDNA指标能否有效预测治疗反应(如无进展生存期PFS、客观缓解率ORR)及疾病进展模式;技术路线为:前瞻性纳入21例RET融合NSCLC患者→采集基线、第8周、进展时的外周血→提取cfDNA并深度测序(20000×)→计算三种ctDNA指标→关联临床结局(PFS、ORR)→分析ctDNA动态模式与进展的关系→解析耐药突变机制。
3.1 患者入组与样本采集
实验目的:建立RET融合NSCLC患者的前瞻性队列,获取纵向时间点的外周血样本,为后续ctDNA分析提供基础。
方法细节:纳入21例经病理证实的RET融合阳性晚期NSCLC患者(均接受普拉替尼治疗),分别在基线(治疗前)、治疗第8周(首次影像学评估时)及疾病进展(PD)时采集外周血样本;血液采集后2小时内分离血浆,置于-80℃冰箱保存。
结果解读:成功纳入所有患者并获取完整纵向样本(无样本丢失或质量问题),为后续分析提供了稳定的样本基础。
产品关联:文献未提及具体外周血分离试剂,领域常规使用EDTA抗凝管采集血液,离心机分离血浆,超低温冰箱(-80℃)保存。
3.2 cfDNA提取与测序
实验目的:从血浆中提取高质量cfDNA,并通过深度测序获取基因组数据。
方法细节:采用QIAamp Circulating Nucleic Acid kit提取cfDNA,用Qubit荧光定量仪定量;随后进行深度测序(测序深度20000×),以确保检测低频率突变的敏感性。
结果解读:提取的cfDNA质量达标(浓度满足测序要求),测序数据覆盖度和均匀度良好,为后续指标计算和突变分析奠定基础。
产品关联:实验所用关键产品:QIAGEN的QIAamp Circulating Nucleic Acid kit(cfDNA提取)、Thermo Fisher Scientific的Qubit荧光定量仪(cfDNA定量)。
3.3 ctDNA指标计算
实验目的:基于测序数据,计算三种ctDNA指标,全面评估肿瘤负荷及恶性程度。
方法细节:计算三种指标:(1)maxAF(最大等位频率):肿瘤特异性突变的最高等位频率(反映突变频率);(2)MTM/mL(cfDNA定量归一化的平均肿瘤分子数):基于cfDNA总量归一化后的肿瘤分子数(反映肿瘤细胞数量);(3)MD ratio(甲基化恶性密度比):通过亚硫酸氢盐测序获取甲基化数据,结合机器学习模型计算的恶性肿瘤甲基化signature密度比(反映肿瘤恶性程度)。
结果解读:成功计算出所有患者各时间点的三种指标,指标间具有一定相关性(如maxAF与MTM/mL正相关),但也能从不同维度(突变、数量、甲基化)互补反映肿瘤状态。
产品关联:亚硫酸氢盐测序所用试剂,文献未提及具体品牌,领域常规使用Zymo Research的EZ DNA Methylation-Gold Kit(亚硫酸氢盐转化)。
3.4 ctDNA指标与临床结局关联分析
实验目的:分析三种ctDNA指标与临床结局(PFS、ORR)的关联,评估其预测价值。
方法细节:用Kaplan-Meier法分析ctDNA指标与PFS的关系(计算风险比HR及95%置信区间CI);用卡方检验分析第8周ctDNA清除状态与ORR的关联(ctDNA清除定义为maxAF<0.1%且MTM/mL检测不到肿瘤特异性变异)。
结果解读:(1)基线PIK3CA共突变患者的PFS显著短于无共突变患者(3.0个月vs 12.4个月,P<0.001);(2)基线低maxAF(HR=0.24,95%CI 0.07-0.80,P=0.012)、低MTM/mL(HR=0.20,95%CI 0.05-0.71,P=0.006)、低MD ratio(HR=0.09,95%CI 0.01-0.85,P=0.010)的患者,PFS显著更长;(3)第8周ctDNA清除的患者,PFS未达到(vs未清除者的4.8个月,P=0.002),且ORR更高(71.4%vs 0%)。
图片插入:
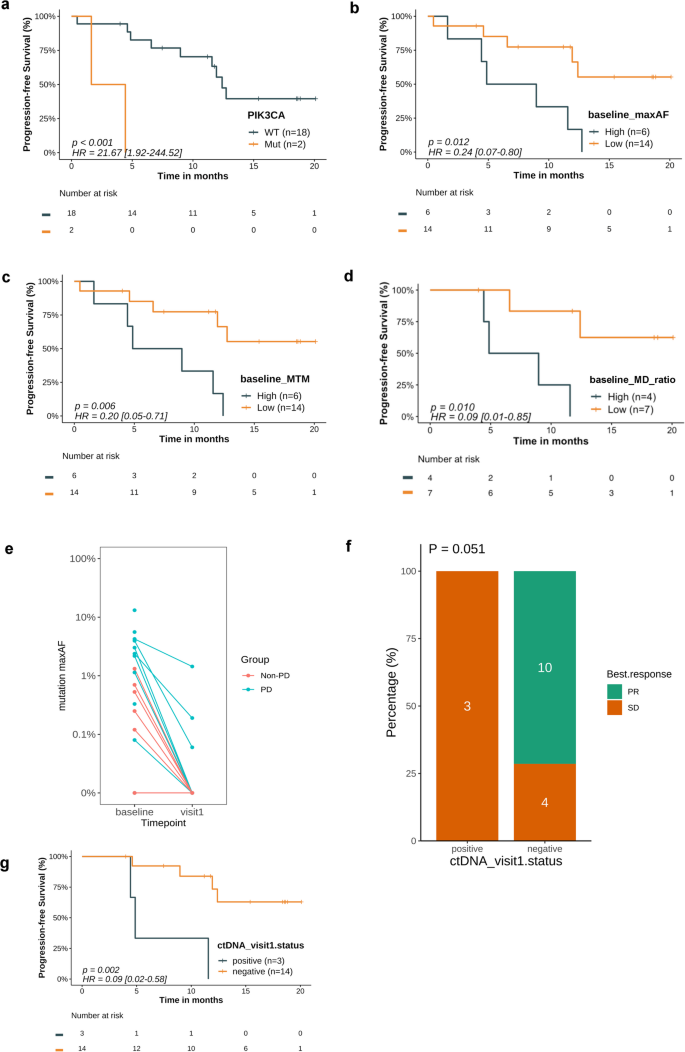
(图1展示了基线ctDNA指标与PFS的关联,如基线低maxAF患者的PFS曲线更平缓)。
3.5 ctDNA动态模式与进展分析
实验目的:解析ctDNA纵向动态模式与疾病进展模式的关联,评估分子进展与影像进展的时间差。
方法细节:根据第8周和进展时的ctDNA水平,将患者分为三种动态模式:(1)清除-反弹(第8周ctDNA清除,进展时重新出现且升高);(2)减少-反弹(第8周ctDNA减少但仍可检测,进展时升高);(3)持续清除(第8周清除,进展时仍未检测到)。分子进展定义为:获得性耐药突变、原有突变等位频率升高>10%或ctDNA清除后重新出现。
结果解读:三种模式对应不同进展模式:清除-反弹患者表现为系统性进展,减少-反弹患者出现新胸外病灶,持续清除患者出现脑转移或小肺结节(可能因ctDNA无法穿透血脑屏障或病灶小未释放ctDNA);分子进展较影像进展提前2.2个月(平均),提示ctDNA可早期预警进展。
图片插入:
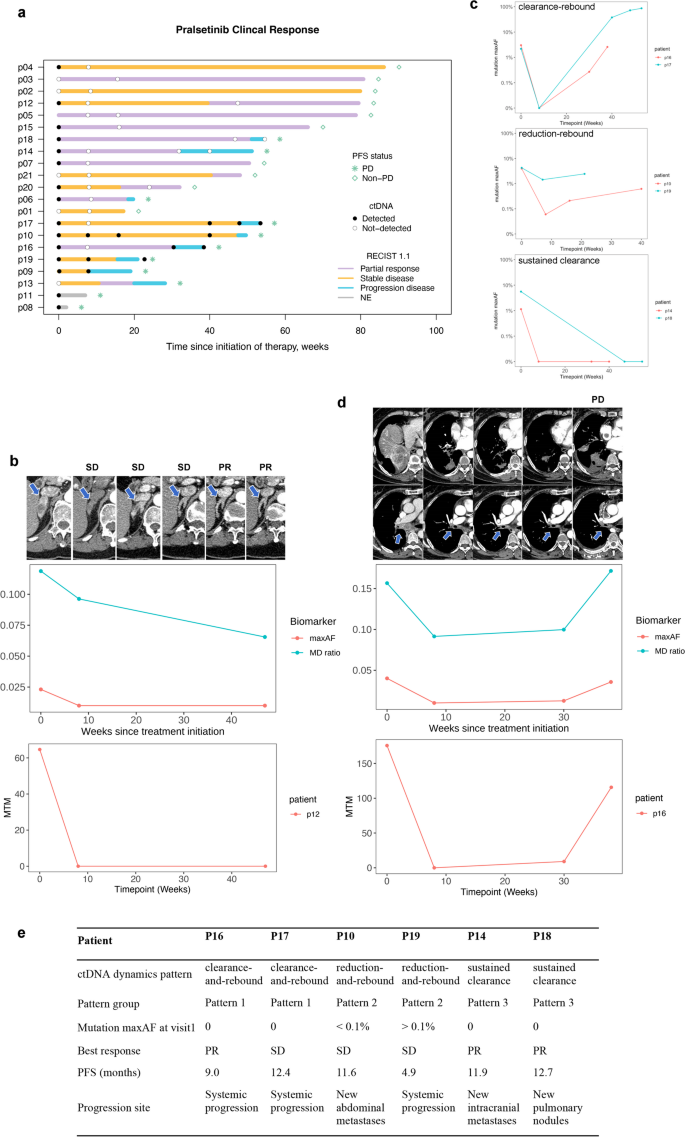
(图2展示了ctDNA动态模式与进展的关联,如清除-反弹患者的ctDNA水平在进展时显著升高)。
3.6 耐药突变分析
实验目的:解析RET融合NSCLC患者对普拉替尼耐药的分子机制。
方法细节:对进展时的ctDNA样本进行测序,分析获得性突变(基线未检测到的突变)。
结果解读:耐药样本中检测到获得性KRAS G12R突变(未发现RET靶点的获得性突变,如RET激酶域突变);此外,还检测到CSMD3扩增、B2M错义突变、EPHA7 E239K突变等非经典耐药突变——这些突变可能通过激活旁路通路(如RAS-MAPK通路)导致耐药。
4. Biomarker研究及发现成果解析
Biomarker定位:本研究涉及的Biomarker包括四类:(1)基线ctDNA指标(maxAF、MTM/mL、MD ratio);(2)早期(第8周)ctDNA清除状态;(3)ctDNA动态模式;(4)基线PIK3CA共突变。筛选与验证逻辑为:通过前瞻性队列采集纵向样本→测序计算Biomarker→关联临床结局(PFS、ORR、进展模式)→验证其预测价值。
研究过程详述
Biomarker的来源为患者外周血浆中的cfDNA;验证方法包括:(1)基线ctDNA指标:通过测序计算maxAF、MTM/mL、MD ratio,用Kaplan-Meier法关联PFS;(2)早期ctDNA清除:通过第8周测序数据判断清除状态,用Kaplan-Meier法关联PFS,卡方检验关联ORR;(3)ctDNA动态模式:通过纵向测序数据划分模式,关联进展模式;(4)基线PIK3CA共突变:通过基线测序检测,用Kaplan-Meier法关联PFS。
特异性与敏感性数据:(1)早期ctDNA清除的特异性:清除患者的ORR为71.4%,显著高于未清除者(0%);(2)基线低maxAF的敏感性:低maxAF患者的PFS风险降低76%(HR=0.24,P=0.012);(3)基线PIK3CA共突变的特异性:共突变患者的PFS显著缩短(3.0个月vs 12.4个月,P<0.001)。
核心成果提炼
- 基线Biomarker:基线PIK3CA共突变是不良预后Biomarker,这类患者的PFS显著缩短(3.0个月vs 12.4个月,P<0.001);基线低ctDNA水平(maxAF、MTM/mL、MD ratio)是良好预后Biomarker,低maxAF患者的PFS风险降低76%(HR=0.24,95%CI 0.07-0.80,P=0.012),低MTM/mL患者降低80%(HR=0.20,95%CI 0.05-0.71,P=0.006),低MD ratio患者降低91%(HR=0.09,95%CI 0.01-0.85,P=0.010)。
- 早期Biomarker:第8周ctDNA清除是良好预后Biomarker,清除患者的PFS未达到(vs未清除者的4.8个月,P=0.002),且ORR显著更高(71.4%vs 0%)。
- 动态Biomarker:ctDNA动态模式可预测进展模式——清除-反弹对应系统性进展,减少-反弹对应新胸外病灶,持续清除对应脑转移或小肺结节;分子进展较影像进展提前2.2个月,提示ctDNA可早期预警进展。
- 耐药Biomarker:获得性KRAS G12R突变是普拉替尼的耐药Biomarker,未发现RET靶点的获得性突变——这为后续联合治疗(如RET抑制剂+KRAS抑制剂)提供了理论依据。
综上,本研究通过整合多维度ctDNA指标,明确了其对RET融合NSCLC患者治疗反应及进展的预测价值,为RET融合NSCLC的精准治疗监测提供了新的生物标志物策略,也为后续ctDNA引导的早期干预研究奠定了基础。
