1. 领域背景与文献引入
文献英文标题:RNAmotifs: prediction of multivalent RNA motifs that control alternative splicing;发表期刊:Genome Biol;影响因子:未公开;研究领域:可变剪接调控(RNA结合蛋白介导的基因表达调控)。
可变剪接是真核生物基因表达多样性的核心机制,约95%的人类基因通过可变剪接产生多个mRNA异构体,参与发育、分化及疾病发生(如肿瘤、神经疾病)。RNA结合蛋白(RBP)是可变剪接的关键调控因子,其作用遵循“位置依赖性原则”——结合外显子上游或内部通常抑制外显子 inclusion,结合下游则增强。然而,现有研究存在显著局限:多数RBP识别短(3-4nt)、简并的RNA基序,且需要多个基序簇状(多价)结合才能实现高亲和力调控(如NOVA蛋白需结合多个YCAY基序);但过去的基序预测方法多关注连续、非简并基序(如RBFOX识别的UGCAUG),依赖CLIP(交联免疫沉淀)等实验数据定义结合位点,无法从头识别多价简并基序。此外,CLIP方法存在序列偏差,限制了对未知RBP或组织特异性剪接的研究。
针对这一空白,本文开发了RNAmotifs计算方法,通过分析差异剪接外显子的基因组序列,从头识别调控可变剪接的多价RNA基序(短简并四聚体簇),并构建核苷酸分辨率的RNA剪接图谱,解析基序的位置依赖性功能。该方法不依赖CLIP数据或保守序列,为解析剪接调控网络及疾病机制提供了新工具。
2. 文献综述解析
本文综述的核心评述逻辑围绕“可变剪接调控的关键瓶颈——多价RNA基序的识别”展开,将现有研究分为三类:
1. 可变剪接的调控机制:RBP通过位置依赖性原则调控外显子 inclusion,如NOVA结合外显子下游增强剪接,结合上游抑制剪接;
2. 现有基序预测方法的局限:早期方法(如HITS-CLIP、PAR-CLIP)依赖实验数据定义结合位点,但存在序列偏差;计算方法多识别连续基序,无法处理简并或多价基序;
3. 多价基序的功能重要性:如NOVA、PTBP1等RBP需多个短基序簇状结合才能有效调控剪接,而过去的方法无法从头识别这类基序。
现有研究的关键结论包括:① RBP的调控功能严格依赖结合位置;② 多数RBP识别短简并基序,多价结合是其核心作用模式;③ CLIP方法揭示了RBP结合位点,但无法替代从头基序预测。现有方法的局限性在于:依赖实验数据、无法处理简并基序、无法解析多价结合的位置依赖性。
本文的创新价值在于:① 首次开发不依赖CLIP数据的多价RNA基序从头识别方法,引入简并四聚体(如YCAY)提高基序识别的全面性;② 构建核苷酸分辨率的RNA剪接图谱,直观展示基序的位置依赖性功能;③ 验证了方法在多个RBP数据集(如hnRNP C、PTBP1)及组织特异性剪接(脑vs心脏)中的有效性,为解析剪接调控网络提供了通用工具。
3. 研究思路总结与详细解析
整体框架概括
研究目标:开发RNAmotifs方法,识别调控可变剪接的多价RNA基序,解析其位置依赖性功能;
核心科学问题:如何从头识别多价简并RNA基序,及其在可变剪接中的位置依赖性调控;
技术路线:定义多价基序→选择感兴趣区域→统计分析基序富集→构建RNA剪接图谱→多数据集验证(RBP敲除/敲低、混合数据集、组织特异性数据)。
3.1 多价RNA基序的定义与区域选择
实验目的:明确多价RNA基序的特征及需要分析的基因组区域。
方法细节:① 基序定义:选择四聚体(多数RBP识别≤4nt)作为核心基序,包括256个非简并(ND)四聚体和64个简并(DG)四聚体(如YCAY,允许边界核苷酸为嘌呤R或嘧啶Y);② 多价定义:基序簇状分布,间距≤15nt(基于PTBP1、NOVA的研究);③ 感兴趣区域:基于NOVA的RNA剪接图谱,选择可变外显子周围3个区域——r1(3′剪接位点上游-35~-5nt内含子)、r2(外显子内部:3′剪接位点下游1~31nt + 5′剪接位点上游-31~-1nt)、r3(5′剪接位点下游10~40nt内含子)。
结果解读:简并四聚体(如YCAY)能识别更全面的调控位置,而非简并四聚体(如CCAT)仅富集于特定位置(如增强外显子下游),说明简并基序分析提高了基序识别的全面性。
产品关联:文献未提及具体实验产品,领域常规使用UCSC Genome Browser获取基因组序列、R语言进行序列分析。
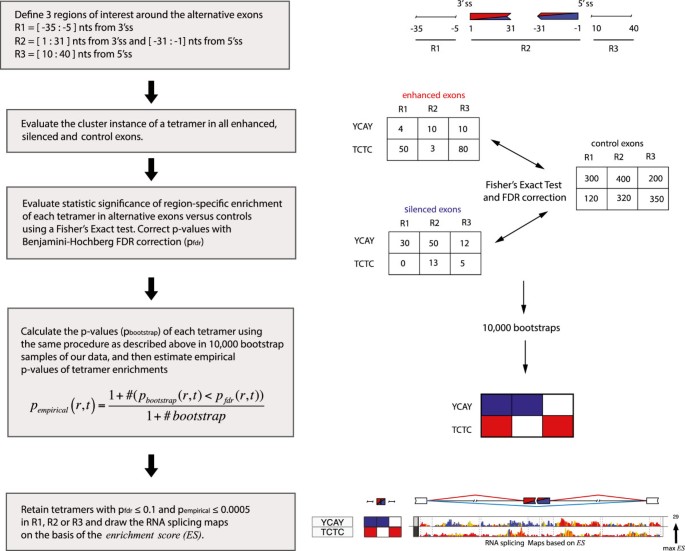
3.2 基序富集的统计分析
实验目的:验证基序在差异剪接外显子与对照外显子中的富集性。
方法细节:① 数据来源:收集RBP敲除/敲低后的差异剪接外显子数据(如NOVA2^{-/-}小鼠脑的98个增强外显子、70个沉默外显子),以及4200个对照外显子(无显著剪接变化);② 统计方法:使用Fisher精确检验比较基序簇在差异外显子与对照外显子中的出现频率,Benjamini-Hochberg校正多重检验(控制FDR≤0.1),通过10000次Bootstrap验证统计显著性(p_{empirical}≤0.0005);③ 阈值设定:保留同时满足p_{fdr}≤0.1和p_{empirical}≤0.0005的基序。
结果解读:对于NOVA调控的外显子,识别到14个富集的四聚体,其中8个属于YCAY家族(如TCAT、CCAT),这些基序在增强外显子下游(r3)和沉默外显子上游(r1)显著富集,与已知的NOVA调控位置完全一致;对于hnRNP C,富集的四聚体为TTTT和YTTY,主要在沉默外显子的3′剪接位点下游富集,与iCLIP数据一致。
产品关联:文献未提及具体实验产品,领域常规使用R语言进行Fisher精确检验和Bootstrap分析。
3.3 RNA剪接图谱的构建
实验目的:解析基序在可变剪接中的位置依赖性功能。
方法细节:① 位置特异性富集分析:计算每个基序在基因组位置(外显子-内含子 junction周围200nt内含子、50nt外显子)的富集得分(ES)——ES = -2(log(p_{enhanced}) + log(p_{silenced})),其中p_{enhanced}和p_{silenced}分别为基序在增强、沉默外显子中的Fisher精确检验p值;② 图谱可视化:用颜色编码ES(蓝色代表沉默子,红色代表增强子),展示基序在不同位置的调控功能。
结果解读:YCAY家族基序的RNA剪接图谱显示,其在增强外显子下游(r3)为红色(增强子),在沉默外显子上游(r1)为蓝色(沉默子),完全符合NOVA的调控模式;PTBP1的YTCY基序在沉默外显子的3′剪接位点上游(r1)为蓝色(沉默子),与已知的PTBP1抑制功能一致;hnRNP C的TTTT基序主要在沉默外显子的3′剪接位点下游富集(蓝色),说明其主要功能是抑制剪接。
产品关联*:文献未提及具体实验产品,领域常规使用ggplot2等R包进行图谱可视化。
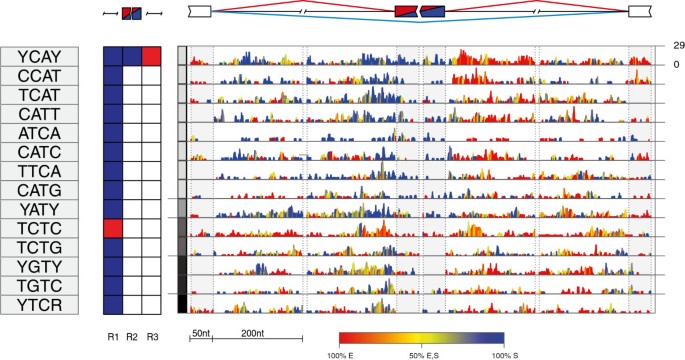
3.4 方法验证与应用
实验目的:验证RNAmotifs在复杂数据集(如混合RBP数据、组织特异性数据)中的有效性。
方法细节:① RBP数据集验证:分析hnRNP C、PTBP1、TIA等RBP敲除/敲低后的差异外显子,验证预测的基序是否与已知RBP的结合特异性一致;② 混合数据集验证:将hnRNP C、PTBP1、TIA的差异外显子混合,验证RNAmotifs能否识别各RBP的特征基序;③ 组织特异性验证:分析脑与心脏的差异剪接外显子,预测调控组织特异性剪接的基序。
结果解读:① RBP数据集验证:预测的基序与已知RBP的结合特异性完全一致(如hnRNP C的TTTT、PTBP1的YTCY、TIA的TTTA);② 混合数据集验证:即使混合多个RBP的数据,RNAmotifs仍能识别各RBP的特征基序,且位置与单独分析一致;③ 组织特异性验证:脑与心脏的差异剪接外显子中,YCAY基序(NOVA结合)在脑增强外显子下游富集(红色),YTCY基序(PTBP1结合)在心脏增强外显子上游富集(蓝色),与NOVA在脑高表达、PTBP1在心脏高表达的模式一致。
产品关联:文献未提及具体实验产品,领域常规使用 exon junction microarray或RNA-seq获取差异剪接数据。
4. Biomarker研究及发现成果解析
本文的“Biomarker”为调控可变剪接的多价RNA基序,属于功能性Biomarker,能反映RBP的结合特异性和位置依赖性功能。
Biomarker定位与筛选逻辑
Biomarker类型:多价RNA基序,如YCAY(NOVA)、TTTT(hnRNP C)、YTCY(PTBP1)、RTGY(TARDBP)、TTTA(TIA)等;
筛选/验证逻辑:① 基于差异剪接外显子数据,分析其周围区域的四聚体簇;② 通过Fisher精确检验和Bootstrap验证基序富集;③ 构建RNA剪接图谱确认位置依赖性;④ 与已知RBP的结合数据(如CLIP)对比验证。
研究过程详述
- Biomarker来源:差异剪接外显子周围的基因组序列(r1、r2、r3区域);
- 验证方法:① 统计验证:基序在差异外显子与对照外显子中的富集(Fisher精确检验,p_{fdr}≤0.1,p_{empirical}≤0.0005);② 位置验证:RNA剪接图谱展示基序的位置依赖性功能;③ 功能验证:与已知RBP的结合特异性对比(如YCAY对应NOVA,TTTT对应hnRNP C);
- 特异性与敏感性:以NOVA的YCAY基序为例,其在增强外显子下游(r3)的富集特异性为p_{empirical}≤0.0005,敏感性覆盖>10%的增强外显子(n=98);脑与心脏差异剪接中,YCAY基序覆盖>15%的脑增强外显子(n=120)。
核心成果提炼
- 功能性Biomarker的鉴定:这些多价RNA基序是RBP调控可变剪接的“分子标签”,如YCAY对应NOVA,TTTT对应hnRNP C,能准确反映RBP的结合特异性和位置依赖性功能;
- 创新性:首次从头识别了多价简并RNA基序,不依赖CLIP数据,如在混合数据集和组织特异性数据中仍能有效识别特征基序;
- 统计结果:NOVA的YCAY基序在增强外显子下游的p_{empirical}≤0.0005(n=98),脑与心脏差异剪接中YCAY基序的覆盖度>15%(n=120);PTBP1的YTCY基序在沉默外显子上游的p_{empirical}≤0.0005(n=80)。
应用价值
这些多价RNA基序可作为剪接调控的功能Biomarker,用于:① 预测未知RBP的结合特异性;② 解析组织特异性剪接的调控机制;③ 识别疾病相关的剪接异常(如肿瘤中PTBP1的YTCY基序富集导致的剪接重编程)。
总结
本文开发的RNAmotifs方法,首次实现了不依赖CLIP数据的多价RNA基序从头识别,构建了核苷酸分辨率的RNA剪接图谱,解析了基序的位置依赖性功能。该方法为解析剪接调控网络、识别疾病相关剪接异常提供了重要工具,也为后续研究(如疾病 Biomarker开发、RBP靶向治疗)奠定了基础。
