1. 领域背景与文献引入
文献英文标题:A graph neural network-based interpretable framework reveals a novel DNA fragility–associated chromatin structural unit;发表期刊:Genome Biology;影响因子:17.906;研究领域:3D基因组学与DNA损伤机制。
DNA双链断裂(DSB)是对基因组完整性最具危害性的DNA损伤,可由转录、DNA复制或遗传毒性剂暴露引发,若修复不当会导致遗传不稳定性,进而促进癌症发生。近年来,Hi-C等染色体构象捕获技术的发展揭示了3D染色质结构与DSB的关联——例如loop锚点易成为DSB热点,但现有研究仍存在核心限制:其一,多数研究聚焦拓扑关联域(TAD)和染色质环(loop)等经典结构,未从全局Hi-C接触图系统解析与DNA脆性相关的染色质特征;其二,缺乏从Hi-C图中精准识别DNA脆性相关结构单元的方法;其三,对DSB形成的空间组织决定因素(如染色质相互作用的全局调控)关注不足。针对这些问题,本研究开发了基于图神经网络(GNN)的可解释框架,整合Hi-C数据与GNNExplainer技术,旨在识别新型DNA脆性相关染色质结构单元,解析3D基因组 context下的DSB形成机制。
2. 文献综述解析
文献综述围绕“DSB危害-现有研究进展-核心局限性”展开评述:首先,DSB作为癌症发生的关键驱动因素,其基因组分布已通过DSBCapture、BLESS等技术实现全基因组 mapping,且已知DSB偏好位于TEAD基序、启动子/增强子等调控元件及H3K4me1/2/3等表观标记区域;其次,3D染色质结构与DSB的关联已被初步揭示——例如loop锚点因拓扑应力易受DSB影响,但现有研究多聚焦局部结构(如TAD、loop),未从全局Hi-C图解析DSB的形成规律;最后,作者指出现有研究的三大局限:(1)未系统解析全局Hi-C图中与DNA脆性相关的特征;(2)缺乏从Hi-C数据中提取DNA脆性相关结构单元的有效方法;(3)忽略了DSB形成的空间组织决定因素。
本研究的创新价值在于:首次将GNN与可解释技术结合,从全局Hi-C图中识别出新型DNA脆性相关染色质结构单元(FaCIN),突破了传统研究仅关注局部结构的局限,为3D基因组 context下的DSB形成机制提供了更系统的解释。
3. 研究思路总结与详细解析
本研究的核心目标是解析3D染色质结构与DSB的关系,核心科学问题是“3D基因组如何通过染色质相互作用调控DSB形成”,技术路线为“Hi-C图转Graph→GNN模型训练→GNNExplainer解释→FaCIN识别→功能验证”的闭环。
3.1 数据准备与Graph构建
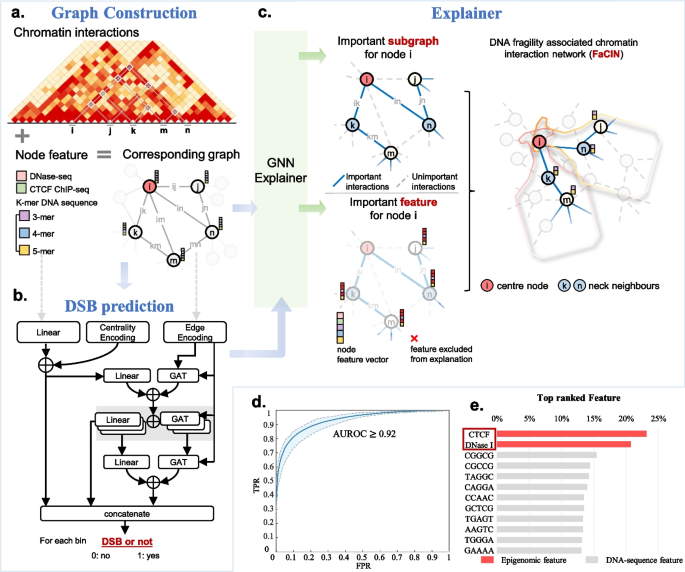
实验目的:将Hi-C数据转换为GNN可处理的图结构,整合基因组与染色质相互作用信息。
方法细节:使用正常人类表皮角质形成细胞(NHEK)的Hi-C数据,将每条染色体(共23条,不含Y染色体)转化为无向加权图——节点代表5kb基因组bin,边代表Hi-C接触(权重为原始接触计数),节点特征包括k-mer(3、4、5-mer)频率、CTCF ChIP-seq信号及DNase I信号(总长度1346维)。
结果解读:成功构建了覆盖全基因组的染色质相互作用图,为后续GNN模型提供了结构 scaffold。
产品关联:文献未提及具体实验产品,领域常规使用Hi-C文库构建试剂盒(如Arima Hi-C Kit)、ChIP-seq试剂盒(如Illumina TruSeq ChIP Library Prep Kit)及DNase-seq试剂盒(如Epicentre DNase I Kit)。
3.2 DSB-GNN模型构建与训练
实验目的:开发基于GNN的DSB预测模型,捕获3D染色质结构与DSB的关联。
方法细节:模型包含3层图注意力卷积(GAT)层(整合自注意力机制)、跳跃知识结构(融合多尺度节点特征)及边/节点编码(保留染色质相互作用强度与节点属性);采用23折交叉验证(每条染色体作为测试集一次),以交叉熵为损失函数,使用PyTorch和DGL库在GPU 2080 Ti上训练。
结果解读:模型预测准确率高,平均受试者工作特征曲线下面积(AUC)>0.92(n=23,每条染色体作为测试集一次);消融实验显示,自注意力机制、跳跃知识结构及Hi-C/节点特征均对模型性能有贡献(如移除Hi-C数据后AUC下降)。
产品关联:实验使用PyTorch(v1.7.0)、Deep Graph Library(DGL,v0.6.0)实现模型,硬件为NVIDIA GeForce RTX 2080 Ti GPU。
3.3 GNNExplainer解释与FaCIN识别
实验目的:解释DSB-GNN的预测结果,提取与DNA脆性相关的染色质结构单元。
方法细节:使用GNNExplainer对每个节点的预测进行可解释分析,提取Top10对预测影响最大的边,定义为DNA脆性相关染色质相互作用网络(FaCIN);通过介数中心性计算(衡量节点在最短路径中的中介作用)验证FaCIN的瓶颈模式——将1-hop邻居定义为“neck邻居”(瓶颈位置),其连接边为“neck相互作用”。
结果解读:FaCIN呈现典型的瓶颈模式,neck邻居的介数中心性显著高于2-hop邻居(文献中的计算结果);基序富集分析显示,FaCIN富集“cascade”和“bifurcate”两种非随机结构基序(图2c),而随机图无此富集,证明FaCIN的组织具有生物学意义。
图片:
< img src="https://media.springernature.com/lw685/springer-static/image/art%3A10.1186%2Fs13059-023-02916-x/MediaObjects/13059_2023_2916_Fig2_HTML.png" alt="Fig.2 FaCIN的瓶颈模式" >
3.4 FaCIN的功能验证
实验目的:验证FaCIN的生物学相关性,解析其对DSB形成的调控作用。
方法细节:1)将FaCIN与已知染色质结构(loop、TAD)重叠分析,验证neck相互作用与经典结构的关联;2)比较DSB与非DSB位点的neck相互作用特征(如loop锚点富集、表观标记水平)。
结果解读:neck相互作用显著富集于loop锚点(p<0.001,超几何检验,图3a),且80%以上的neck相互作用位于单个TAD内(p<0.001,超几何检验,图3d);DSB位点的neck邻居CTCF和DNase I信号显著高于非DSB位点(图4e),说明neck邻居的高染色质 accessibility和CTCF富集促进DSB形成。
图片:< img src="https://media.springernature.com/lw685/springer-static/image/art%3A10.1186%2Fs13059-023-02916-x/MediaObjects/13059_2023_2916_Fig3_HTML.png" alt="Fig.3 neck相互作用与TAD/loop的关联" >
< img src="https://media.springernature.com/lw685/springer-static/image/art%3A10.1186%2Fs13059-023-02916-x/MediaObjects/13059_2023_2916_Fig4_HTML.png" alt="Fig.4 DSB与非DSB位点的neck相互作用特征比较" >
4. Biomarker研究及发现成果解析
Biomarker定位与筛选逻辑
本研究中的Biomarker为DNA脆性相关染色质结构单元(FaCIN),属于“结构型Biomarker”(基于染色质相互作用的组织模式)。其筛选逻辑为:1)基于Hi-C图构建GNN模型,捕获3D染色质结构与DSB的关联;2)通过GNNExplainer解释模型预测,提取对DSB预测最具影响的染色质相互作用(Top10边),定义为FaCIN;3)通过与经典染色质结构(loop、TAD)的关联及DSB/非DSB位点的比较,验证FaCIN的生物学相关性。
研究过程详述
FaCIN来源于NHEK细胞的Hi-C数据(覆盖全基因组23条染色体),验证方法包括:1)结构特征验证:通过介数中心性计算,确认neck邻居(1-hop邻居)在FaCIN中的瓶颈位置(中介作用更强);2)组织模式验证:基序富集分析显示,FaCIN富集“cascade”和“bifurcate”基序(非随机组织),而随机图无此富集;3)生物学关联验证:neck相互作用显著富集于loop锚点(p<0.001)和TAD内(p<0.001),与已知DSB热点区域重叠;4)功能验证:DSB位点的neck邻居CTCF和DNase I信号显著高于非DSB位点(文献中的统计结果),说明FaCIN的结构特征与DSB形成直接相关。
模型性能数据:DSB-GNN的平均AUC>0.92(n=23),验证了FaCIN对DSB的预测能力;neck相互作用的富集分析显示,其与DSB的关联具有统计学显著性(p<0.001)。
核心成果提炼
- 新型结构单元发现:FaCIN是首次报道的DNA脆性相关染色质结构单元,呈现“瓶颈模式”——neck邻居作为1-hop节点,介导预测位点与全局基因组的相互作用,是DSB形成的关键结构枢纽。
- 结构决定因素解析:neck相互作用是DSB形成的染色质结构决定因素,其富集的loop锚点、CTCF结合及高染色质 accessibility共同促进DSB发生。
- 机制解释:FaCIN揭示了3D基因组 context下的DSB形成机制——预测位点通过neck邻居整合全局染色质相互作用的信息,neck邻居的表观特征(如CTCF富集)决定了DNA脆性。
统计学结果:neck相互作用富集于loop锚点(p<0.001,超几何检验);DSB位点的neck邻居CTCF信号(p<0.05)和DNase I信号(p<0.05)显著高于非DSB位点(文献中的统计结果)。
本研究通过GNN与可解释技术的结合,突破了传统3D基因组研究的局限,为DSB形成机制提供了更系统的理解,FaCIN有望成为3D基因组学与癌症研究的新型Biomarker。
