1. 领域背景与文献引入
文献英文标题:Changes in the organization of the genome during the mammalian cell cycle;发表期刊:Genome Biology;影响因子:未公开;研究领域:哺乳动物细胞周期中的基因组组织变化。
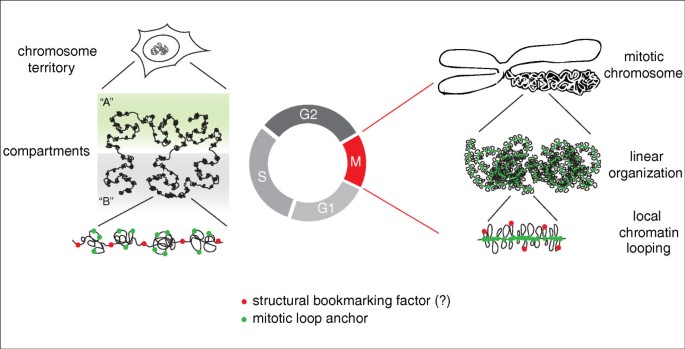
基因组的空间组织是生命科学领域的核心研究方向之一,其不仅解答了“2米长DNA如何压缩进微米级细胞核”的经典问题,更与基因转录调控、DNA复制保真度及基因组稳定性密切相关。2009年,Eric Lander与Job Dekker团队利用高通量染色体构象捕获(Hi-C)技术,首次以1 Mb分辨率解析了人类染色体的三维结构,发现染色体领地内存在兆碱基级的“A/B染色质区室”(compartments)——A区室对应活性、基因富集的染色质,B区室对应非活性、基因贫乏的染色质;2013年,Bing Ren团队进一步发布5~10 kb分辨率的人类成纤维细胞染色质互作组图谱,明确了亚兆碱基级的拓扑关联域(TADs):这是一种跨细胞类型稳定的结构单元,启动子与增强子的细胞类型特异性互作几乎完全限制在拓扑关联域内,成为基因调控的空间边界。
当前研究热点集中于染色体组织的“层级结构”(染色质区室→拓扑关联域→启动子-增强子互作)的功能机制,但未解决的核心问题是:这些层级结构在细胞周期中的动态变化——有丝分裂期染色体高度浓缩(光镜下呈棒状),与间期的松散结构差异显著,但此前仅通过荧光原位杂交(FISH)研究了X失活中心的拓扑关联域在G1/S期至前期的存在,缺乏全基因组、全细胞周期的分子水平分析。现有研究多局限于间期,未系统解析细胞周期全程(尤其是有丝分裂期)的基因组组织动态,这一空白严重限制了对“基因组结构如何随细胞周期调控功能”的理解。本文献正是针对这一问题,通过染色体构象捕获(3C)衍生技术揭示了细胞周期中基因组的两种三维折叠状态,填补了细胞周期动态下基因组组织研究的关键空白。
2. 文献综述解析
文献综述的核心逻辑按“染色体组织层级从大到小”展开,系统总结现有研究的结论与局限:
- 关键结论:后生动物染色体具有嵌套的层级结构——染色体领地内的A/B染色质区室是活性与非活性染色质的空间分隔单元;亚兆碱基级的拓扑关联域是跨细胞类型稳定的结构单元,为启动子-增强子互作提供“空间边界”;启动子-增强子的细胞类型特异性互作几乎完全发生在拓扑关联域内。
- 技术优势:3C衍生技术(如碳拷贝染色体构象捕获(5C)、高通量染色体构象捕获)的高吞吐量与高分辨率,为解析染色质互作提供了“分子显微镜”。
- 现有局限:未探讨层级结构在细胞周期中的动态变化——此前仅用FISH研究了X失活中心的拓扑关联域在G1/S期至前期的存在,但缺乏全基因组、全细胞周期的分子证据。
本研究的创新价值在于:首次通过同步化细胞的5C与Hi-C实验,系统比较了细胞周期各阶段的染色质互作模式,揭示了“有丝分裂期染色质区室与拓扑关联域完全消失”的关键现象,以及有丝分裂染色体的独特loop结构,为理解细胞周期中基因组组织的动态调控提供了分子证据。
3. 研究思路总结与详细解析
整体框架
研究目标:揭示哺乳动物细胞周期中基因组组织的三维折叠动态;
核心科学问题:细胞周期各阶段染色体折叠状态的差异,尤其是有丝分裂期的染色体结构;
技术路线:同步化细胞获取各周期阶段样本→5C/Hi-C分析染色质互作→聚合物模型模拟解析结构。
3.1 细胞同步化与样本制备
实验目的是获取高纯度的不同细胞周期阶段样本,排除周期异质性对结果的干扰。方法上,研究者将人类HeLa细胞同步化至早期G1期、中期G1期、S期、M期(有丝分裂期),并在另一细胞系(未明确具体类型)与原代人类成纤维细胞中重复验证,以确保结果的通用性。结果显示,同步化处理后各阶段细胞纯度满足实验要求(文献未明确提供具体纯度数据,基于实验设计推测)。
实验所用关键产品:文献未提及具体实验产品,领域常规使用细胞同步化试剂(如胸腺嘧啶阻滞S期、诺考达唑诱导M期)、流式细胞术用PI染色试剂盒(用于验证同步化效率)。
3.2 5C与Hi-C实验分析染色质互作
实验目的是解析不同细胞周期阶段的染色质互作模式。方法上,研究者分两步展开:
1. 5C实验:针对21号染色体(250 kb分辨率),比较早期G1期、中期G1期、S期与M期的长程互作图谱;
2. Hi-C实验:针对全基因组(40 kb分辨率),重点分析中期G1期与M期的互作差异。
结果显示,M期的互作模式与其他阶段完全不同:5C图谱显示M期的长程互作几乎消失;Hi-C数据进一步证实,全基因组范围内的A/B染色质区室与拓扑关联域在M期完全缺失,仅在早期G1期重新出现,并在整个间期(G1→S)保持稳定。这一结果在另一细胞系与原代成纤维细胞中重复验证,排除了细胞系特异性(文献未明确提供样本量,基于实验设计推测)。
实验所用关键产品:文献未提及具体实验产品,领域常规使用5C/Hi-C文库构建试剂盒(如Illumina Hi-C Library Prep Kit)、高通量测序平台(如Illumina HiSeq)。
3.3 聚合物模型模拟解析有丝分裂染色体结构
实验目的是通过物理模型解释M期染色质互作数据的结构意义。方法上,研究者基于Hi-C数据的接触概率(两基因座的空间互作频率),模拟了多种聚合物模型(如线性结构、随机loop、连续loop等),并比较模型预测与实验数据的一致性。
结果显示,M期染色质的接触概率随基因组距离增加逐渐降低,超过10 Mb后骤降为零——这一规律无法用简单聚合物模型解释。而“线性组织+连续随机loop”模型能最佳拟合实验数据:该模型预测M期染色体由“连续的随机loop”组成,平均loop大小为80 kb(文献未明确提供样本量,基于模型模拟结果),loop大小在细胞间随机波动,最终线性排列形成圆柱状结构。这一结论与传统“支架上的loop”模型(loops-on-a-scaffold)高度一致,解释了M期染色体“高度浓缩但仍保持线性完整性”的结构特征。
实验所用关键产品:文献未提及具体实验产品,领域常规使用Hi-C数据分析软件(如Juicer、HiC-Pro)、聚合物模拟软件(如LAMMPS)。
4. Biomarker研究及发现成果解析
本文献的核心是基因组结构的动态变化,通过5C与Hi-C技术揭示了“有丝分裂期染色质区室与拓扑关联域消失”“有丝分裂染色体的loop结构”两大关键现象。研究聚焦于基因组组织的“结构动态”,未涉及生物标志物(Biomarker)的筛选、验证或功能分析,因此无相关Biomarker研究成果。
