1. 领域背景与文献引入
文献英文标题:ONTraC characterizes spatially continuous variations of tissue microenvironment through niche trajectory analysis;发表期刊:Genome Biology;影响因子:未公开;研究领域:空间转录组学与组织微环境分析。
空间转录组学技术的发展为解析组织微环境的空间异质性提供了契机,但其核心挑战在于捕捉组织微环境的连续空间变化。现有研究可分为两类:一类是离散空间模式识别(如细胞邻域[1-3]、空间域[4-12]),仅能识别非连续的空间结构;另一类是空间轨迹分析,传统方法(如pseudotime[13-17])基于基因表达相似性构建轨迹,完全忽略空间信息;改进方法(如stLearn[18]、SpaceFlow[7]、spatialPCA[19])虽结合空间信息,但仍未解决细胞状态与空间连续性的内在冲突——即基于基因表达的细胞状态轨迹可能与物理空间的连续变化不一致。
针对这一空白,本研究开发了有序生态位轨迹构建(ONTraC)框架,以多细胞生态位(而非单个细胞)为基本单位,通过图神经网络整合空间信息与细胞类型组成,构建空间连续的生态位轨迹,旨在克服现有方法的局限,为组织微环境的连续变化分析提供新工具。
2. 文献综述解析
作者对现有研究的分类逻辑清晰:首先区分离散空间模式识别与空间轨迹分析两大方向,再进一步细化空间轨迹分析的发展(传统pseudotime到结合空间的改进方法)。
现有研究的核心结论与局限
- 离散空间模式识别:通过细胞邻域或空间域划分,揭示组织的离散空间结构(如肿瘤边界、皮层分层),但无法捕捉连续的空间变化。
- 传统pseudotime分析:基于基因表达相似性构建细胞状态轨迹,用于解析发育或分化过程,但完全忽略空间信息,导致轨迹与物理空间脱节。
- 结合空间的改进方法:如stLearn(融合pseudotime与空间距离)、SpaceFlow(用空间嵌入优化pseudotime)、spatialPCA(空间正则化的主成分分析),虽提升了空间相关性,但仍未解决细胞状态与空间连续性的冲突——即细胞状态的连续变化可能与空间的连续变化不一致。
本研究的创新价值
作者通过三个关键创新突破现有局限:①以多细胞生态位为基本单位,而非单个细胞,天然整合了局部空间的细胞互作信息;②用图卷积网络(GCN)整合空间邻接与细胞类型组成,保持空间连续性与属性相似性;③通过图池化与轨迹排序,直接构建空间连续的生态位轨迹,避免了细胞状态与空间的冲突。
3. 研究思路总结与详细解析
本研究的核心目标是开发ONTraC框架,构建生态位水平的空间轨迹,表征组织微环境的连续变化。技术路线遵循“生态位定义→信息编码→轨迹构建→验证应用”的闭环逻辑,分以下关键环节:
3.1 生态位网络构建
实验目的:定义多细胞生态位,量化细胞与生态位的关联,总结生态位的属性。
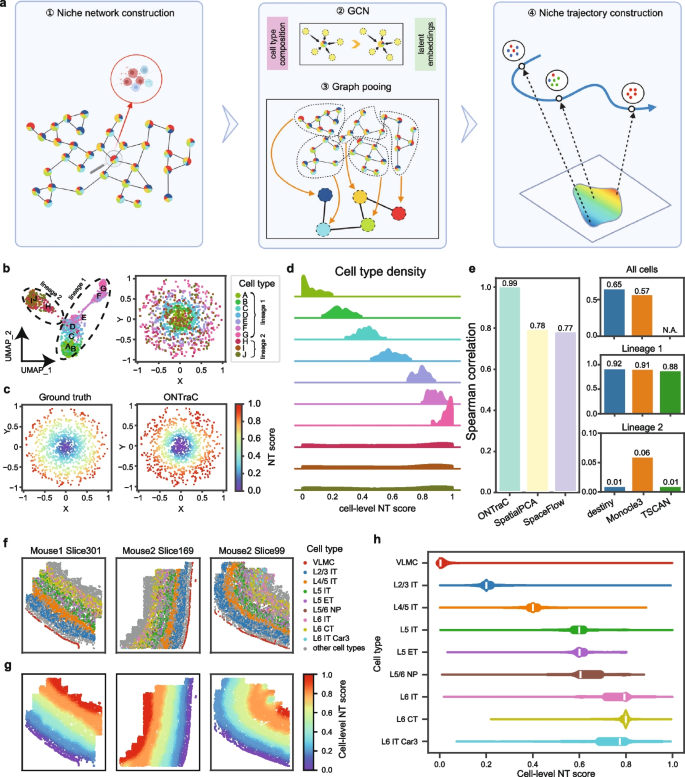
方法细节:①每个细胞作为“锚点细胞”,包含其k近邻(默认k=50),形成以该细胞为中心的多细胞生态位;②细胞-生态位关联分数:用指数函数量化细胞i与锚点细胞j的生态位的关联((w_{ij} = e^{-d_{ij}^2/sigma_j^2}),(d_{ij})为细胞i与锚点细胞的物理距离,(sigma_j)为锚点细胞第20近邻的距离,确保关联分数随距离衰减);③生态位属性:用细胞类型组成向量总结((v_{jm} = sum_{i}w_{ij}I_{im}/sum_{i}w_{ij}),(I_{im})为细胞i是否属于类型m的二元指标)。
结果解读:成功构建了覆盖所有细胞的生态位网络,每个生态位的属性由其包含的细胞类型组成决定,为后续轨迹分析提供了基础。
产品关联:文献未提及具体实验产品,领域常规使用Python的NumPy、Pandas库处理空间坐标与细胞类型数据。
3.2 图卷积网络(GCN)编码与低维嵌入
实验目的:将生态位的空间邻接与细胞类型属性整合为低维特征向量,同时保持空间连续性。
方法细节:①使用两层GCN模型,输入为生态位的细胞类型组成向量,输出为低维嵌入(默认维度4);②GCN公式:(X_t = ext{SeLU}(( ilde{A} + I)X_{t-1}W_t)),其中( ilde{A})是归一化的生态位邻接矩阵(( ilde{A} = D^{-1/2}AD^{1/2}),(D)为度矩阵),(W_t)为可训练参数。
结果解读:GCN编码将生态位的空间信息与细胞类型属性融合为低维向量,既保持了生态位间的空间连续性(相邻生态位的嵌入相似),又保留了属性相似性(细胞类型组成相似的生态位嵌入相似)。
产品关联:文献未提及具体实验产品,领域常规使用PyTorch、PyTorch Geometric实现图神经网络模型。
3.3 生态位簇识别与轨迹构建
实验目的:识别生态位簇,构建空间连续的生态位轨迹,并计算细胞水平的轨迹分数(NT分数)。
方法细节:①生态位簇识别:用改进的图池化方法(基于GCN嵌入)概率性聚类生态位,得到生态位簇网络;②轨迹排序:通过最大化簇间边缘连接性((sum_{p_k=1}^{K-1} ilde{E}(p_k,p_{k+1})))确定簇的顺序,为每个簇分配分数((s(p_k) = (k-1)/(K-1)),范围0~1);③生态位NT分数:生态位j的分数为其所属簇分数的加权和((s_j = sum_{k=1}^K c_{jk}s(k)),(c_{jk})为生态位j属于簇k的概率);④细胞NT分数:细胞i的分数为其所在所有生态位NT分数的加权平均(( ilde{s}i = sum_j w{ij}s_j/sum_j w_{ij}))。
结果解读:成功构建了生态位轨迹(NT),输出的NT分数定量反映了生态位/细胞在空间轨迹中的位置——分数越高,对应轨迹的“末端”(如分化晚期、肿瘤周边)。
产品关联:文献未提及具体实验产品,领域常规使用scikit-learn库进行聚类分析。
3.4 模拟数据集验证
实验目的:验证ONTraC的准确性与鲁棒性。
方法细节:①生成4个模拟数据集(包含不同空间模式:环形、线性、非线性、分离轨迹;不同细胞 lineage 关系:分叉、不连通);②与5种现有方法比较:传统pseudotime(destiny[14]、Monocle 3[17]、TSCAN[15])、结合空间的方法(SpaceFlow[7]、spatialPCA[19]);③用Spearman相关系数评估轨迹与真实值的一致性。
结果解读:ONTraC在所有模拟数据集上的性能均优于现有方法:例如模拟数据集1(环形轨迹)中,ONTraC的Spearman相关系数达0.99(n=1000细胞),而destiny仅0.65、Monocle 3仅0.57;对参数(如GCN层数、k值)具有鲁棒性——调整参数后结果仍稳定。
产品关联:文献未提及具体实验产品,领域常规使用dyngen生成模拟单细胞转录组数据。
3.5 真实数据集应用
实验目的:验证ONTraC在真实组织中的实用性。
方法细节:应用于3类真实空间转录组数据:①小鼠运动皮层MERFISH(64张切片,28万个细胞,258基因 panel);②小鼠胚胎背中脑stereo-seq(E12.5、E14.5、E16.5,2.7万个细胞);③乳腺癌Xenium(6912个细胞,包含肿瘤与非肿瘤细胞)。
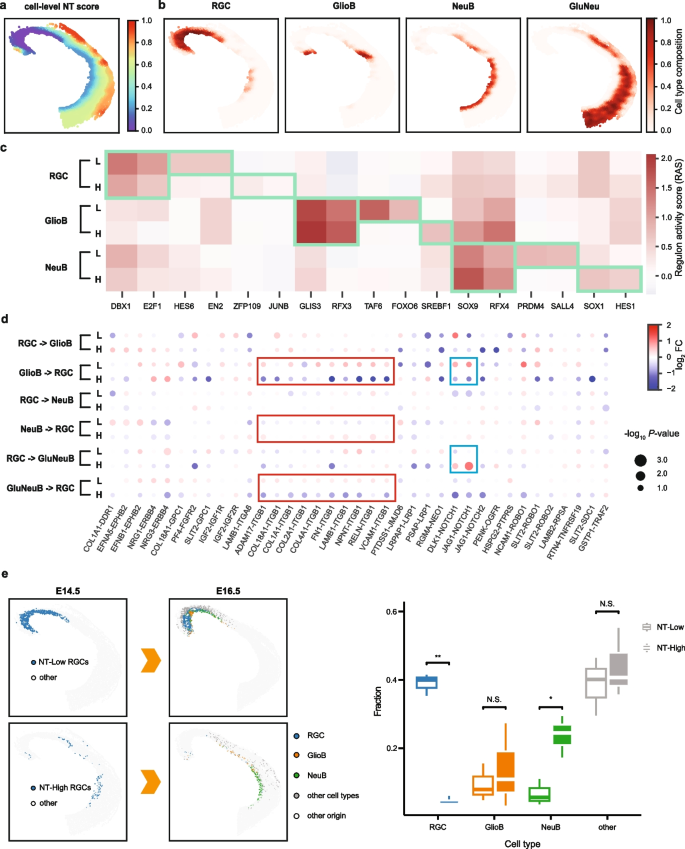
结果解读:①小鼠运动皮层:NT分数与皮层深度高度相关(87%样本的Spearman相关系数≥0.90),且捕捉到层内的连续变化(如L4标记基因Rspo1、L5标记基因Fezf2的层内表达梯度);②小鼠背中脑:NT分数从尾侧到吻侧、腹侧到背侧连续变化,径向胶质细胞(RGC)的NT分数与分化状态正相关——分数越高,分化程度越高;③乳腺癌:NT分数反映肿瘤核心到周围组织的连续变化,肿瘤核心富集侵袭性细胞,边界富集增殖性细胞,周围组织依次富集免疫细胞、内皮细胞。
产品关联:文献未提及具体实验产品,领域常规使用Seurat、Giotto处理空间转录组数据。
4. Biomarker研究及发现成果解析
Biomarker定位与筛选逻辑
本研究的Biomarker为与生态位微环境连续变化相关的基因(如Ppia、Ccnd2、Efna5、Cdkn1c等)。筛选逻辑遵循“生态位轨迹关联→功能验证”的流程:①基于E14.5小鼠背中脑的RGC细胞,按NT分数排序并构建元细胞(每10个细胞合并为一个元细胞);②计算元细胞基因表达与NT分数的Pearson相关系数;③筛选P<0.01且|相关系数|>0.4的基因,共得到109个候选Biomarker。
研究过程详述
- Biomarker来源:小鼠胚胎背中脑的RGC细胞(空间转录组数据);
- 验证方法:①相关性分析:Pearson系数验证基因表达与NT分数的线性关联;②功能富集:基因集富集分析(GSEA)显示,候选基因显著富集“DNA复制起始”(NES=-2.36,P<1×10^-308)、“非经典WNT信号通路”(NES=1.80,P=2.0×10^-3)等与干细胞更新、神经发生相关的通路;③调控网络分析:SCENIC分析发现,候选基因的调控因子(如HES6、EN2)的活性随NT分数变化——NT分数越高,调控因子活性越强,提示其参与分化过程。
核心成果提炼
- 功能关联:候选Biomarker直接参与组织微环境介导的细胞状态变化。例如:①Ppia(肽酰脯氨酰异构酶A)促进海马神经发生[26];②Ccnd2(细胞周期蛋白D2)调控神经干细胞的细胞周期[27];③Efna5( Ephrin-A5)参与海马神经发生与血管形成[28]。
- 创新性:首次在生态位水平鉴定与组织微环境连续变化相关的基因Biomarker,突破了传统离散空间域方法的局限——例如GraphPCA无法检测到层内的基因表达梯度,而ONTraC通过NT分数捕捉到了这一连续变化。
- 统计学结果:109个Biomarker中,95个与NT分数正相关(如Ccnd2,相关系数=0.52,P=1.2×10^-4),14个负相关(如Ppia,相关系数=-0.45,P=3.1×10^-3)。
综上,ONTraC通过以生态位为单位的空间轨迹分析,为组织微环境的连续变化研究提供了新工具,其鉴定的基因Biomarker为解析微环境介导的细胞状态调控提供了分子靶点。