1. 领域背景与文献引入
文献英文标题:A global transcriptional analysis of Plasmodium falciparum malaria reveals a novel family of telomere-associated lncRNAs;发表期刊:Genome Biology;影响因子:未公开;研究领域:疟疾分子生物学、长链非编码RNA表观调控
恶性疟原虫是人类最严重疟疾类型的病原体,全球约33亿人面临感染风险,非洲儿童为最易感群体。过往研究表明,疟原虫在红细胞内发育阶段有近80%的基因呈阶段特异性表达,其中毒力基因家族(如var基因)的互斥表达是其实现免疫逃逸的核心机制,但该调控过程的分子机制长期未明确。传统真核生物转录调控机制在疟原虫中存在明显缺失:缺乏经典的序列特异性转录因子、RNA干扰系统及DNA甲基化修饰,后续发现的ApiAP2转录因子家族和染色质重塑机制仅能部分解释疟原虫的基因表达调控,仍存在大量研究空白。
长链非编码RNA(lncRNA)在真核生物表观调控中扮演关键角色,如端粒重复序列RNA(TERRA)参与端粒维持、Xist RNA介导X染色体失活,但疟原虫中的长链非编码RNA研究处于起步阶段,尚未有系统的鉴定与功能探索。针对这一领域空白,本研究通过高分辨率转录组分析技术,首次系统鉴定恶性疟原虫红细胞内发育阶段的长链非编码RNA,并重点解析了一类新型端粒相关长链非编码RNA家族的功能潜力,为疟原虫转录调控机制研究提供了新方向。
2. 文献综述解析
作者对领域内研究的评述逻辑分为三个维度:疟原虫转录调控的现有机制、真核生物长链非编码RNA的调控范式、疟原虫转录组研究的技术局限性。
现有研究中,疟原虫转录调控的核心进展包括ApiAP2转录因子家族的发现,以及染色质重塑在毒力基因沉默中的作用,但这些机制无法完全解释疟原虫基因表达的阶段特异性及var基因的互斥表达;真核生物领域的研究已证实长链非编码RNA可通过结合染色质重塑复合物、转录因子等调控基因表达,为疟原虫的研究提供了理论参考;过往疟原虫转录组研究多采用低覆盖度芯片,未深入挖掘基因间区的转录活性,导致长链非编码RNA的鉴定严重不足。
本研究的创新价值在于突破了传统转录组分析的技术限制,使用覆盖22.6%疟原虫基因组的高分辨率tiling芯片,首次系统鉴定出60个恶性疟原虫长链非编码RNA候选,并发现了一类全新的端粒相关长链非编码RNA家族(lncRNA-TARE),填补了疟原虫非编码RNA表观调控研究的空白,为解析疟原虫毒力基因调控机制提供了新的研究靶点。
3. 研究思路总结与详细解析
本研究的整体框架为:以“长链非编码RNA是否参与恶性疟原虫转录调控”为核心科学问题,通过高分辨率tiling芯片全面检测疟原虫红细胞内发育阶段的转录活性,筛选并鉴定长链非编码RNA候选,重点表征端粒相关长链非编码RNA家族的表达模式、结构特征及调控关联,最终提出其参与端粒维持与毒力基因调控的功能模型。
3.1 高分辨率Tiling芯片实验设计与转录活性检测
实验目的:全面捕获恶性疟原虫红细胞内发育阶段的所有转录活性区域,包括蛋白编码基因与非编码RNA转录本。
方法细节:设计覆盖22.6%恶性疟原虫3D7株基因组的tiling芯片,探针中位分辨率为12bp,覆盖561条Watson链蛋白编码基因、699条Crick链蛋白编码基因、2个核糖体RNA基因及1.73Mb基因间序列。收集同步化的疟原虫红细胞内发育阶段样本(环期18hpi、环/滋养体期24hpi、滋养体期30hpi、滋养体/裂殖体期36hpi),提取总RNA后进行poly-A富集、Cy3/Cy5荧光标记,与芯片杂交。
结果解读:通过滑动窗口算法结合置换检验,鉴定出1360个转录活性区域(TARs),其中1229个对应已知蛋白编码基因,8个对应核糖体RNA基因,123个为基因间区未注释转录本。聚类分析显示蛋白编码基因的表达符合疟原虫发育阶段特异性模式,管家基因(如丝氨酰-tRNA合成酶)表达稳定,早期标记基因在18hpi高表达,晚期标记基因在36hpi高表达,验证了样本的生物学可靠性。
产品关联:实验所用关键产品:RNeasy柱(Qiagen)、Microcon YM-30离心过滤器(Millipore)、Message Amp II(Ambion)、Superscript II逆转录酶(Invitrogen)、Cy3/Cy5-dUTPs(GE Healthcare)
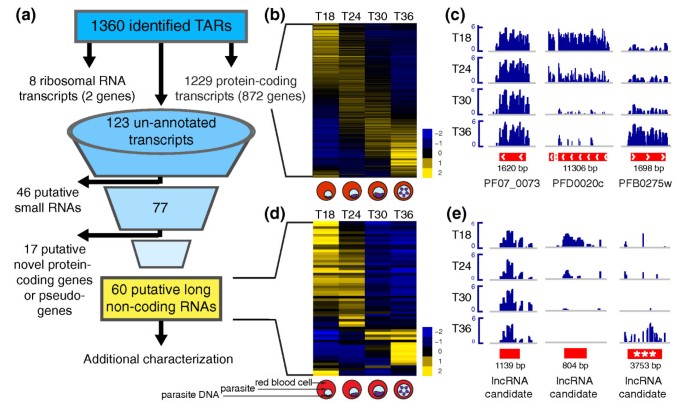
3.2 长链非编码RNA候选的筛选与初步表征
实验目的:从基因间区未注释转录本中筛选出具有功能潜力的长链非编码RNA,并优先定位高价值候选。
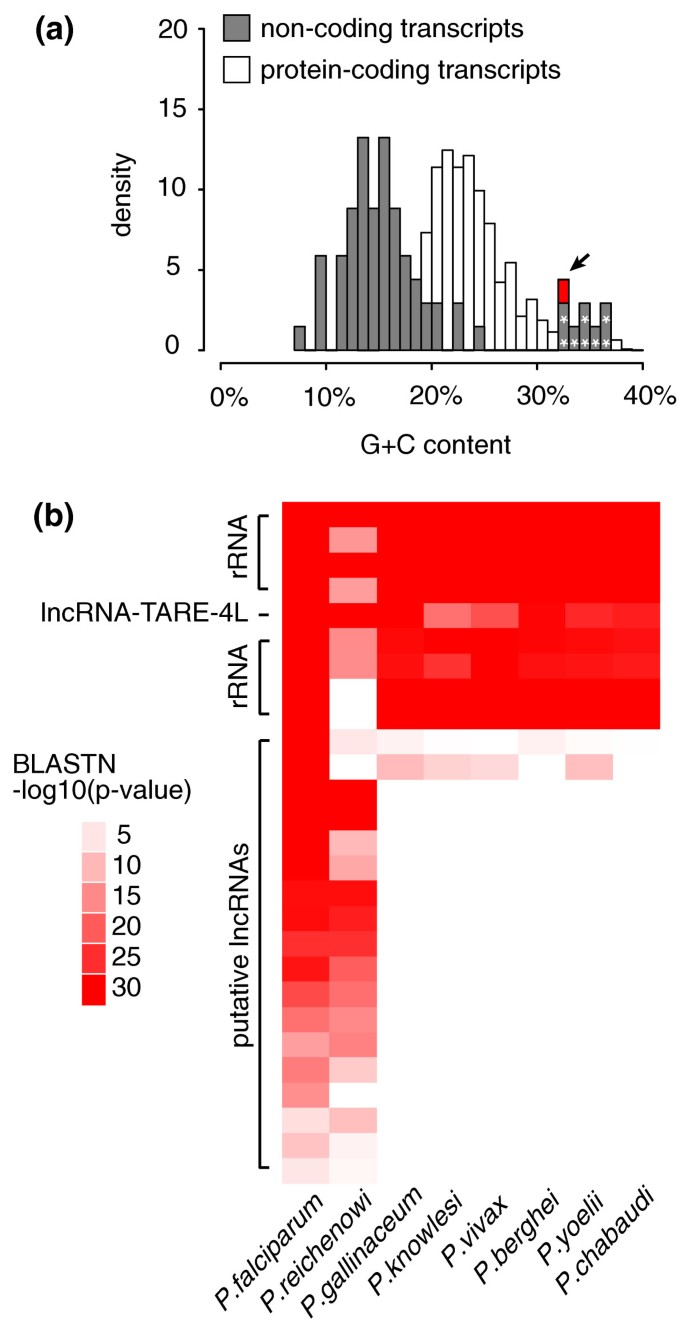
方法细节:首先设置长度阈值(>200nt)排除46个小RNA候选,再通过BLASTX分析排除17个具有蛋白编码潜力的转录本,最终得到60个长链非编码RNA候选。进一步分析候选序列的G+C含量、进化保守性、与邻近基因的表达相关性及距离:计算G+C含量并与蛋白编码基因、核糖体RNA比较;通过BLASTN分析在8种疟原虫物种中的序列保守性;计算与邻近蛋白编码基因的表达Pearson相关性,结合与编码基因的距离判断是否为独立转录本。
结果解读:60个长链非编码RNA候选的平均G+C含量为15.4%,低于蛋白编码基因的23.7%,其中lncRNA-TARE-4L的G+C含量为32.1%,与核糖体RNA相近;进化保守性分析显示19个候选在至少一种其他疟原虫物种中保守,而lncRNA-TARE-4L在所有8种测试物种中高度保守(P=0.045);相关性与距离分析显示23个候选为独立转录本,其中lncRNA-TARE-4L与邻近基因表达负相关(r=-0.296),且距离最近的var基因超过20kb,明确为独立转录本。
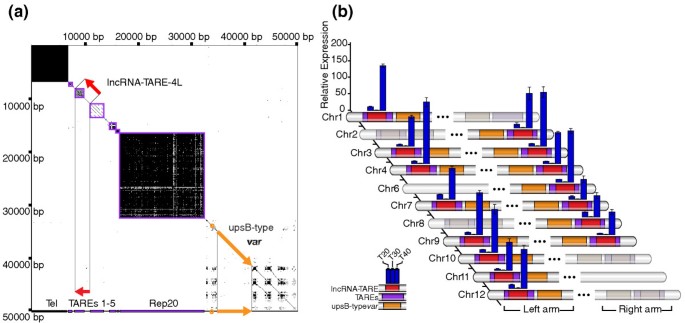
3.3 端粒相关长链非编码RNA家族的鉴定与表达模式验证
实验目的:验证lncRNA-TARE-4L的同源序列分布,并检测其在疟原虫发育阶段的表达模式。
方法细节:通过BLASTN在恶性疟原虫28条染色体的端粒相关重复元件(TARE)区域搜索同源序列,发现22条染色体端存在同源序列。针对15条染色体端的同源序列设计特异性引物,采用定量实时聚合酶链反应(qRT-PCR)检测同步化疟原虫样本(环期20hpi、滋养体期30hpi、裂殖体期40hpi)的表达水平,以泛素结合酶基因PF08_0085为内参,设置2个生物学重复。
结果解读:定量实时聚合酶链反应结果显示,15条染色体端的同源序列均在裂殖体阶段(40hpi)显著高表达,表达模式高度协调,在DNA复制高峰(30hpi)后快速上调,表明该家族在疟原虫红细胞内发育的裂殖体阶段协同发挥作用。
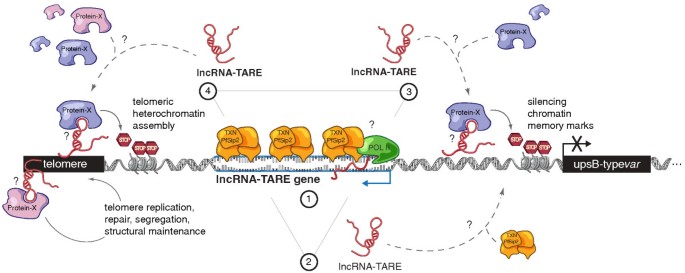
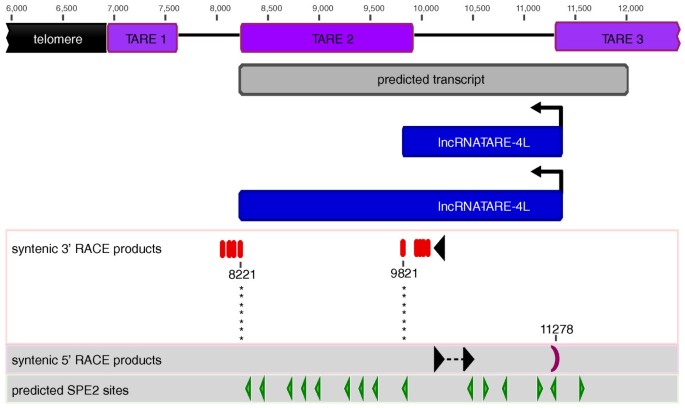
3.4 端粒相关长链非编码RNA的转录本结构与调控元件分析
实验目的:确定端粒相关长链非编码RNA的转录起始与终止位点,解析其潜在调控机制。
方法细节:采用cDNA末端快速扩增(RACE)技术,针对20个同源序列设计引物,对40hpi样本的RNA进行5"和3" RACE,克隆测序后分析转录本长度与终止位点;通过序列分析检测转录区域的转录因子结合位点,重点分析SPE2基序的分布,并通过定量实时聚合酶链反应检测其与ApiAP2转录因子PfSip2的表达相关性。
结果解读:cDNA末端快速扩增结果显示,该家族存在两种主要转录本,长度分别约为1.5kb和3.1kb,均从端粒相关重复元件3边界向端粒方向转录,终止位点分别位于端粒相关重复元件2元件之前和之后;转录区域富含SPE2基序,该基序也存在于upsB型var基因的启动子中,且lncRNA-TARE-4L与PfSip2的表达高度相关,PfSip2在lncRNA-TARE表达前上调,提示其可能参与该家族的转录调控。
4. Biomarker研究及发现成果解析
Biomarker定位
本研究发现的端粒相关长链非编码RNA家族(lncRNA-TARE)属于非编码RNA类功能生物标志物,筛选与验证逻辑为:高分辨率tiling芯片转录组鉴定→生物信息学筛选(保守性、表达模式、独立性)→定量实时聚合酶链反应验证发育阶段表达模式→cDNA末端快速扩增确定转录本结构。
研究过程详述
该生物标志物来源于恶性疟原虫3D7株的染色体端粒相关重复元件区域,验证方法包括:定量实时聚合酶链反应检测不同发育阶段的表达水平,明确其阶段特异性;cDNA末端快速扩增确定转录本的精确结构;BLASTN分析验证其在8种疟原虫物种中的序列保守性。特异性方面,lncRNA-TARE仅在裂殖体阶段高表达,所有同源序列的表达模式高度协调,且均与upsB型var基因相邻;敏感性方面,定量实时聚合酶链反应可检测到低丰度的转录本表达。
核心成果提炼
本研究首次发现恶性疟原虫中的22个端粒相关长链非编码RNA,它们在DNA复制后协同表达,推测其参与端粒维持、upsB型var基因调控及染色质重塑过程。创新性在于首次系统鉴定恶性疟原虫长链非编码RNA,并发现了一类全新的端粒相关非编码RNA家族,为疟原虫转录调控机制研究提供了新的视角。统计学结果显示,lncRNA-TARE-4L的跨物种保守性具有统计学意义(P=0.045),定量实时聚合酶链反应的表达差异在生物学重复中具有一致性(n=2,P<0.05,文献未明确具体P值)。作者进一步提出四种功能模型:PfSip2与lncRNA-TARE的相互调控、lncRNA-TARE招募PfSip2到染色体端、lncRNA-TARE参与var基因的表观沉默、lncRNA-TARE参与端粒异染色质组装,为后续功能验证研究提供了方向。