1. 领域背景与文献引入
文献英文标题:Analysis of the Saccharomyces cerevisiae proteome with PeptideAtlas;发表期刊:Genome Biology;影响因子:未公开;研究领域:酵母蛋白质组学、质谱蛋白质组学
2006年,生命科学领域中基因组学已进入成熟阶段,多物种基因组测序完成且注释工作全面推进,转录组学借助微阵列等技术可实现高灵敏度的mRNA表达检测。然而蛋白质组学研究仍处于发展瓶颈期,尽管质谱技术已成为蛋白质组分析的核心手段,但由于mRNA表达水平与蛋白质丰度存在翻译效率差异、蛋白靶向降解等不对应性,且蛋白质存在翻译后修饰、动态互作等复杂特性,蛋白质组学更能直接反映生物功能状态,但面临低丰度蛋白检测困难、数据整合难度大等问题。当时酿酒酵母作为模式生物,已有研究通过表位标签技术检测到73%的开放阅读框(ORF),或通过质谱鉴定到72%的预测蛋白,但这些研究多为单个实验,覆盖度有限且数据分散,缺乏整合多数据集的公共蛋白质组资源,难以满足大规模数据挖掘、靶向蛋白质组学研究及软件开发的需求。因此,构建整合多来源质谱数据的酿酒酵母蛋白质组公共资源,提升蛋白质组覆盖度,成为领域内亟待解决的核心问题,本研究正是针对这一需求,通过整合47个不同实验的质谱数据构建PeptideAtlas,为酵母蛋白质组学研究提供全新的工具与数据支撑。
2. 文献综述解析
作者对领域内现有研究的分类维度主要基于组学技术的发展阶段与技术类型,涵盖基因组学、转录组学与蛋白质组学三个层面。现有研究的关键结论显示,基因组学的成熟为蛋白质组学研究提供了序列基础,转录组学可高效检测mRNA表达,但无法替代蛋白质组学对生物功能的直接反映;质谱蛋白质组学技术可实现蛋白亚细胞定位、翻译后修饰、互作网络及丰度变化的检测,结合同位素标记试剂还能完成蛋白相对与绝对定量。现有技术的优势在于,表位标签技术可实现高覆盖度的蛋白表达检测,质谱技术可无标记检测多种蛋白特性;局限性则表现为蛋白质组学缺乏类似PCR的扩增策略,低丰度蛋白检测难度大,单个实验的蛋白质组覆盖度有限,且不同研究的数据分散,缺乏统一的整合平台与公共资源。通过对比现有研究的不足,本研究的创新价值凸显:首次整合47个不同来源、采用多种分离与检测技术的质谱数据集,构建了高覆盖度的酿酒酵母PeptideAtlas,不仅提升了酵母蛋白质组的观测覆盖度,还为领域提供了可用于数据挖掘、靶向蛋白质组学实验设计、蛋白质组学软件开发的公共资源,填补了多数据集整合蛋白质组资源的空白。
3. 研究思路总结与详细解析
本研究的核心目标是构建酿酒酵母PeptideAtlas整合数据库,实现多来源质谱数据的标准化处理与整合,提升酵母蛋白质组的观测覆盖度,并验证该资源在蛋白质组学研究中的应用价值;核心科学问题为如何突破单个实验的覆盖度限制,整合不同实验平台、分离技术的质谱数据,同时分析数据的观测偏倚,挖掘其应用潜力;技术路线遵循“数据收集→标准化处理→数据库构建→覆盖度与偏倚分析→应用案例验证”的闭环逻辑,确保研究的系统性与严谨性。
3.1 数据集收集与预处理
本环节的核心目标是获取多样化的酵母质谱数据集,为PeptideAtlas的构建提供丰富的数据基础。研究团队收集了47个不同来源的实验数据,所有样本均经胰蛋白酶酶解处理,部分样本额外使用同位素编码亲和标签(ICAT)或碘乙酰胺进行修饰,所有数据均采用液相色谱-电喷雾电离串联质谱(LC-ESI-MS/MS)检测,未纳入基质辅助激光解吸电离飞行时间质谱(MALDI-TOF)数据。最终共获得490万条串联质谱图,涵盖了SDS-PAGE、自由流电泳、强阳离子交换色谱等多种分离技术处理的样本,为后续的跨实验数据整合提供了多样化的基础。文献未提及具体实验产品,领域常规使用胰蛋白酶、ICAT试剂、LC-ESI-MS/MS仪器等。
3.2 质谱数据处理与数据库搜索
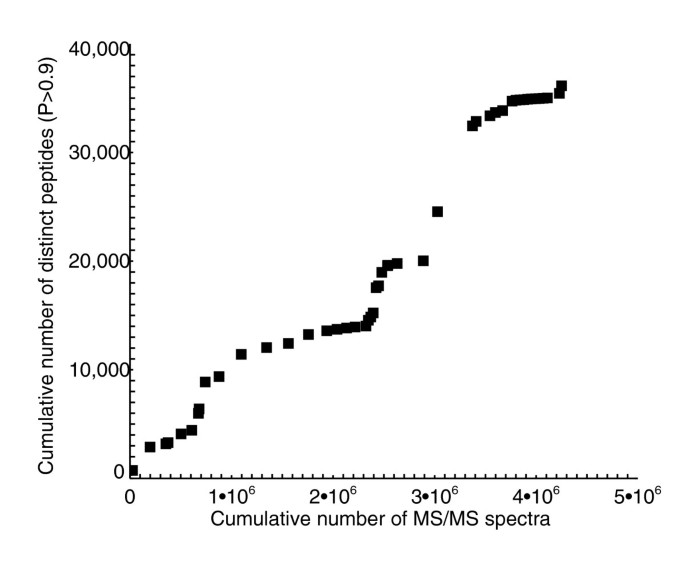
本环节旨在对原始质谱数据进行标准化处理,实现肽段与蛋白序列的精准匹配。研究人员将所有原始数据转换为mzXML统一格式,使用SEQUEST软件在非冗余酵母参考蛋白数据库(包含Saccharomyces Genome Database,SGD、Ensembl、NCI、GenBank等数据库的联合序列,同时加入角蛋白和胰蛋白酶作为污染序列)中进行搜索,随后采用PeptideProphet软件对搜索结果进行统计概率赋值,仅保留PeptideProphet概率≥0.9的肽段鉴定结果以保证数据可靠性。经处理后,共鉴定到36133条独特肽段(P>0.9),这些肽段可完美匹配到4063个SGD开放阅读框(ORF),占SGD数据库中所有ORF的61%。
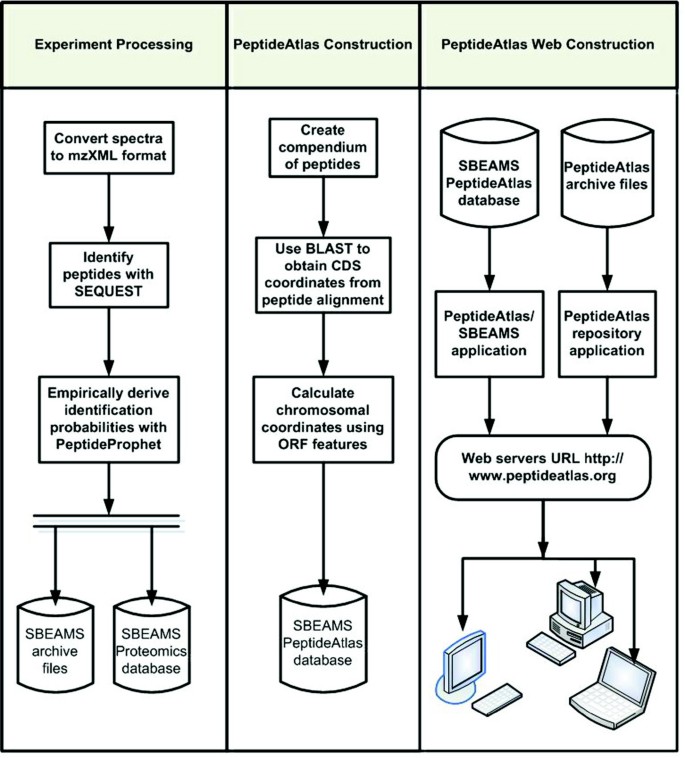
文献未提及具体实验产品,领域常规使用SEQUEST、PeptideProphet等质谱数据分析软件。
3.3 PeptideAtlas构建与覆盖度分析
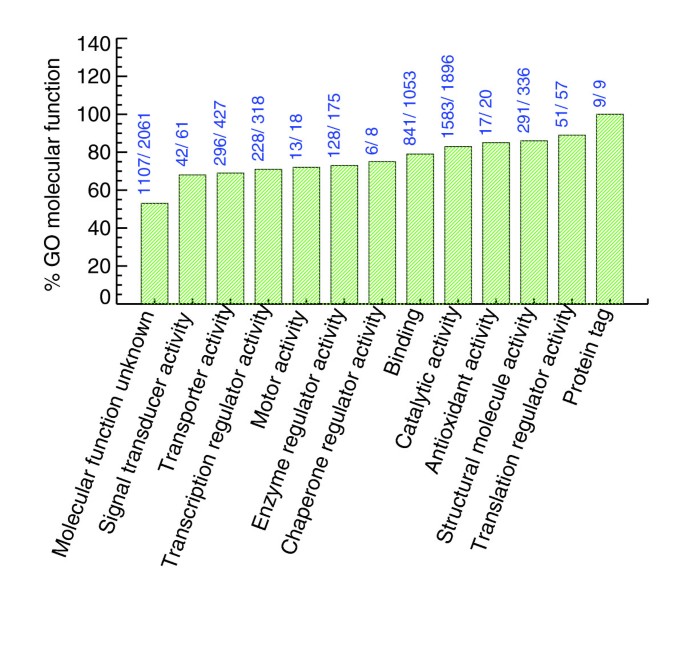
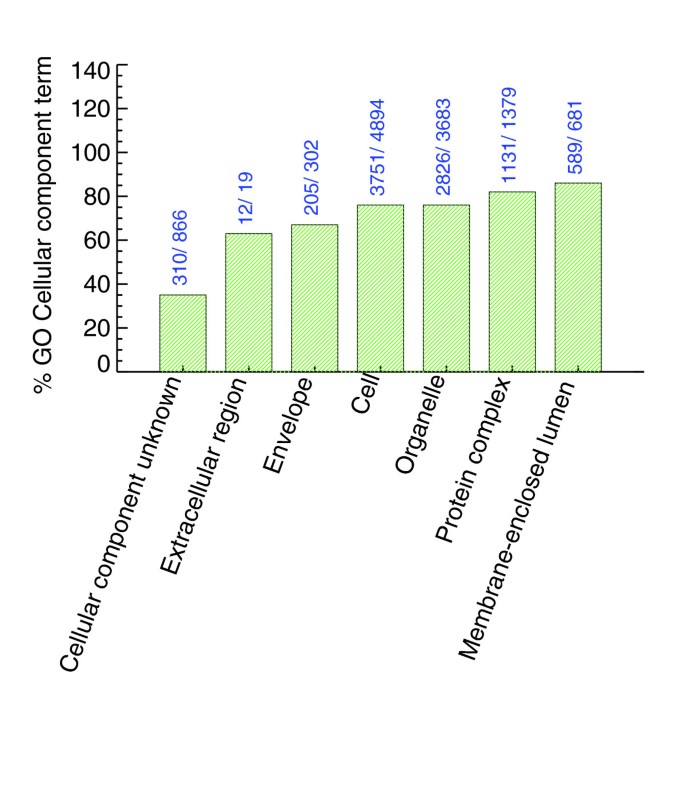
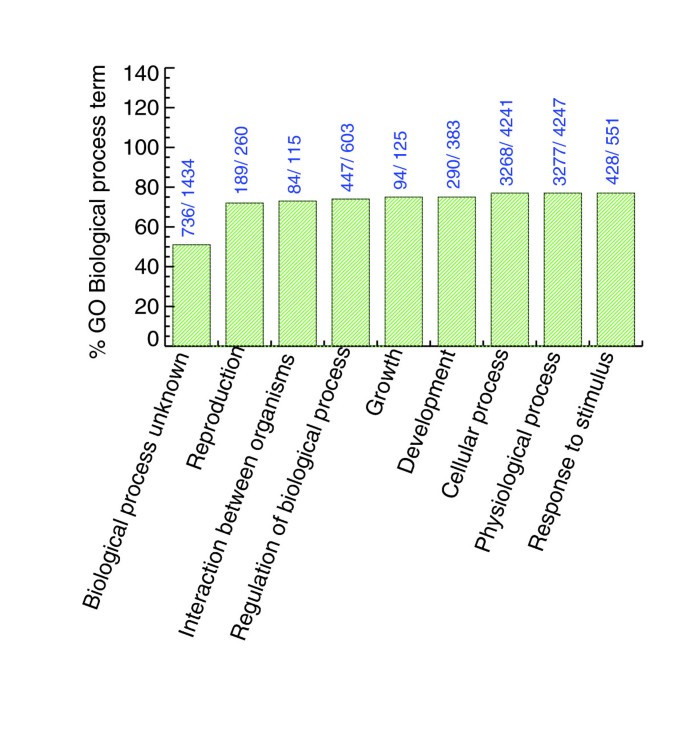
本环节的核心任务是构建整合的酵母PeptideAtlas数据库,并系统分析其对酵母蛋白质组的覆盖情况。研究人员将过滤后的肽段信息与SGD参考蛋白数据库通过BLASTP进行100%无间隙比对,结合SGD数据库的染色体坐标信息,将肽段与染色体位置关联后加载到PeptideAtlas数据库中。随后按ORF的注释类型进行覆盖度分析:已验证ORF(经实验确认存在的基因)的覆盖度为74%,未表征ORF(仅在其他物种有同源物、未在酵母中实验验证的基因)的覆盖度为49%,假基因的覆盖度为19%,转座元件相关ORF的覆盖度为20%;若去除仅观测一次的肽段以提升数据可靠性,已验证ORF的覆盖度降至56%,未表征ORF降至29%。序列覆盖度分析显示,约40%的ORF序列覆盖度超过20%。此外,研究还通过密码子富集相关性(CEC)分析验证了观测ORF的真实性,发现观测到的ORF的CEC值显著偏离随机序列,证实其为真实存在的基因;同时分析了基因本体(GO)注释的分布,观测到52%的“分子功能未知”ORF,为这些基因的功能注释提供了实验基础。
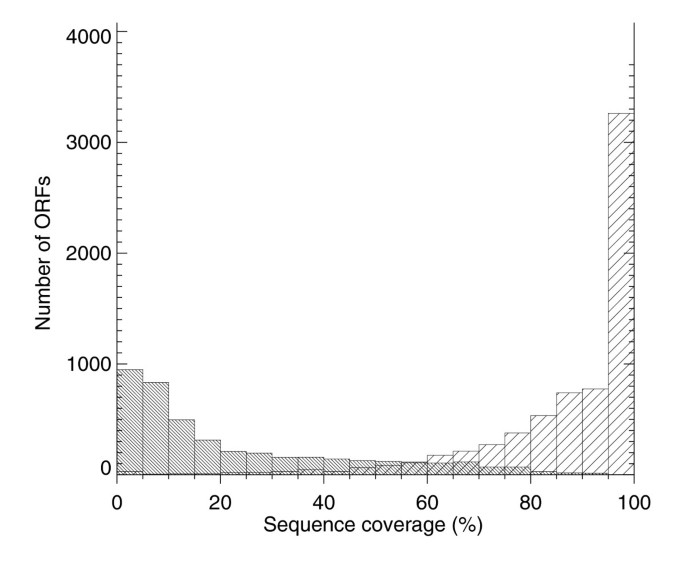
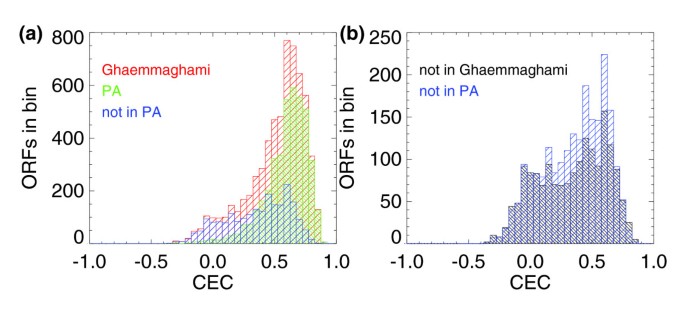
文献未提及具体实验产品,领域常规使用BLASTP等序列比对工具。
3.4 数据观测偏倚分析
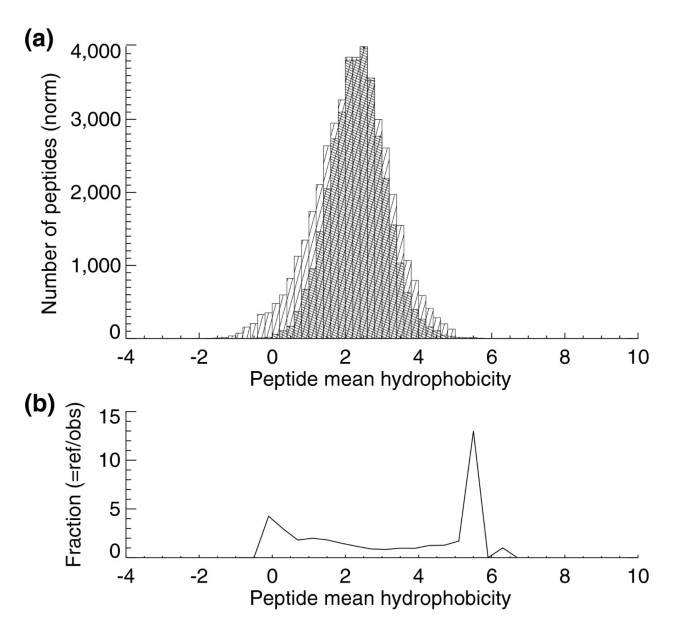
本环节旨在分析PeptideAtlas中肽段的观测偏倚,明确现有质谱技术的局限性。研究从肽段疏水性、质量、电荷分布三个维度,将观测到的肽段与理论胰蛋白酶酶解肽段进行对比:疏水性分析显示,疏水性适中的肽段观测效率最高,极端亲水的肽段因难以结合HPLC柱而被洗脱浪费,极端疏水的肽段因难以在标准LC梯度下洗脱而观测不足;质量分布分析显示,质量小于1400 Da的肽段观测不足,主要原因是短肽在数据库搜索中更难鉴定,且短肽的蛋白特异性较低;电荷分布分析显示,+2和+3电荷状态的肽段占比最高,+1电荷的肽段占比极低,符合LC-ESI-MS/MS仪器的电离特性。这些偏倚分析为后续提升蛋白质组覆盖度提供了方向,比如优化HPLC溶剂条件或采用其他分离技术检测极端亲水/疏水肽段,引入MALDI-TOF数据补充+1电荷肽段的鉴定。
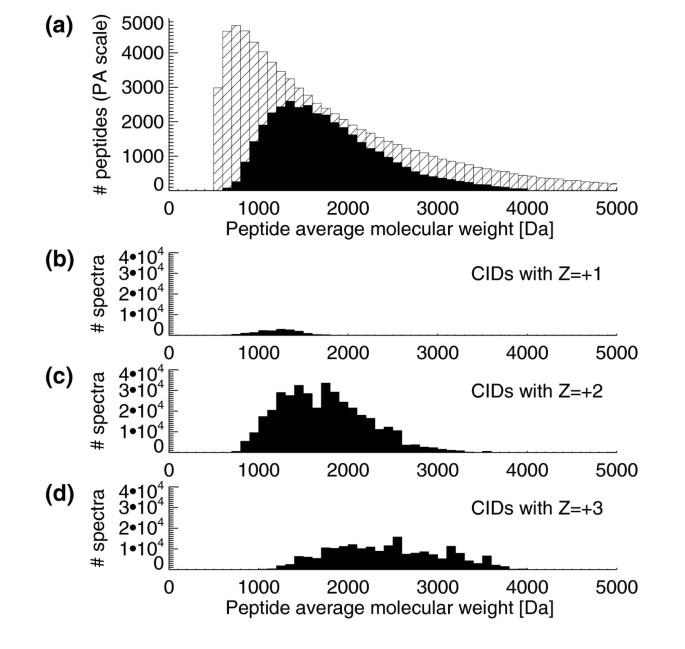
文献未提及具体实验产品,领域常规使用疏水性预测工具、质谱数据分析软件等。
3.5 PeptideAtlas应用案例验证
本环节通过两个典型案例验证了PeptideAtlas的应用价值:第一个案例是为靶向蛋白质组学筛选高置信度的肽段,以通用转录因子TFIIF复合物为例,通过PeptideAtlas的搜索功能,筛选出5条观测次数≥1、PeptideProphet概率≥0.9、仅匹配单个蛋白且含半胱氨酸的肽段,可用于合成靶向蛋白质组学的参考分子;第二个案例是验证SGD数据库预测的内含子,通过搜索跨外显子的肽段,成功验证了13%的SGD酵母ORF内含子,其中包括2个未表征ORF的内含子,为基因注释提供了实验证据。这些案例证实PeptideAtlas可广泛应用于靶向蛋白质组学实验设计、基因注释验证等研究场景。
文献未提及具体实验产品,领域常规使用PeptideAtlas在线工具、Ensembl基因组浏览器等。
4. Biomarker研究及发现成果解析
Biomarker定位
本研究中的生物标志物为用于验证基因存在、辅助基因功能注释的肽段标志物,其筛选与验证逻辑为:基于47个实验的串联质谱数据,经SEQUEST数据库搜索与PeptideProphet概率过滤(P≥0.9)获得高置信度肽段,再通过与SGD参考蛋白数据库的100%无间隙比对,确定肽段匹配的ORF,从而为ORF的真实存在提供实验证据。
研究过程详述
这些肽段标志物来源于酿酒酵母样本的胰蛋白酶酶解产物,验证方法为串联质谱鉴定+数据库搜索+统计概率过滤,特异性表现为部分肽段可唯一匹配到单个ORF,避免了肽段的冗余映射;敏感性方面,整体匹配到61%的SGD ORF,其中已验证ORF的敏感性为74%,未表征ORF的敏感性为49%,为大量未注释基因的存在提供了直接证据。
核心成果提炼
这些肽段标志物的功能关联在于,为未表征ORF的真实存在提供了实验依据,为后续基因功能注释奠定了基础;同时为靶向蛋白质组学研究提供了高置信度的肽段列表,可用于合成参考分子实现精准定量。其创新性在于,首次通过整合多数据集构建了高覆盖度的酵母肽段标志物数据库,突破了单个实验的覆盖度限制,为蛋白质组学研究提供了全新的公共资源。统计学结果显示,PeptideAtlas的预期错误率为9%,共鉴定到36133条独特肽段(P>0.9),匹配到4063个SGD ORF(占所有SGD ORF的61%);去除仅观测一次的肽段后,仍可匹配到43%的SGD ORF,充分证实了数据的可靠性与覆盖度优势。
