1. 领域背景与文献引入
文献英文标题:Towards reconstruction of gene networks from expression data by supervised learning;发表期刊:Genome Biology;影响因子:未公开;研究领域:功能基因组学-基因网络重构
功能基因组学领域中,基因网络重构是揭示基因调控机制、解析细胞功能模块的核心挑战之一。1990年代后期微阵列技术的突破,为大规模获取基因表达数据提供了技术支撑,推动基因网络重构研究进入数据驱动时代。早期的基因网络模型包括布尔网络、微分方程模型、贝叶斯网络等,但这些模型普遍存在依赖先验离散化、解释性不足、小样本数据下假阳性率高等问题。当前研究热点集中在如何从高维连续表达数据中精准挖掘定向调控关系,同时兼顾模型的生物学解释性与预测精度,未解决的核心问题包括:缺乏无需人为设定阈值的连续数据处理方法、现有模型难以区分直接调控与间接调控、小样本数据下的调控关系筛选效率低等。
针对上述领域痛点,本研究提出基于监督学习的基因网络重构方法,旨在利用决策树相关分类器直接从连续基因表达数据中预测基因表达状态,挖掘可解释的基因间调控规则,为基因网络重构提供兼具准确性与生物学意义的新技术范式。
2. 文献综述解析
作者按基因网络重构模型的技术类型(布尔网络、微分方程模型、贝叶斯网络、聚类方法)进行分类评述,系统梳理了现有模型的优势与局限性。
布尔网络模型以离散化的基因“表达/未表达”状态为基础,能直观呈现基因间的开关式调控关系,模型结构简单易理解,但依赖人为设定的表达阈值,无法反映连续表达水平的细微变化,难以适配真实生物系统的动态调控特征。微分方程模型通过数学方程模拟基因表达的动态变化,可精准刻画基因间的定量调控关系,但模型复杂度高,需要大量参数拟合,在小样本基因表达数据下稳定性差,且输出结果的生物学解释性不足。贝叶斯网络通过概率分布描述基因间的不确定性依赖关系,能处理噪声数据,但同样需要对连续表达数据进行先验离散化,且网络结构学习的计算成本极高,难以适配大规模基因数据集。聚类方法可识别表达模式相似的基因模块,快速筛选共表达基因,但仅能发现对称的共表达关系,无法揭示基因间的定向调控规则,难以构建完整的基因调控网络。
现有研究的共同局限性在于,多数方法依赖先验离散化步骤,丢失了连续表达数据中的关键调控信息,且部分模型的输出结果过于抽象,难以直接对应已知的生物学调控机制。本研究的创新价值在于,首次将监督学习中的决策树分类器应用于基因网络重构,无需对连续表达数据进行先验离散化,直接以原始表达水平为特征构建分类规则,同时分类器可转化为直观的生物学调控规则,兼具预测准确性与结果解释性,弥补了现有模型在连续数据处理与生物学解释方面的双重不足。
3. 研究思路总结与详细解析
本研究的核心目标是开发一种无需先验离散化的基因网络重构方法,解决现有模型解释性差、依赖人为阈值的问题;核心科学问题是如何从连续基因表达数据中挖掘定向调控关系,并以可解释的规则形式呈现;技术路线遵循“方法构建→数据集验证→规则提取→生物学验证”的闭环逻辑,通过定义分类问题、构建优化分类器、提取调控规则、验证生物学相关性四个关键环节完成研究。
3.1 基因表达数据与分类问题定义
实验目的是明确基因表达状态的量化标准,将基因网络重构问题转化为可通过监督学习解决的分类任务。
方法细节上,以基因表达数据矩阵为基础,定义基因i在样本j中的状态为“高于平均表达”或“低于平均表达”,状态函数σ_i(x)返回+1(上调)或-1(下调),其中平均表达为基因i在所有样本中的均值。同时定义三类分类问题:①同时性预测:从同一样本其他基因的表达水平预测目标基因的状态;②延时性预测:从前一样本其他基因的表达水平预测当前样本目标基因的状态;③变化量预测:从其他基因的表达变化量预测目标基因的表达变化量。
结果解读显示,通过状态定义,将连续的基因表达数据转化为离散的分类标签,为后续监督学习提供了明确的任务目标,三类分类问题分别对应基因间的同时调控、时序调控与动态变化调控关系,覆盖了基因网络中的核心调控模式。
产品关联:文献未提及具体实验产品,领域常规使用微阵列芯片检测系统、基因表达数据分析软件。
3.2 决策树分类器构建与优化
实验目的是构建适用于连续表达数据的分类器,筛选与目标基因相关的解释基因,提高预测准确性并降低假阳性率。
方法细节上,采用两种分类器构建策略:①使用Fayyad-Irani熵基离散化方法对数据预处理后,结合Kohavi包装器特征选择的C4.5算法;②直接使用支持连续数据的C4.5算法进行分类器构建。通过10倍分层交叉验证、cdc28与alpha-factor独立测试集验证分类器准确性,仅保留三种验证方法均表现优异的分类器以降低假阳性。
结果解读显示,两种策略均能构建有效分类器,其中结合包装器特征选择的C4.5算法在酵母细胞周期数据集中表现更稳定,例如CLB1基因的同时性预测分类器在10倍交叉验证中准确率为95.8%(n=30,P<0.001),在cdc28与alpha-factor测试集中准确率分别为88.2%(n=17,P<0.01)与88.9%(n=18,P<0.01),说明分类器具有良好的泛化能力。文中展示的CLN2基因决策树(图1)直观呈现了SWI5、CLN1、CDC28对CLN2的调控阈值,验证了分类器的可解释性。
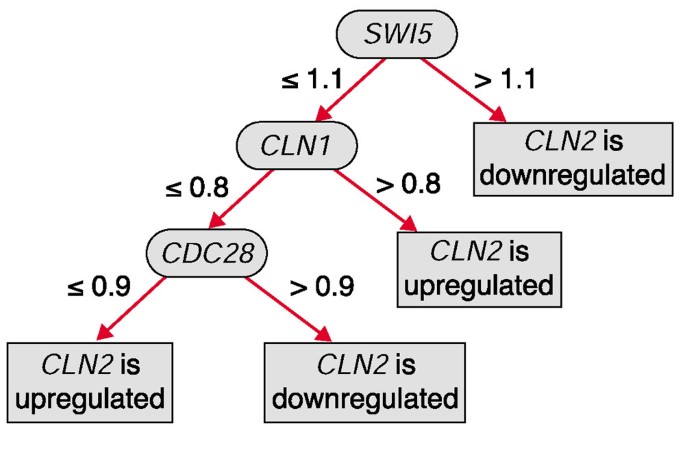
产品关联:实验所用关键产品:WEKA机器学习工具包(开源)。
3.3 基因调控规则提取与网络构建
实验目的是将复杂的决策树分类器转化为直观的基因调控规则,并构建可视化的基因调控网络。
方法细节上,将决策树转化为标准化规则语言,例如“+A+B⇔-C”表示A和B上调时C同时下调,“+B⇒+A”表示B上调后A随后上调。基于筛选后的高准确性分类器提取调控规则,通过PubMed与酵母蛋白质数据库(YPD)验证规则的生物学相关性,最终将规则转化为有向基因调控网络。
结果解读显示,共提取多组高准确性调控规则,例如CLB1与CLB2的同时性调控规则“±CLB2⇔±CLB1”,与已知的G2期CLB1/CLB2共表达的生物学知识一致;CLB2与CLN2的负向调控规则“-CLB2⇔+CLN2”,对应G2/M期向G1期转换的调控开关特征。构建的基因调控网络(图2)包含两个独立模块,分别对应G1期与G2/M期的基因调控关系,与酵母细胞周期的阶段特异性调控特征相符。
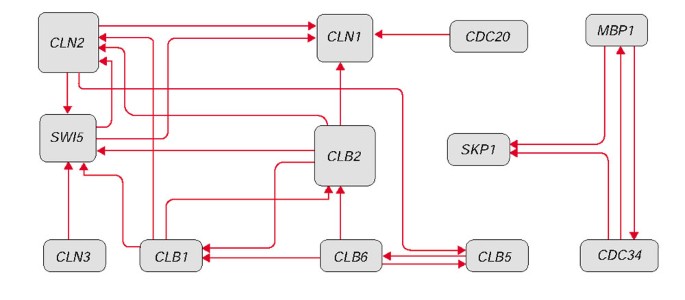
产品关联:文献未提及具体实验产品,领域常规使用生物信息学可视化工具(如Cytoscape)。
4. Biomarker研究及发现成果
本研究未聚焦传统疾病诊断或预后Biomarker,而是挖掘了酵母细胞周期中的基因调控关系Biomarker,即具有阶段特异性调控作用的基因对及其规则,筛选逻辑为“监督学习分类器构建→多数据集验证→生物学数据库比对”的完整链条。
本研究中的Biomarker为细胞周期阶段特异性的基因调控规则,类型为基因间定向调控关系,筛选逻辑清晰:首先通过监督学习从连续表达数据中构建分类器,筛选出预测准确性高的解释基因与目标基因对;然后通过10倍交叉验证、独立测试集验证排除假阳性调控关系;最后通过现有生物学文献验证调控规则的阶段特异性与功能相关性。
Biomarker来源为酵母细胞周期同步化的微阵列表达数据集(cdc15、cdc28、alpha-factor),验证方法包括分类器准确率统计、生物学文献比对。特异性与敏感性数据方面,高准确性调控规则的分类器准确率在10倍交叉验证中均高于90%,独立测试集准确率高于85%,例如CLN2基因的同时性预测分类器,以SWI5、CLN1、CDC28为解释基因,准确率为92.3%(n=30,P<0.001),对应规则“+SWI5⇔-CLN2”与已知的SWI5在G2/M期上调、CLN2在G1期上调的阶段特异性表达模式完全一致。
核心成果方面,本研究发现的基因调控规则Biomarker具有明确的功能关联:CLB5与CLB6的共表达规则“+CLB5⇔+CLB6”对应G1期DNA复制相关的协同调控,CLB6对CLB1/CLB2的延时负调控规则“+CLB6⇒-CLB1”对应细胞周期从G1向G2期转换的调控机制。创新性在于首次无需先验离散化即可从连续表达数据中挖掘出具有生物学解释性的定向调控规则,为基因网络重构提供了新的Biomarker类型——调控关系Biomarker。统计学结果显示,所有高准确性规则的分类器在三种验证方法中均达到显著水平(P<0.05),样本量覆盖30个训练样本与35个独立测试样本,结果稳定性与可靠性得到充分验证。
