1. 领域背景与文献引入
文献英文标题:Spatio-temporal regulation of circular RNA expression during porcine embryonic brain development;发表期刊:Genome Biology;影响因子:10.806(2015年);研究领域:环状RNA与哺乳动物胚胎脑发育。
环状RNA(circRNA)是近年来RNA生物学的研究热点,已在人、小鼠、果蝇等物种中发现数千种环状RNA,其通过miRNA海绵、剪接调控等机制参与基因表达。然而,哺乳动物胚胎脑发育过程中环状RNA的时空表达模式尚未被系统研究——现有研究多聚焦于成体组织或细胞系,且缺乏大型动物(如猪)模型的分析。猪的脑结构(如回旋脑)与人高度相似,是研究脑发育的理想模型,但此前未涉及环状RNA的研究。本研究针对这一空白,首次分析猪胚胎脑5个区域、6个发育阶段的环状RNA表达,旨在揭示其时空调控规律及潜在功能,为理解环状RNA在哺乳动物脑发育中的作用提供大型动物证据。
2. 文献综述解析
作者对现有研究的评述围绕“环状RNA的功能发现”“脑发育的非编码RNA调控”“现有研究的局限性”展开:
- 现有研究结论:环状RNA广泛存在于多物种,具有组织/细胞特异性,可通过结合miRNA(如CiRS-7作为miR-7海绵)或蛋白质(如MBL1环状RNA调控剪接)发挥功能;脑发育中,microRNA和长链非编码RNA的作用已明确,但环状RNA的角色未知。
- 现有研究局限性:① 未涉及哺乳动物胚胎脑发育的时空表达分析;② 缺乏大型动物模型的验证;③ 未解析环状RNA的亚细胞定位动态。
- 本研究创新点:① 首次以猪(大型动物)为模型,系统分析胚胎脑环状RNA的时空表达;② 结合猪、小鼠、人的保守性分析,筛选功能重要的环状RNA;③ 利用RNase R+原位杂交技术,首次展示环状RNA在发育中的亚细胞定位变化。
3. 研究思路总结与详细解析
整体框架:研究目标是解析猪胚胎脑环状RNA的时空表达、保守性及亚细胞定位;核心科学问题包括:① 环状RNA是否具有时空特异性?② 是否在哺乳动物中保守?③ 亚细胞定位如何动态变化?技术路线:样本收集→rRNA depletion测序→环状RNA鉴定→保守性分析→表达聚类→热点基因验证→原位杂交。
3.1 胚胎猪脑样本收集与测序
实验目的:获取覆盖时空的转录组数据,为环状RNA分析奠定基础。
方法细节:收集猪胚胎5个脑区(皮层、基底神经节、脑干、小脑、海马)、6个时间点(E23至E115)的样本;提取总RNA后,用Epicentre的Ribo-Zero Magnetic Kit去除核糖体RNA,ScriptSeq v2 kit构建文库,Illumina 2×100 nt测序。
结果解读:每个样本获得≥2100万条raw reads,rRNA depletion确保了环状RNA(非poly(A)尾)的有效检测。
产品关联:实验所用关键产品:Epicentre的Ribo-Zero Magnetic Kit、ScriptSeq v2 kit;Illumina测序平台。
3.2 环状RNA鉴定与表达分析
实验目的:鉴定环状RNA并分析其表达模式。
方法细节:使用Memczak pipeline鉴定环状RNA,提升过滤标准(环化外显子两端映射质量≥40)以去除误注释的线性RNA;用“回环reads数/百万raw reads(RPM)”归一化,设置RPM≥0.05为表达阈值。
结果解读:共鉴定到5585个环状RNA,其中皮层E60时表达最丰富(4634个),E80后表达骤降(仅1200个);环状RNA的表达具有严格时空特异性(如皮层E60的环状RNA数量是E115的7倍)。
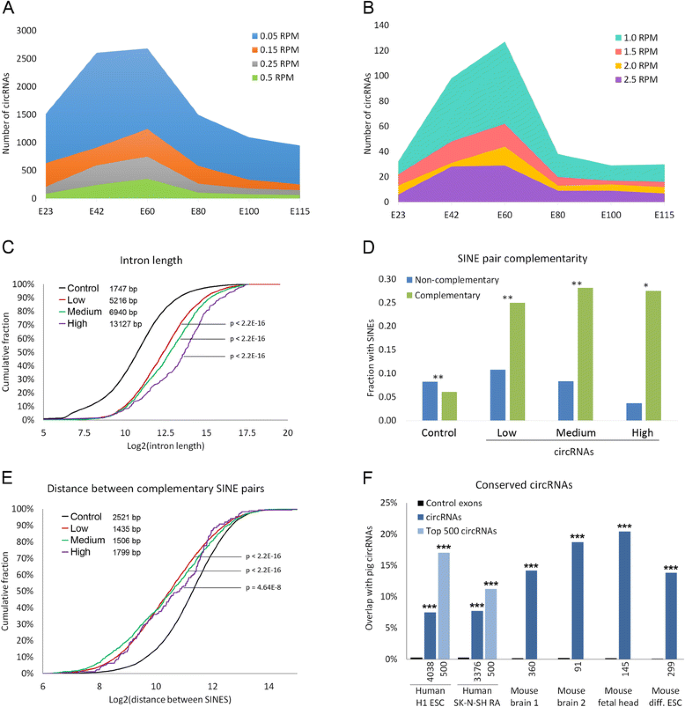
3.3 环状RNA保守性分析
实验目的:研究猪环状RNA与人和小鼠的功能保守性。
方法细节:用UCSC liftOver工具将猪环状RNA的剪接位点映射到小鼠(mm10)和人(hg19)基因组;比较猪与小鼠胎儿头部、人类神经母细胞瘤细胞(SK-N-SH)的环状RNA重叠情况;生成随机对照环状RNA评估显著性。
结果解读:88%的猪环状RNA剪接区域能映射到小鼠基因组,20.4%的小鼠胎儿头部环状RNA剪接位点与猪完全一致(P<10^-10,显著高于随机对照);人类高表达环状RNA(前500个)与猪的重叠率更高,说明高表达环状RNA更保守。
3.4 表达模式聚类与通路分析
实验目的:分析环状RNA的表达模式及功能关联。
方法细节:对top 200高表达环状RNA进行皮尔逊相关聚类;用DAVID工具对E60高表达的环状RNA宿主基因进行KEGG通路分析(背景为所有表达基因)。
结果解读:聚类得到6个表达组(如早期高表达、E60峰值、晚期高表达);通路分析显示,E60高表达的环状RNA宿主基因显著富集于Wnt信号通路(P<0.01)、轴突导向(P<0.05),提示环状RNA可能参与神经发生和神经元迁移。
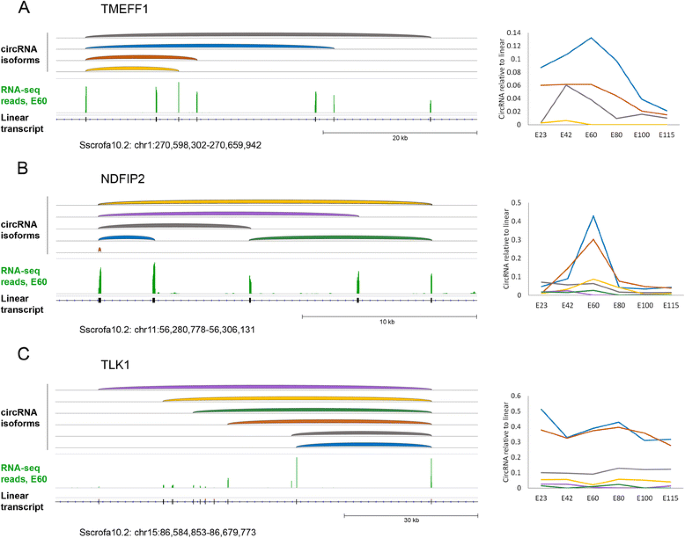
3.5 热点基因与多isoform验证
实验目的:验证“热点基因”(产生多个环状RNA isoform)的存在及表达差异。
方法细节:选择TMEFF1、NDFIP2、TLK1三个热点基因,设计发散引物进行RT-PCR;对产物测序验证isoform序列;分析不同isoform的RPM表达模式。
结果解读:每个基因产生2-3个isoform,且表达模式不同(如TMEFF1的一个isoform在E60峰值,另一个在E42峰值);RT-PCR验证了这些isoform的存在,说明环状RNA的生成具有转录后调控特异性。
产品关联:实验所用关键产品:Invitrogen的M-MLV Reverse Transcriptase、Taq DNA polymerase。
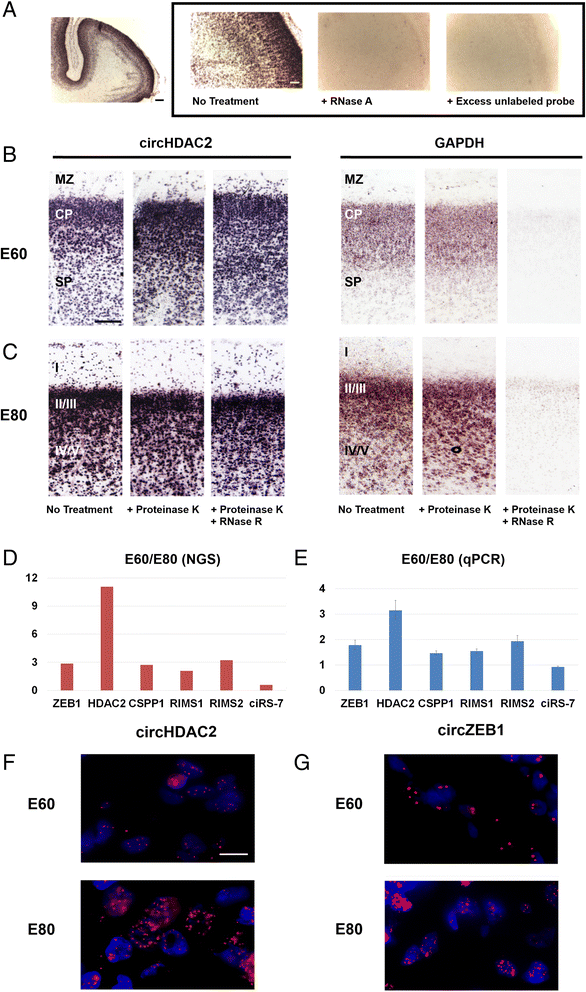
3.6 原位杂交与亚细胞定位
实验目的:验证环状RNA的组织定位及亚细胞分布动态。
方法细节:选择circHDAC2(高表达)、circZEB1(动态表达),设计跨越回环 junction的LNA探针;对E60和E80的脑切片进行原位杂交,结合RNase R处理(去除线性RNA)验证环形结构;用panomics探针进行高分辨率杂交,DAPI染色显示细胞核。
结果解读:circHDAC2在E60和E80均定位于细胞核;circZEB1在E60时分布于胞质和核,E80时仅定位于核,说明亚细胞定位随发育动态变化;RNase R处理后信号未消失,证实目标RNA为环状。
产品关联:实验所用关键产品:DNA Technology A/S的LNA探针、Affymetrix的panomics探针。
4. Biomarker研究及发现成果解析
Biomarker定位:本研究中的Biomarker为“发育阶段/区域特异性环状RNA”,包括circCSPP1、circHDAC2、CiRS-7。筛选逻辑:通过测序筛选高表达、时空特异性的环状RNA→qPCR/northern blot验证表达→原位杂交确认定位。
研究过程详述
- Biomarker来源:猪胚胎脑组织样本。
- 验证方法:① qPCR检测回环 junction的表达(如circCSPP1在E60皮层的表达是线性转录本的3.7倍,n=3,P<0.01);② northern blot验证RNase R抗性(环状RNA特征);③ 原位杂交确认组织分布(如circHDAC2在皮层E60高表达)。
- 特异性与敏感性:circCSPP1在皮层E60的RPM值(12.3)显著高于其他时间点(E42为2.1,E80为1.5,n=3,P<0.001),具有高阶段特异性;CiRS-7在E115小脑的RPM值(19)是其他组织的5-10倍,具有高组织特异性。
核心成果提炼
这些环状RNA可作为猪胚胎脑发育的阶段/区域标志物:① circCSPP1是皮层E60(神经发生高峰期)的标志物;② circHDAC2是皮层E60-E80(神经元迁移期)的标志物;③ CiRS-7是小脑E115(出生前)的标志物。创新性在于首次报道环状RNA作为哺乳动物胚胎脑发育的分子标志物,为脑发育的分子分期提供新靶点。
本研究通过系统解析猪胚胎脑环状RNA的时空表达、保守性及定位,为环状RNA在脑发育中的功能研究奠定了基础,也为大型动物模型的环状RNA研究提供了方法学参考。
