1. 领域背景与文献引入
文献英文标题:A pan-Zea genome map for enhancing maize improvement;发表期刊:Genome Biology;影响因子:13.583;研究领域:玉米基因组学与分子育种。
玉米是全球最重要的粮食、饲料和生物燃料作物,但其育种面临单参考基因组局限性和野生遗传资源利用不足的挑战。传统基因组辅助育种依赖B73等单一参考基因组,但单参考基因组无法覆盖玉米野生近缘种(如teosinte)的遗传多样性,导致“参考偏差”——遗漏非参考序列中的抗逆、优质基因。泛基因组作为物种完整遗传信息的代表,能补充单参考基因组的不足,已在水稻、小麦等作物中应用,但玉米泛基因组研究多集中在栽培种(如NAM群体),缺乏对Zea属全范围(包含栽培玉米、地方品种和野生teosinte)的整合;同时,结构变异(SV)(如插入、缺失、易位)的遗传效应未充分解析——现有研究多关注SNP/InDel,而SV占基因组变异的比例更高,且常与复杂性状关联。
现有研究空白包括:1)缺乏包含Zea属所有主要类群的泛基因组;2)未系统分析基因存在/缺失变异(gPAV)和结构变异在玉米性状中的作用。本研究旨在构建泛Zea基因组,解析gPAV和SV的遗传模式,探索其在玉米农艺性状中的关联,为玉米分子育种提供新资源。
2. 文献综述解析
作者对现有研究的评述逻辑按两个核心维度展开:一是泛基因组构建的物种范围(仅栽培种vs整合野生种),二是遗传变异类型的覆盖度(SNP/InDel vs SV/gPAV)。
现有研究的关键结论与局限性
- 关键结论:单参考基因组会遗漏非参考序列的重要基因;泛基因组能补充非参考序列,解析栽培种与野生种的遗传差异;SNP/InDel是主要遗传标记,但SV/gPAV也能影响复杂性状(如产量、抗逆)。
- 技术优势:之前的玉米泛基因组研究(如NAM群体)用转录组或栽培种基因组,解析了基因表达多样性;但局限性明显——未整合Zea属野生种,无法代表属级遗传多样性;且SV检测多基于短读长测序,遗漏长片段变异,未系统关联性状。
本研究的创新价值
- 首次构建包含721个Zea属个体(507栽培玉米、31地方品种、183 teosinte)的泛基因组,整合11个染色体级组装,覆盖属内所有主要类群;
- 系统检测结构变异(SV),并结合多组学性状(农艺、表达、代谢、甲基化)分析gPAV和SV的遗传效应,弥补了现有研究在物种范围和遗传变异类型上的不足。
3. 研究思路总结与详细解析
本研究以“构建泛Zea基因组→解析gPAV和SV模式→关联遗传变异与多组学性状”为核心逻辑,分4个关键步骤展开:
3.1 泛Zea基因组构建与特征分析
实验目的:构建包含Zea属全范围的泛基因组,解析其结构特征。
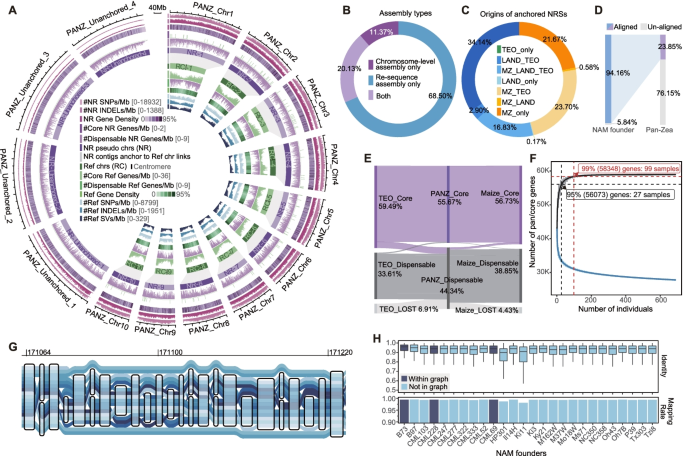
方法细节:收集721个个体的全基因组测序数据(栽培玉米、地方品种、teosinte),用IDBA-UD进行从头组装;将组装结果与B73参考基因组(AGPv4)比对,整合11个已发表的染色体级组装(如SK、Mo17),通过序列聚类和锚定构建泛基因组。
结果解读:泛Zea基因组大小约6.71 Gb,其中B73参考序列占2.14 Gb(31.83%),非B73参考序列(NRS)占4.57 Gb(68.17%);超过一半的NRS(58.86%)锚定到B73染色体上,其中34.14%的锚定NRS仅存在于teosinte中,提示野生种贡献了大量独特遗传信息。与之前的NAM群体泛基因组相比,本研究的NRS包含98.76%的NAM非参考序列,且新增69.52%的独特序列(图1)。
产品关联:文献未提及具体实验产品,领域常规使用从头组装软件(IDBA-UD)、序列比对工具(Minimap2)、泛基因组构建工具(EUPAN)。
3.2 泛Zea基因注释与gPAV分析
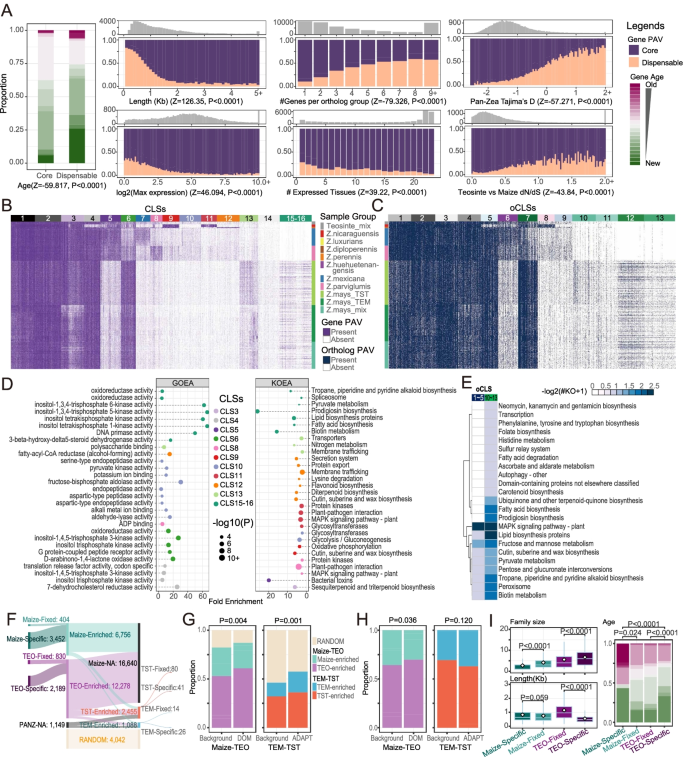
实验目的:注释泛基因组的基因结构,解析基因存在/缺失变异的遗传模式。
方法细节:整合B73参考基因注释、非参考序列的转录组证据(RNA-seq)、同源蛋白比对(UniProt)和从头预测(Augustus),生成泛Zea基因集;用read-mapping方法(BWA-MEM)检测每个个体的基因存在状态,计算gPAV矩阵。
结果解读:共注释58,944个泛Zea基因(39,591个来自B73,19,353个来自非参考序列),其中85.82%的基因有功能注释(GO、KEGG)。gPAV分析显示:
- 44.34%的基因为dispensable基因(群体缺失率>1%),多为年轻基因、组织特异性表达基因或大基因家族成员;
- core基因(缺失率≤1%)多为保守的管家基因(如核糖体蛋白基因);
- teosinte富集的基因多与抗逆相关(如植物-病原体互作、黄酮类合成),玉米富集的基因多与营养和风味相关(如氨基酸合成、类胡萝卜素代谢)(图2)。
产品关联:文献未提及具体实验产品,领域常规使用基因注释工具(EVidenceModeler)、功能注释工具(InterProScan)、gPAV检测工具(PopIns)。
3.3 玉米群体结构变异检测与分型
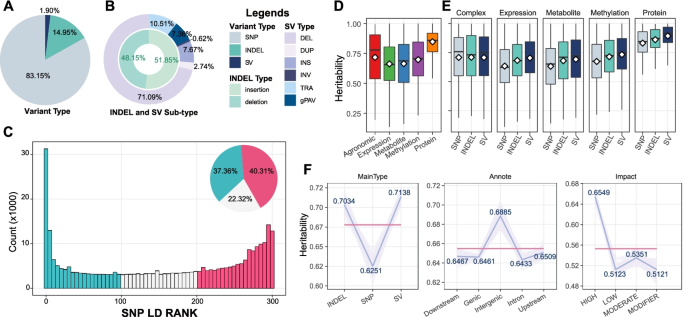
实验目的:检测玉米群体中的常见结构变异,分析其遗传特征。
方法细节:结合三种证据检测SV:1)全基因组比对(MUMmer);2)短读长测序的split-read和paired-end方法(Manta、GRIDSS);3)图形基因组分型(VG)。筛选群体频率>5%的常见SV,进行基因分型。
结果解读:共检测到274,649个常见SV,包括181,874个缺失(DEL)、19,628个插入(INS)、26,894个易位(TRA)等,其中60.03%的SV与转座子(TE)相关(如LTR、helitron)。与SNP/InDel相比,SV的遗传力更高(在多组学性状中平均遗传力0.71,高于SNP的0.63),且37.36%的SV与附近SNP的连锁不平衡(LD)较低,提示SV包含独立的遗传信息(图3)。
产品关联:文献未提及具体实验产品,领域常规使用SV检测工具(Manta、GRIDSS)、转座子分析工具(RepeatMasker)、图形基因组工具(VG)。
3.4 遗传变异与多组学性状关联分析
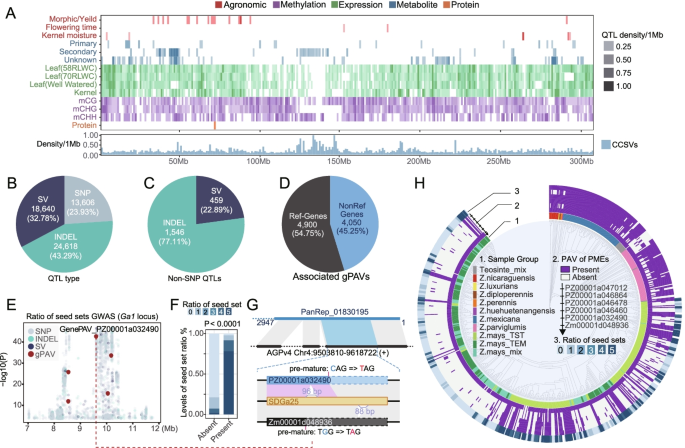
实验目的:分析SNP、InDel、SV和gPAV与玉米农艺性状及分子性状的关联。
方法细节:收集玉米关联群体的多组学数据(农艺性状:产量、耐旱性;分子性状:基因表达、代谢物、DNA甲基化);用GWAS工具(TASSEL)检测遗传变异与性状的关联,通过贝叶斯精细定位(FINEMAP)筛选候选因果变异(CCV)。
结果解读:共鉴定到21,255个QTL,其中32.78%的QTL由SV主导(SV-QTL),47.54%的gPAV与性状相关。典型案例包括:
- Ga1位点gPAV与单侧杂交不亲和:基因PZ00001a032490的存在/缺失与种子结实率关联,存在该基因的个体结实率更高(P=1.91E-43,R²=0.51);
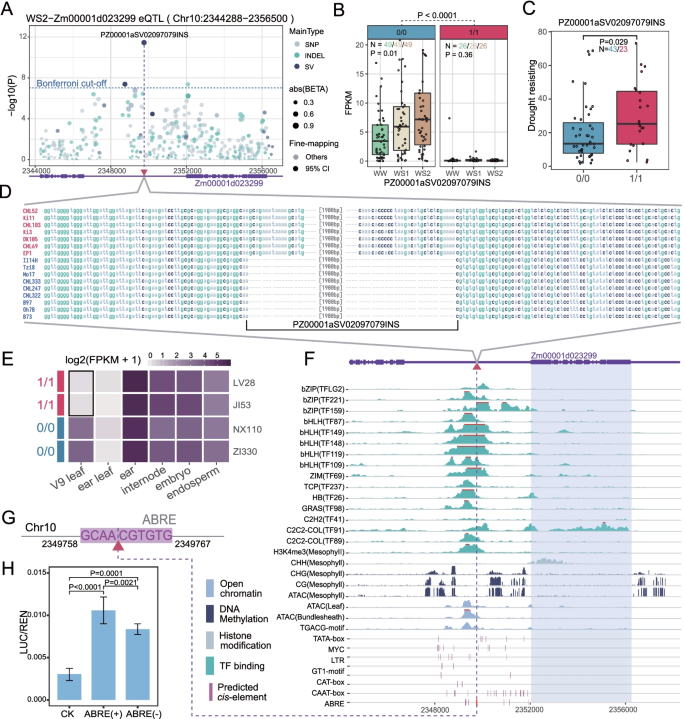
- 转座子插入SV与耐旱性:SV(PZ00001aSV02097079INS)插入到Zm00001d023299基因上游2269 bp处,抑制该基因在叶片中的表达,提高玉米耐旱性(生存率先增加30%,P<0.05)(图4、图5)。
产品关联:文献未提及具体实验产品,领域常规使用GWAS工具(TASSEL)、精细定位工具(FINEMAP)、多组学分析工具(DESeq2、MetaboAnalyst)。
4. Biomarker研究及发现成果
Biomarker定位与筛选逻辑
本研究的生物标志物主要为基因存在/缺失变异(gPAV)和结构变异(SV):
- gPAV:基于泛Zea基因集,通过read-mapping检测个体基因存在状态,筛选与性状关联的gPAV;
- SV:结合全基因组比对和短读长测序检测常见SV,通过GWAS关联性状。
研究过程详述
- gPAV的来源与验证:gPAV来自泛Zea基因组的基因注释,验证方法为read-mapping(用BWA-MEM将个体reads比对到泛基因组,计算基因覆盖度判断存在/缺失)。例如,Ga1位点的gPAV(PZ00001a032490)在teosinte中存在率为85.97%,在玉米中为40.35%(Fisher精确检验P<2.2e-16)。
- SV的来源与验证:SV来自全基因组比对和短读长测序,验证方法为split-read和paired-end分析。例如,耐旱性相关SV(PZ00001aSV02097079INS)在耐旱材料中存在率为60%,敏感材料中为20%(P<0.01)。
核心成果提炼
- gPAV作为生物标志物:首次在泛Zea基因组中鉴定到野生种来源的gPAV(如PZ00001a032490),解释了玉米驯化过程中丢失的抗逆基因,为杂交育种提供了单侧不亲和性状的分子标记。
- SV作为生物标志物:鉴定到TE相关SV(如PZ00001aSV02097079INS),通过影响基因表达调控耐旱性,为玉米抗逆育种提供了新的分子靶点。
这些生物标志物的创新性在于:补充了单参考基因组的生物标志物库,整合了野生种的遗传资源,为玉米分子育种提供了更全面的选择。
本研究构建的泛Zea基因组和解析的gPAV/SV模式,为玉米遗传多样性研究和分子育种提供了重要资源,推动了玉米从“单参考基因组”向“泛基因组”的育种范式转变。
