1. 领域背景与文献引入
文献英文标题:Widespread duplications in the genomes of laboratory stocks of Dictyostelium discoideum;发表期刊:Genome Biology;影响因子:未公开;研究领域:盘基网柄菌遗传学与基因组结构变异。
结构变异(如基因重复、缺失、易位)是真核生物遗传变异的关键来源,已在人类、小鼠、酵母等物种中被广泛报道:人类基因组中存在数百个拷贝数变异(CNVs)位点,与帕金森病、胰腺炎等疾病直接相关;小鼠的CNVs影响复杂表型;酵母的染色体重复与环境适应相关。盘基网柄菌(Dictyostelium discoideum)是研究细胞发育、社会行为及信号通路的经典模式生物,其实验室菌株主要衍生自1933年分离的NC4株。此前研究发现部分菌株存在染色体重复(如Ax3的染色体2重复)或易位,但未系统调查实验室菌株的基因组重复广度、分布及功能影响,这导致菌株间表型差异的遗传基础不明,严重干扰遗传研究的可重复性。
本文旨在通过阵列比较基因组杂交(aCGH)系统解析盘基网柄菌实验室菌株的基因组重复情况,揭示其分布、起源、稳定性及对基因表达的影响,为解决菌株间表型差异问题提供遗传依据。
2. 文献综述解析
作者的综述逻辑围绕“结构变异的普遍性→盘基网柄菌的研究空白”展开:
1. 真核生物结构变异的研究基础:人类CNVs与疾病相关(如α-突触核蛋白重复导致帕金森病);模式生物(小鼠、酵母)的CNVs影响表型(如酵母重复与药物抗性)。
2. 盘基网柄菌的前期研究:部分菌株存在染色体重复(如Ax3的染色体2重复)、易位或大小差异(脉冲场电泳结果),但未系统调查实验室菌株的重复情况,也未明确重复的功能效应。
3. 现有研究的局限性:缺乏对盘基网柄菌实验室菌株重复的全基因组分析,不清楚重复的起源、稳定性及对基因表达的影响。
本文的创新点在于:首次用aCGH系统调查盘基网柄菌实验室菌株的重复情况,揭示了重复的广泛性、稳定性及剂量效应,为菌株标准化提供了遗传标记。
3. 研究思路总结与详细解析
整体框架
研究目标:系统调查盘基网柄菌实验室菌株的基因组重复情况。
核心科学问题:重复的染色体分布、起源稳定性、对基因表达的影响。
技术路线:aCGH检测CNVs→分析重复的染色体分布→谱系分析验证稳定性→转录组分析剂量效应→野生株遗传变异分析。
3.1 菌株选择与DNA样本制备
实验目的:获取覆盖不同谱系的盘基网柄菌菌株DNA,包括axenic菌株(适应无菌培养)、NC4衍生菌株(原始分离株后代)、野生分离株。
方法细节:选择11株axenic菌株(Ax2、Ax3/4)、8株NC4衍生菌株(如NC4(S)、NC4(L))、7株野生分离株(如NYA64、WS205);用SM琼脂(与Klebsiella aerogenes共培养)或axenic培养基培养;蛋白酶K消化+酚-氯仿抽提基因组DNA。
结果解读:获得高纯度DNA(OD260/280≈1.8),满足aCGH要求。
产品关联:文献未提及具体DNA提取试剂,领域常规使用蛋白酶K、酚-氯仿等。
3.2 阵列比较基因组杂交(aCGH)
实验目的:全基因组检测不同菌株的拷贝数变异。
方法细节:
- 微阵列设计:定制DNA微阵列,包含9247个PCR产物(覆盖8579个基因,基于Ax4(Ku)基因组);
- 标记与杂交:参考菌株为Ax2(Ka),样本DNA用Cy3/Cy5标记(随机引物延伸法,Klenow片段);杂交后用Genepix 4000B扫描仪扫描;
- 数据分析:Genepix 3软件定量→Kooperberg背景扣除→print-tip loess归一化→Limma模型拟合;过滤低强度探针(log2强度<6)。
结果解读:
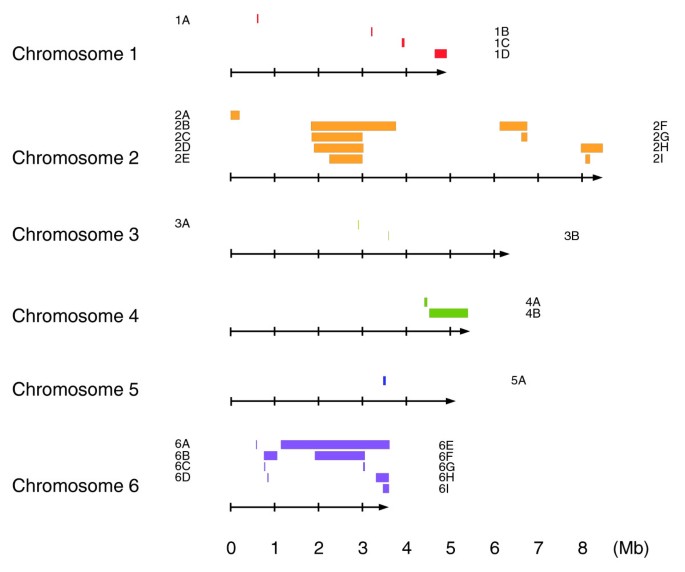
- Axenic菌株:Ax3/4均携带染色体2的750kb重复(log2比值均值0.60),Ax2携带染色体1的26kb重复;
- NC4衍生菌株:多携带亚端粒重复(如NC4(L)的染色体2重复、NC4(S)的染色体6重复);
- 野生分离株:CNVs较少,存在少量缺失或高 divergence区域(如NYA64的染色体3区域)。
产品关联:实验所用关键产品:Genepix 4000B扫描仪(Molecular Devices)、Klenow片段(Invitrogen)、Limma软件。
3.3 重复区域的验证
实验目的:验证aCGH检测到的重复断点准确性。
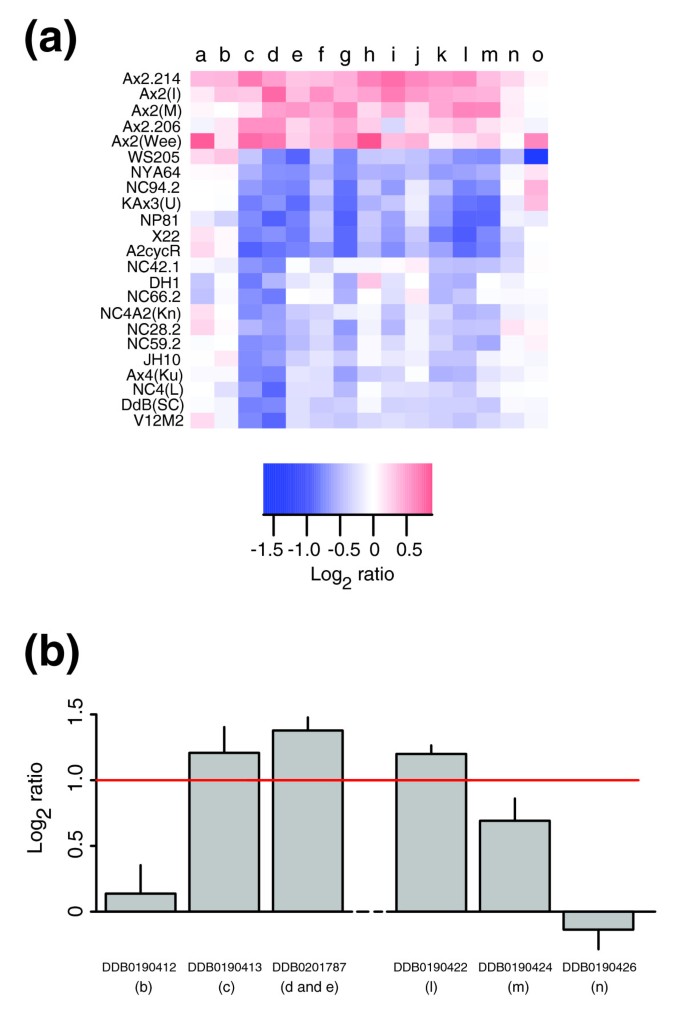
方法细节:以Ax2(Ka)的染色体1重复为例,设计引物对重复区域(DDB0190411-DDB0190427)和对照区域进行qPCR;用Mx3000P热循环仪检测Ct值,计算log2比值(Ax2(Ka) vs Ax4(Ku))。
结果解读:qPCR确认重复的断点,log2比值与aCGH一致(如DDB0190418的log2比值为0.8±0.1,n=4)。
产品关联:实验所用关键产品:Mx3000P热循环仪(Stratagene)、Superscript III和Platinum Taq(Invitrogen)。
3.4 转录组分析
实验目的:研究重复对基因表达的剂量效应。
方法细节:
- RNA提取:Ax2(Wee)(携带染色体2的1179kb重复)和Ax2(Ka)的总RNA用RNeasy试剂盒提取;
- 转录组标记:锚定oligo(dT)引物+Superscript III反转录,Cy3/Cy5标记mRNA;
- 杂交与分析:用同一微阵列杂交,分析重复区域的基因表达log2比值。
结果解读:
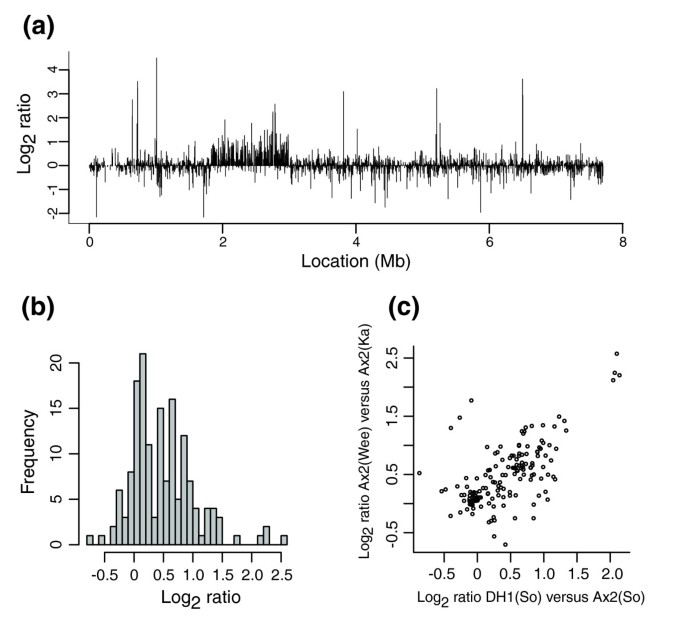
- 重复区域的基因表达呈 bimodal分布:部分基因表达随剂量增加(log2比值≈1),部分存在剂量补偿(log2比值≈0);
- 与DH1(Ax3衍生株)的转录组数据高度相关(r=0.65,p<0.001),说明剂量效应具有株系稳定性。
产品关联:实验所用关键产品:RNeasy试剂盒(Qiagen)、Superscript III(Invitrogen)。
3.5 野生分离株的遗传变异分析
实验目的:检测野生分离株的遗传变异(缺失或高 divergence)。
方法细节:用aCGH检测野生株(如NYA64、WS205)的log2比值,筛选log2比值< -3的区域(缺失或高 divergence)。
结果解读:
- 野生株的CNVs较少,但存在少量缺失或高 divergence区域(如NYA64的染色体3区域);
- 地理相关性:日本的NYA64最 diverged(log2比值< -3的基因占0.2%),美国分离株聚类更紧密。
产品关联:同3.2的aCGH试剂。
4. Biomarker研究及发现成果解析
Biomarker定位
本文的Biomarker为拷贝数变异(CNVs),作为盘基网柄菌菌株鉴别的遗传标记,类型包括:
- 染色体片段重复(如Ax3的染色体2重复、Ax2的染色体1重复);
- 亚端粒重复(如NC4(L)的染色体2重复、NC4(S)的染色体6重复)。
筛选逻辑:aCGH全基因组扫描→菌株谱系分析验证CNVs的稳定性→qPCR验证CNVs的断点→转录组验证CNVs的功能效应。
研究过程详述
- 来源:不同实验室菌株的基因组DNA(如Ax2来自Gerisch实验室,Ax3来自Loomis实验室);
- 验证方法:
- aCGH:检测全基因组CNVs(分辨率≥30kb);
- qPCR:验证CNVs的断点(如Ax2的染色体1重复);
- 转录组分析:验证CNVs的功能效应(如重复区域的基因表达变化);
- 特异性与敏感性:
- 特异性:不同菌株有特有CNVs(如Ax3的染色体2重复仅存在于Ax3 lineage的7株菌株中);
- 敏感性:aCGH能检测到log2比值>0.2的CNVs(对应≥30kb的重复)。
核心成果
- CNVs作为菌株谱系的遗传标记:
- Ax3 lineage的染色体5重复稳定存在于7株菌株中,可追溯株系历史(如DH1继承自Ax3(Dev));
- NC4衍生菌株的亚端粒重复可区分不同实验室的NC4株(如NC4(L) vs NC4(S))。
- CNVs影响基因表达:
- 部分基因表达随剂量增加(如染色体2重复区域的基因log2比值≈1,n=11,P<0.05);
- 部分基因存在剂量补偿(log2比值≈0,如DDB0233427,n=5,P>0.05),可能涉及转录调控。
- 野生分离株的CNVs特征:
- 野生株的CNVs较少,可能反映自然环境中的选择压力(如抗逆性);
- 地理来源相关的高 divergence区域(如NYA64的染色体3区域),可能与局部适应有关。
统计学结果:
- Ax3的染色体2重复log2比值均值0.60(n=11,P<0.05);
- Ax2的染色体1重复qPCR log2比值±标准误(n=4);
- 转录组分析中重复区域的基因表达log2比值分布非正态(Shapiro-Wilk test,p=3.6×10^-6)。
综上,本文揭示了盘基网柄菌实验室菌株基因组重复的广泛性,为菌株标准化和遗传研究的可重复性提供了关键遗传依据。
