1. 领域背景与文献引入
文献英文标题:Interaction between the microbiome and TP53 in human lung cancer;发表期刊:Genome Biology;影响因子:未公开;研究领域:肺癌微生物组与基因-环境相互作用。
肺癌是全球最常见的癌症诊断类型,也是癌症相关死亡的首要原因(年死亡约158,080例)。吸烟作为肺癌的主要风险因素,可通过损伤呼吸道上皮细胞、降低屏障完整性,增加肺部感染的易感性。近年来,肺部微生物组的研究逐渐兴起,已有证据表明微生物组失调与慢性阻塞性肺疾病(COPD)、囊性纤维化等呼吸系统疾病密切相关,但微生物组在肺癌发生发展中的作用尚未完全明确。现有研究发现,结肠癌症中微生物(如Fusobacterium nucleatum、Bacteroides fragilis)可通过促进炎症、破坏DNA等机制参与肿瘤发生,且TP53突变会破坏结肠上皮屏障,导致细菌浸润并促进肿瘤进展。然而,肺癌中微生物组与TP53突变、吸烟的相互作用仍不清楚——这一研究空白为本研究提供了核心探索方向:即吸烟与体细胞突变(如TP53)是否会共同诱导肺部微生物组失调,进而参与肺癌发生。
2. 文献综述解析
作者在综述中围绕“微生物组与肿瘤发生”“TP53突变的屏障功能”两条主线梳理现有研究:
其一,微生物组在呼吸系统疾病中的作用:COPD患者肺部微生物多样性显著增加,囊性纤维化患者气道微生物组成以Pseudomonas为主,提示微生物组失调与呼吸道慢性炎症密切相关;其二,微生物在结肠癌症中的致癌机制:F. nucleatum通过招募肿瘤促进性髓样细胞、激活炎症信号通路促进肿瘤进展,B. fragilis分泌的毒素可直接损伤DNA,诱导结肠上皮细胞突变;其三,TP53突变的屏障功能:结肠癌症中,TP53突变会破坏上皮细胞间连接,允许肠道细菌浸润至肿瘤组织,激活NF-κB信号通路促进肿瘤生长。但现有研究未涉及肺癌中微生物组与TP53突变的关联,且缺乏大样本队列验证。
本研究的创新点在于:①结合16S核糖体RNA(16S rRNA)测序与TCGA RNA-seq数据,系统分析肺癌组织中微生物组的特征;②首次探索肺癌中微生物组与TP53突变、吸烟的相互作用;③重点关注与吸烟关联更紧密的鳞癌(SCC)亚型,揭示其特异性微生物特征。
3. 研究思路总结与详细解析
本研究采用“队列研究+多组学验证+功能确认”的闭环设计,核心实验环节包括样本收集、微生物组测序、TCGA验证、TP53突变分析及FISH确认。
3.1 样本收集与队列设置
实验目的:建立包含肺癌患者与健康对照的研究队列,为微生物组分析提供样本基础。
方法细节:纳入两个独立队列——①NCI-MD队列:33例对照(16例医院活检的良性肺组织、17例立即尸检的非癌肺组织)、143例肺癌病例(肿瘤及癌旁组织),收集临床信息(吸烟状态、组织学亚型、TP53突变状态)并排除抗生素暴露等干扰因素;②TCGA队列:1112例肺癌患者的RNA-seq数据,作为验证集。
结果解读:队列覆盖正常肺、癌旁、肿瘤三种组织类型,以及鳞癌、腺癌两种主要组织学亚型,为分析微生物组与肺癌的关联提供了全面的样本支撑。
产品关联:文献未提及具体实验产品,领域常规使用液氮冷冻保存组织、Qiagen DNeasy试剂盒提取DNA/RNA。
3.2 16S核糖体RNA测序与微生物组多样性分析
实验目的:解析肺癌及对照组织的微生物组成与多样性差异。
方法细节:对NCI-MD队列样本进行16S rRNA V3-V5区测序(Illumina MiSeq平台),数据处理流程包括:①质量控制:过滤含5个以上连续低质量碱基(Phred < Q20)的reads,去除PhiX污染;②污染菌去除:通过对比实验环境拭子、试剂空白等阴性对照,去除Halomonas、Herbaspirillium等潜在污染菌;③OTU聚类:使用QIIME软件将高质量序列聚类为操作分类单元(OTU),基于GreenGenes数据库进行分类注释。
结果解读:①alpha多样性(物种丰富度与均匀度)分析显示,正常肺组织的Chao1指数(丰富度)和Inverse Simpson指数(多样性)显著低于肿瘤组织和癌旁组织(Mann-Whitney p<0.05);②beta多样性分析显示,正常肺、癌旁、肿瘤组织的微生物组成存在显著差异(PERMANOVA p=0.001),且鳞癌与腺癌的微生物组成差异更显著(PERMANOVA p=0.001);③差异丰度分析发现,鳞癌中Acidovorax、Klebsiella等属的丰度显著高于腺癌,且Acidovorax在吸烟者中的丰度显著高于非吸烟者(Kruskal-Wallis p<0.05)。
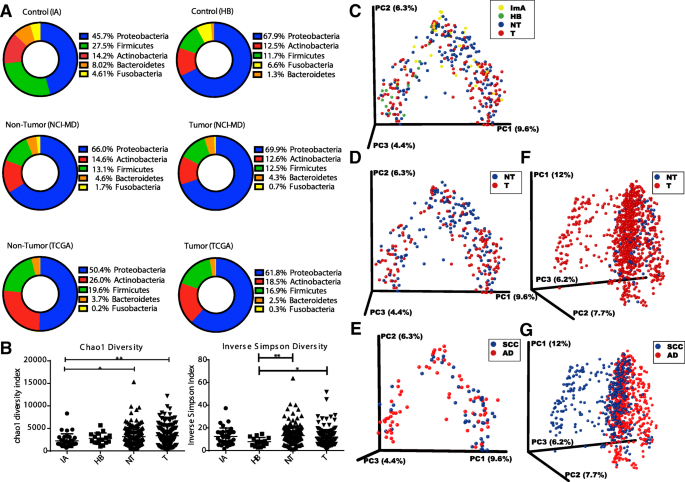
产品关联:测序使用Illumina MiSeq平台及MiSeq Reagent Kit v3试剂,数据分析用QIIME、USEARCH工具,文献未提及具体品牌。
3.3 TCGA RNA-seq数据验证
实验目的:验证NCI-MD队列的微生物组分析结果。
方法细节:提取TCGA队列肺癌组织RNA-seq数据中的“未映射reads”(即非人类来源的序列),使用MetaPhlAn、Kraken、PathoScope三种工具进行微生物分类注释(优先选择PathoScope结果,因其一贯性与准确性更高)。
结果解读:TCGA数据完全验证了NCI-MD队列的发现:①鳞癌中Acidovorax、Klebsiella等属的丰度显著高于腺癌;②Acidovorax在吸烟者中的丰度高于非吸烟者(ANOVA p=0.02);③TP53突变的鳞癌中,这些属的丰度显著高于野生型(FDR校正后p<0.01)。
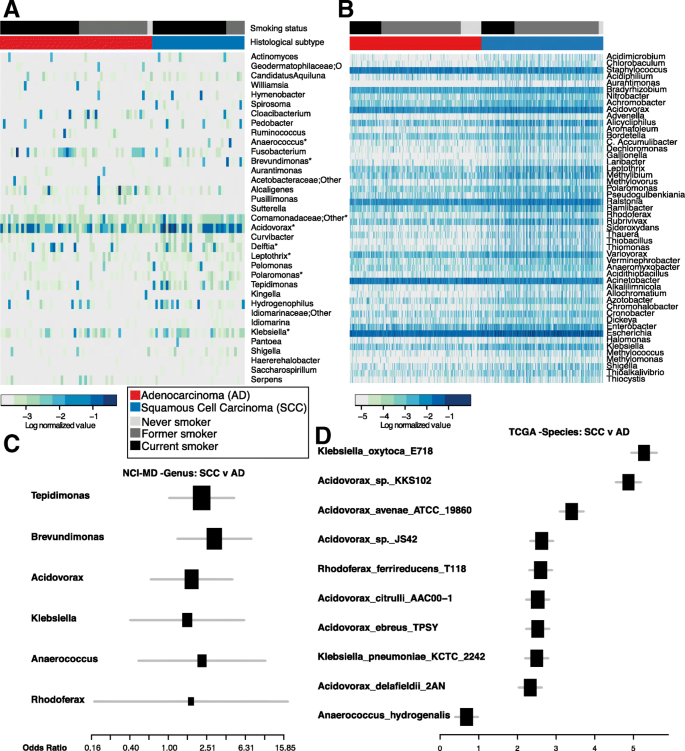
产品关联:文献未提及具体实验产品,领域常规使用Trimmomatic(质量控制)、Bowtie2(人类reads比对)等软件。
3.4 TP53突变状态与微生物组关联分析
实验目的:探索TP53突变对肺癌组织微生物组的影响。
方法细节:对NCI-MD队列的肺癌样本进行TP53靶向测序(Illumina MiSeq平台),检测外显子及邻近剪接位点的突变;TCGA队列直接使用已发表的TP53突变数据。通过比较TP53突变型与野生型样本的微生物丰度,分析两者的关联。
结果解读:①鳞癌中TP53突变率高达75%-80%,显著高于腺癌;②NCI-MD队列中,TP53突变的鳞癌样本中Acidovorax、Klebsiella等属的丰度高于野生型(虽未达统计学显著,但趋势一致);③TCGA队列中,TP53突变的鳞癌样本中这些属的丰度显著高于野生型(FDR校正后p<0.01);④腺癌样本中未观察到TP53突变与微生物丰度的关联。
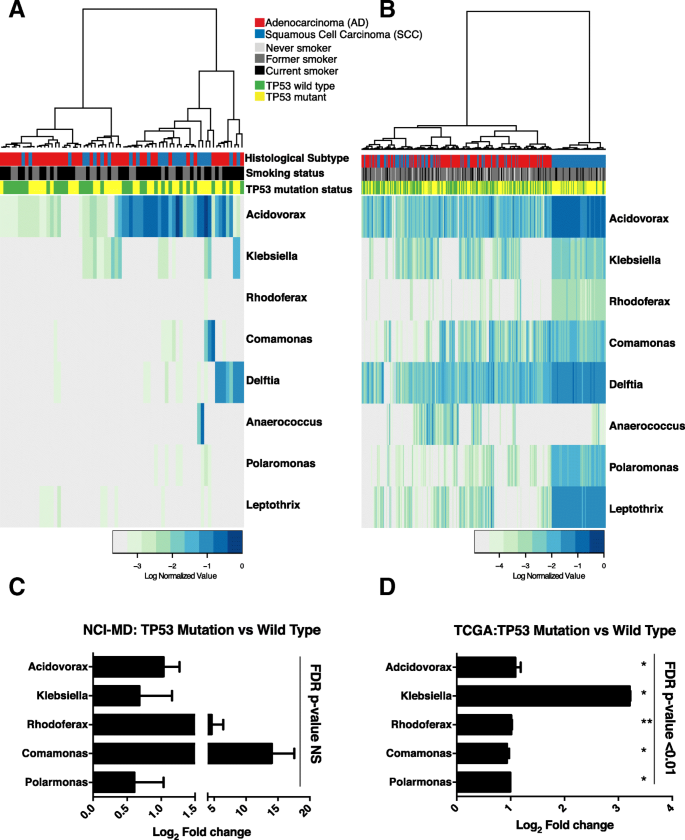
产品关联:TP53测序使用Illumina MiSeq平台及Fluidigm Access Array IFC芯片,文献未提及具体试剂盒品牌。
3.5 荧光原位杂交(FISH)验证微生物存在
实验目的:直观确认Acidovorax在肺癌组织中的物理存在。
方法细节:选取NCI-MD队列的鳞癌样本,制备10 μm冷冻切片,使用Acidovorax特异性荧光探针(序列:CTT TCG CTC CGT TAT CCC,5′标记Alexa Fluor 532)进行FISH实验。杂交后通过Zeiss 710共聚焦显微镜获取图像,使用Imaris软件进行3D重建和定量分析。
结果解读:FISH结果显示,Acidovorax明确存在于鳞癌肿瘤组织中,且主要定位于细胞核周围(图3c、d);定量分析发现,吸烟者鳞癌样本中Acidovorax的阳性细胞比例显著高于非吸烟者(每例计数≥300细胞,n=2)。
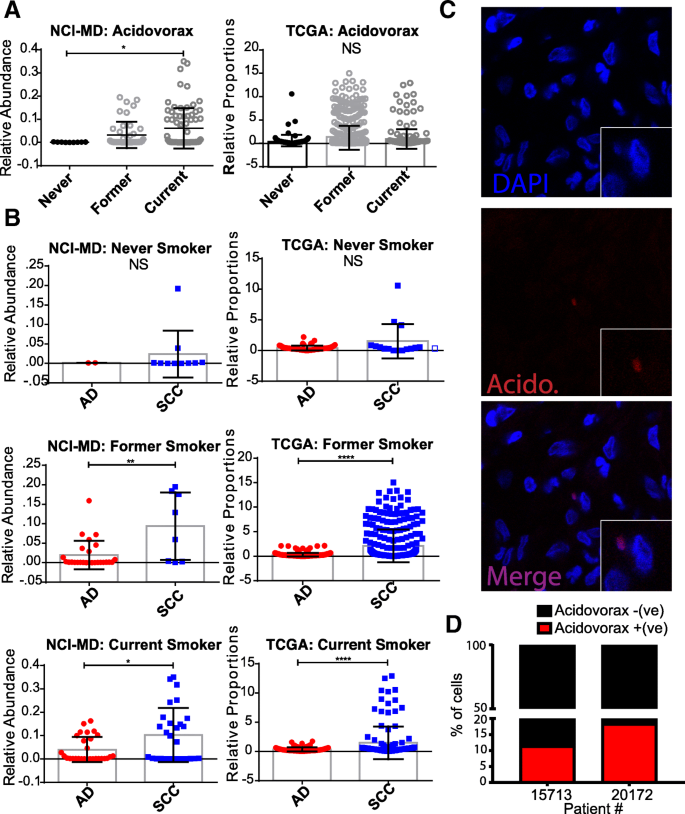
产品关联:FISH探针由Integrated DNA Technologies定制合成,显微镜使用Zeiss 710共聚焦系统,文献未提及具体试剂品牌。
4. Biomarker研究及发现成果解析
Biomarker定位与筛选逻辑
本研究的核心Biomarker为“鳞癌相关微生物群”(包括Acidovorax、Klebsiella、Rhodoferax等属),其筛选与验证逻辑形成完整闭环:①通过NCI-MD队列16S rRNA测序,筛选出鳞癌中差异丰度的微生物属;②利用TCGA队列RNA-seq数据,验证这些属与鳞癌、吸烟、TP53突变的关联;③通过FISH实验,确认Acidovorax在鳞癌组织中的物理存在。
研究过程与数据支撑
- 样本来源:NCI-MD队列的肺癌组织(肿瘤及癌旁)、TCGA队列的肺癌组织RNA-seq数据;
- 验证方法:16S rRNA测序(检测微生物组成)、RNA-seq(验证微生物丰度)、FISH(确认微生物存在);
- 特异性与敏感性:Acidovorax在吸烟者鳞癌中的丰度显著高于非吸烟者(Kruskal-Wallis p<0.05),在TP53突变的鳞癌中的丰度显著高于野生型(TCGA中FDR校正后p<0.01)。
核心成果与创新性
本研究的核心成果在于揭示了“微生物组-基因-环境”的三方相互作用:①鳞癌中存在特异性微生物群(如Acidovorax),其丰度与吸烟、TP53突变显著正相关;②Acidovorax作为关键属,在吸烟者和TP53突变的鳞癌中富集,且通过FISH实验确认其在肿瘤组织中的存在;③这些微生物群仅在鳞癌中与TP53突变关联,腺癌中无此特征——提示其为鳞癌的特异性生物标志物。
创新性体现在:①首次发现肺癌中微生物组与TP53突变、吸烟的相互作用;②明确了鳞癌中微生物群的特异性,为肺癌的精准分型提供了微生物组层面的依据;③结合多组学数据与功能验证,为微生物组参与肺癌发生的机制研究提供了重要基础。
本研究通过系统解析肺癌组织中微生物组的特征,为肺癌的早期诊断、精准治疗提供了新的生物标志物方向——未来若能进一步验证这些微生物群的功能(如是否促进肿瘤生长),将有望为肺癌的微生物组靶向治疗提供理论支持。
