1. 领域背景与文献引入
文献英文标题:Epigenetic modifications are associated with inter-species gene expression variation in primates;发表期刊:Genome Biology;影响因子:未公开;研究领域:灵长类表观遗传调控与基因表达进化。
基因调控的进化是灵长类物种形成和适应性表型分化的核心驱动力。过去二十年,比较基因组研究揭示灵长类间存在广泛的基因表达水平差异(如Enard等2002年报道的种间基因表达差异率达10-20%),但这些差异的调控机制(尤其是表观遗传修饰的贡献)仍不清楚。现有研究多聚焦单一表观标记(如DNA甲基化、H3K4me3)与基因表达的关联,仅能解释小部分差异(如Pai等2011年发现DNA甲基化仅解释约5%的种间基因表达差异),且缺乏多组蛋白修饰与RNA聚合酶II(Pol II)的联合分析——这两类分子共同构成转录起始的核心调控网络。
针对这一空白,本文以灵长类淋巴母细胞系(Lymphoblastoid Cell Lines, LCLs)为模型,系统分析Pol II和四种组蛋白修饰(H3K4me1、H3K4me3、H3K27ac、H3K27me3)与基因表达种内变异和种间变异的关联,旨在阐明表观遗传修饰在灵长类基因调控进化中的作用。
2. 文献综述解析
作者将现有研究分为三类:
1. 基因表达进化研究:早期研究(如Enard等2002年、Khaitovich等2005年)通过微阵列或RNA-seq揭示灵长类间基因表达差异广泛,但未深入调控机制;
2. 单一表观标记关联研究:Pai等2011年发现DNA甲基化差异与基因表达差异相关,Cain等2011年报道H3K4me3差异与基因表达变化关联,但均仅关注单一标记,解释率有限(<10%);
3. 多物种表观比较研究:Mikkelsen等2007年在干细胞中发现组蛋白修饰差异与基因表达差异相关,但未涉及灵长类成体细胞。
现有研究的局限性在于:① 缺乏多标记联合分析,难以揭示调控网络的复杂性;② 灵长类中的系统研究不足,尤其是种间差异的调控机制。
本文的创新点:① 同时分析Pol II和四种组蛋白修饰(覆盖转录起始的关键调控因子);② 以灵长类LCLs为模型,系统比较种内/种间的表观标记-基因表达关联;③ 用贝叶斯变量选择回归(BVSR)分析标记间交互作用,明确边际效应主导关联的核心结论。
3. 研究思路总结与详细解析
3.1 灵长类淋巴母细胞系模型构建
实验目的:获取同源性高、可重复的灵长类细胞模型,用于表观遗传与转录组分析。
方法细节:选取人类(8个个体)、黑猩猩(8个)、恒河猴(7个,H3K27ac样本缺失1个)的LCLs,培养于含15%胎牛血清的RPMI培养基(补充L-谷氨酰胺、青霉素、链霉素),37℃、5% CO₂培养。
结果解读:三物种细胞形态与生长状态一致,保证后续实验的可比性。
产品关联:细胞系来自Coriell Institute等机构;培养基为常规细胞培养试剂(文献未提及具体品牌)。
3.2 ChIP-seq与RNA-seq数据生成
实验目的:检测Pol II和四种组蛋白修饰的基因组分布,以及基因表达水平。
方法细节:
- ChIP-seq:用特异性抗体沉淀染色质(H3K4me3:Abcam ab8580,4μg;H3K4me1:Millipore 07-436,12μg;H3K27ac:Abcam ab4729,4μg;H3K27me3:Millipore 07-449,4μg;Pol II:Santa Cruz sc-9001,10μg),Covaris S2剪切染色质(40分钟, duty cycle 20%),构建Illumina测序文库;
- RNA-seq:提取总RNA,构建链特异性文库,Illumina GAII/HiSeq测序。
结果解读:获得高质量数据,同物种内个体间标记丰度(Spearman相关系数>0.8)与基因表达(相关系数>0.9)一致性高,验证数据可靠性。
产品关联:ChIP抗体品牌与货号明确;测序文库构建试剂为Illumina标准试剂盒(未提及具体货号)。
3.3 数据处理与正交区域筛选
实验目的:筛选跨三物种的保守区域,确保种间比较的可靠性。
方法细节:
- 数据比对:用BWA将reads比对到参考基因组(人类hg19、黑猩猩panTro3、恒河猴rheMac2),过滤低质量(Q<10)和重复reads;
- 峰calling:用MACS(窄峰,针对H3K4me1/3、H3K27ac、Pol II)和RSEG(宽峰,针对H3K27me3)鉴定富集区域;
- 正交区域筛选:用liftOver保留跨三物种可映射(≥80%碱基相似)的转录起始位点(TSS)和外显子(共26115个基因)。
结果解读:筛选得到308514个正交染色质状态区域,聚焦跨物种保守的功能区域。
产品关联:数据处理用BWA、MACS等开源工具;参考基因组来自UCSC Genome Browser。
3.4 种内标记丰度与基因表达的关联分析
实验目的:验证表观标记与基因表达的种内关联。
方法细节:
- 标记丰度计算:在TSS±2kb区域计算标准化峰read数(RPKM),平方根转换后作为标记丰度;
- 基因分组:将基因按表达水平分为低、中、高三组(每组约8700个基因);
- 关联分析:用线性模型+10-fold交叉验证计算R²(解释方差比例),BVSR分析标记间交互作用。
结果解读:
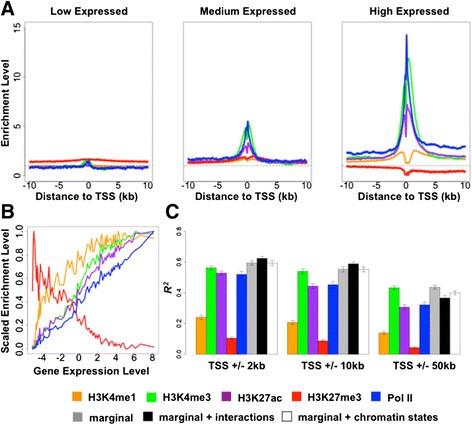
低表达基因的TSS区域H3K27me3丰度高(抑制性标记),高表达基因的H3K4me3、H3K27ac、Pol II丰度高(活性标记,图2A);单一标记中H3K4me3(R²≈25%)、H3K27ac(≈22%)、Pol II(≈20%)关联最强,五种标记联合解释约58%的种内方差(人类59%、黑猩猩58%、恒河猴57%,n=8个体/物种,图2C);交互作用贡献极小(R²增加<2%)。
产品关联:统计分析用R、GEMMA(BVSR);图形绘制用ggplot2。
3.5 种间标记差异与基因表达差异的关联分析
实验目的:探讨表观标记差异与基因表达种间差异的关联。
方法细节:
- 差异鉴定:用线性模型鉴定差异表达基因(DE,FDR<5%)和差异标记区域(FDR<5%);
- 关联分析:比较DE基因与非DE基因的标记差异频率,用线性模型计算R²。
结果解读:
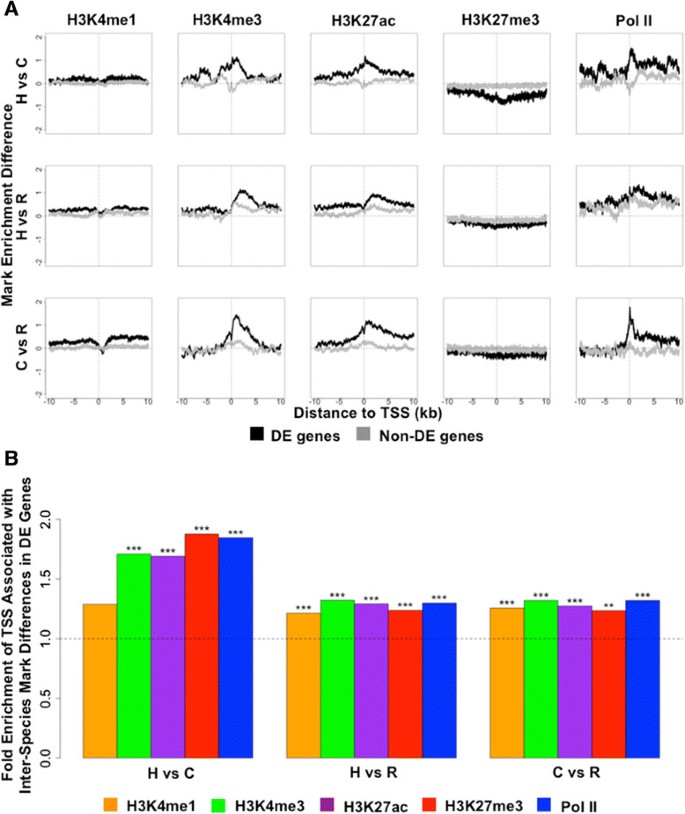
DE基因的标记差异频率显著高于非DE基因(fold enrichment>1.5,P<0.001,binomial test,图3A);标记差异与基因表达差异方向一致(H3K27me3负相关,其他标记正相关,图3B);五种标记联合解释约40%的种间方差(人类vs黑猩猩42%、人类vs恒河猴40%、黑猩猩vs恒河猴38%,FDR<5%,图4B)。
产品关联:差异分析用DESeq2(RNA-seq)、edgeR(ChIP-seq);统计用R、GEMMA。
4. Biomarker研究及发现成果解析
Biomarker定位:本文未聚焦传统疾病Biomarker,而是探讨表观遗传标记作为灵长类基因表达调控进化的Biomarker潜力。研究的标记包括Pol II和四种组蛋白修饰,筛选逻辑基于其在转录起始中的已知功能(如H3K4me3标记活性启动子、Pol II直接参与转录),通过ChIP-seq在灵长类LCLs中系统检测。
研究过程详述:
- 种内关联:标记丰度与基因表达显著相关(H3K4me3等标记R²>20%,n=8个体/物种);
- 种间关联:标记差异与基因表达差异显著相关(联合解释约40%方差,FDR<5%);
- 特异性:DE基因中标记差异的频率是 non-DE基因的1.5-2倍(P<0.001,binomial test)。
核心成果:
1. 表观遗传标记是灵长类基因表达种间变异的关键调控机制,解释约40%的进化差异;
2. H3K4me3、H3K27ac、Pol II是关联最强的标记,可作为灵长类进化调控机制的Biomarker候选;
3. 虽非疾病Biomarker,但为理解灵长类表型进化(如脑容量、行为)的分子基础提供了关键线索。
本文通过系统的表观遗传-转录组分析,首次明确多表观标记的边际效应主导灵长类基因表达种间变异,为灵长类进化的调控机制研究提供了重要框架。
