1. 领域背景与文献引入
文献英文标题:Genome-wide analysis of plant nat-siRNAs reveals insights into their distribution, biogenesis and function;发表期刊:Genome Biology;影响因子:未公开;研究领域:植物小分子RNA(siRNA)生物学。
真核生物基因组中广泛存在顺式天然反义转录本(cis-natural antisense transcripts, cis-NATs),其与正义转录本形成的双链RNA可被RNA干扰(RNAi) machinery加工为小干扰RNA(small interfering RNAs, siRNAs),这类siRNAs被称为nat-siRNAs。此前在植物(如拟南芥)、哺乳动物、果蝇及酵母中虽有少数nat-siRNAs报道,但关于其核心特征(如全基因组分布模式)、生物发生机制(如依赖的Dicer-like蛋白)及生理功能的系统性认知仍存在显著空白。具体而言,现有研究多为个案分析,缺乏全基因组水平的植物nat-siRNAs鉴定;nat-siRNAs的分布规律(如是否富集于cis-NATs重叠区)、生物发生的普适性分子元件(如依赖DCL1还是DCL3)及在胁迫响应中的调控角色尚未明确,限制了对其作为基因表达调控因子的理解。
针对植物nat-siRNAs研究的系统性不足,本研究以生物/非生物胁迫处理的拟南芥和非生物胁迫处理的水稻为材料,通过深度测序结合遗传分析,全基因组鉴定nat-siRNAs并解析其分布、生物发生及功能,为植物nat-siRNAs的存在提供了全基因组尺度的证据,填补了其特性研究的关键空白,推进了对植物小分子RNA调控网络的理解。
2. 文献综述解析
作者对现有研究的分类维度主要基于物种(植物、动物、酵母)和研究方向(nat-siRNAs的鉴定、生物发生机制、功能)。现有研究的关键结论包括:nat-siRNAs普遍存在于真核生物中,由cis-NATs的双链RNA经RNAi途径加工生成;植物中部分nat-siRNAs依赖Dicer-like 1(DCL1)、RNA-dependent RNA polymerase 6(RDR6)及植物特异RNA聚合酶IV(PolIV)等元件,参与盐胁迫响应(如SRO5-P5CDH来源的nat-siRNAs)或生殖发育调控(如KOKOPELLI-ARIADNE14来源的nat-siRNAs)。技术方法上,此前研究通过小RNA测序或Northern blot实现nat-siRNAs的鉴定,为后续研究奠定了基础,但局限性同样明显:一是研究多聚焦于个别基因对,缺乏全基因组水平的系统分析;二是对nat-siRNAs的分布模式(如是否存在位点特异性或分布式)、生物发生的普适机制(如不同DCL蛋白的分工)及功能的认知仍不完整。
本研究的创新价值在于,首次在全基因组水平解析了植物(拟南芥、水稻)胁迫条件下nat-siRNAs的分布特征,明确其富集于cis-NATs重叠区且存在两种分布模式;系统揭示了其生物发生依赖DCL1和/或DCL3的分子机制,区分了不同DCL依赖的nat-siRNAs对RDR/PolIV的需求;关联了nat-siRNAs与植物胁迫响应的功能,为其作为胁迫调控因子的角色提供了证据,弥补了现有研究的系统性缺陷。
3. 研究思路总结与详细解析
本研究的整体框架为:胁迫处理植物→小RNA测序鉴定nat-siRNAs→分布模式分析→遗传突变体验证生物发生机制→表达分析验证功能。核心科学问题包括:植物nat-siRNAs的全基因组分布特征是什么?其生物发生依赖哪些分子元件?是否参与胁迫响应的基因调控?
3.1 植物材料处理与小RNA文库构建
实验目的:获取生物/非生物胁迫下的植物小RNA样品,为nat-siRNAs鉴定提供基础。
方法细节:拟南芥经生物胁迫(非致病性Pseudomonas syringae pv tomato DC3000 hrcC⁻、致病性菌株DC3000及携带avrRpt2的无毒菌株侵染)和非生物胁迫(200 mM NaCl处理24 h、5°C冷处理24 h、干旱处理10天)处理;水稻经非生物胁迫(400 mM NaCl处理24 h、5°C冷处理24 h、干旱处理3天)处理。采集处理后的植物组织(拟南芥全苗、水稻花序),提取总RNA并分离18-26 nt小RNA,连接5’和3’ adaptors后进行RT-PCR及Illumina测序。
结果解读:获得拟南芥(生物胁迫6、14小时,非生物胁迫30天)和水稻(非生物胁迫24小时)的小RNA测序数据,为后续nat-siRNAs鉴定提供了高质量数据集。
实验所用关键产品:文献未提及具体实验产品,领域常规使用T4 RNA连接酶(如New England Biolabs)、Illumina小RNA测序试剂盒等。
3.2 nat-siRNAs的全基因组鉴定与分布模式分析
实验目的:全基因组水平鉴定nat-siRNAs并解析其分布特征。
方法细节:①利用拟南芥(TAIR8)和水稻(MSU RGAP 6.1)基因组注释,筛选重叠长度≥25 nt的cis-NAT对;②将小RNA测序reads与cis-NATs进行100%序列比对,鉴定nat-siRNAs;③通过聚类分析(reads首核苷酸位置≤10 nt为一个簇)解析分布模式:位点特异性(簇数≤10且簇内reads占比>50%)、分布式(否则);④统计链偏好性(某链reads数是另一链2倍以上)。
结果解读:拟南芥鉴定出17141条独特nat-siRNAs(对应33826条总reads),水稻鉴定出56209条独特nat-siRNAs(对应162397条总reads);nat-siRNAs显著富集于cis-NATs重叠区域(拟南芥重叠区siRNA密度是非重叠区的6倍以上,P=0.0077;水稻为6倍以上,P=0.0242),且存在两种分布模式:
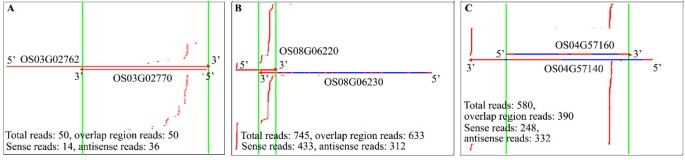
- 分布式:siRNAs分散于重叠区或整个转录本(如拟南芥At2g41590/At2g41600对,图1j;水稻OS08G06220/OS08G06230对,图2b);
- 位点特异性:siRNAs来自1-2个特定位点(如拟南芥At1g51400/At1g51402对,图3c;水稻OS04G57140/OS04G57160对,图2c)。
此外,拟南芥50.5%、水稻52.9%的cis-NAT对显示链偏好性(如At1g13330/At1g13340的nat-siRNAs主要来自At1g13340链,图1o)。


实验所用关键产品:文献未提及具体实验产品,领域常规使用Bowtie序列比对工具、RNAfold预测RNA二级结构。
3.3 nat-siRNAs生物发生机制的遗传分析
实验目的:解析nat-siRNAs生物发生依赖的分子元件(DCL、RDR、PolIV等)。
方法细节:选取15个高丰度nat-siRNAs(reads数≥10 per million),设计锁定核酸(LNA)探针,通过Northern blot检测其在小RNA通路突变体(dcl1-7 fwf2、dcl3-1、rdr2-1、polIV-3等)中的表达水平。
结果解读:nat-siRNAs的生物发生依赖DCL1和/或DCL3:
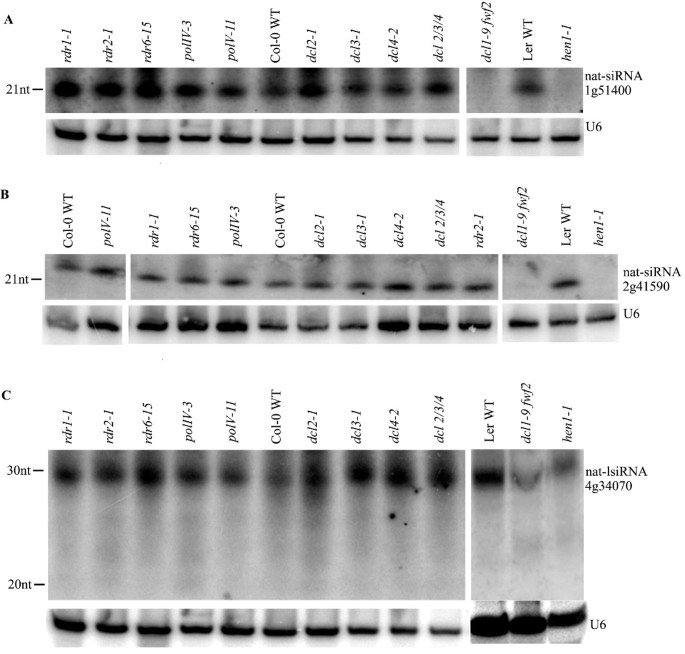
- 21 nt左右的nat-siRNAs:主要依赖DCL1(如nat-siRNA1g51400在dcl1突变体中消失,图4a;nat-siRNA2g41590在dcl1突变体中消失,图4b);
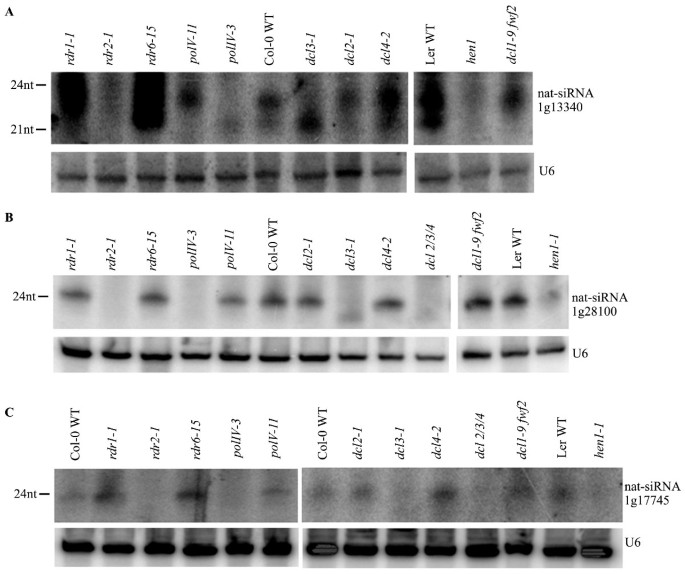
- 24 nt左右的nat-siRNAs:主要依赖DCL3(如nat-siRNA1g13340在dcl3突变体中消失,图5a;nat-siRNA1g28100在dcl3突变体中消失,图5b)。
进一步分析发现,DCL3依赖的nat-siRNAs同时依赖RDR2和PolIV(如nat-siRNA1g13340在rdr2、polIV突变体中均消失,图5a);而部分DCL1依赖的nat-siRNAs不依赖RDR或PolIV(如nat-siRNA1g51400在rdr1/2/6、polIV突变体中仍存在,图4a)。
实验所用关键产品:LNA探针(序列见附加文件7)、PerfectHyb Plus杂交缓冲液(Sigma-Aldrich)、U6 snRNA探针(内参)。
3.4 nat-siRNAs的功能分析
实验目的:验证nat-siRNAs对顺式NAT转录本的调控作用及与胁迫响应的关联。
方法细节:①利用dcl1-7突变体与野生型的微阵列数据(GEO accession GSE2473),通过Rank Product法鉴定差异表达的cis-NAT基因(筛选标准:dcl1-7中上调≥1.5倍且P<0.05);②选取差异基因(如At1g51402、At1g13330),通过qRT-PCR验证其在dcl1-7 fwf2、dcl3-1突变体中的表达;③统计nat-siRNAs在不同胁迫下的表达变化(≥2倍差异)。
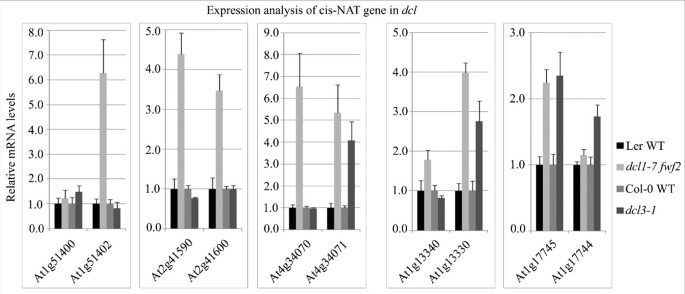
结果解读:①dcl1-7突变体中23.7%的cis-NAT基因(22/93)上调,显著高于全基因组水平(7.9%),表明DCL1依赖的nat-siRNAs可顺式下调靶标NAT转录本;②qRT-PCR验证显示,At1g51402在dcl1-7 fwf2突变体中上调6倍(图6),At1g13330在dcl1-7 fwf2突变体中上调4倍(图6),而在dcl3-1突变体中无显著变化,进一步支持DCL1依赖的nat-siRNAs的调控作用;③胁迫响应分析显示,拟南芥80对、水稻37对cis-NATs的nat-siRNAs在至少一种胁迫下表达变化≥2倍(如拟南芥At2g41590/At2g41600的nat-siRNAs在盐胁迫下上调),提示其参与植物胁迫响应。
实验所用关键产品:qRT-PCR试剂(如Invitrogen SuperScript II反转录酶)、实时PCR仪(Bio-Rad MyiQ)、Actin2引物(内参)。
4. Biomarker研究及发现成果解析
Biomarker定位
本研究中的nat-siRNAs可作为植物胁迫响应的潜在分子标记(Biomarker),其筛选与验证逻辑为:胁迫处理植物→小RNA测序鉴定nat-siRNAs→突变体验证生物发生机制→表达分析关联胁迫响应→功能验证(调控NAT转录本)。
研究过程详述
- 来源:生物/非生物胁迫处理的拟南芥全苗及非生物胁迫处理的水稻花序的小RNA文库;
- 验证方法:①通过Northern blot验证其在突变体中的表达(如nat-siRNA1g51400在dcl1突变体中消失,确认依赖DCL1);②通过qRT-PCR验证其靶标NAT转录本在突变体中的表达变化(如At1g51402在dcl1突变体中上调,确认nat-siRNAs的调控作用);③通过测序数据统计其在不同胁迫下的表达变化(如拟南芥80对cis-NATs的nat-siRNAs在胁迫下变化≥2倍);
- 特异性:nat-siRNAs显著富集于cis-NATs重叠区域(拟南芥P=0.0077,水稻P=0.0242),且部分具有链偏好性(如At1g13330/At1g13340的nat-siRNAs主要来自At1g13340链);
- 敏感性:部分nat-siRNAs在胁迫下的表达变化超过2倍(如拟南芥80对cis-NATs的nat-siRNAs)。
核心成果提炼
- 分布特征:植物nat-siRNAs富集于cis-NATs重叠区,存在分布式和位点特异性两种模式,部分具有链偏好性;
- 生物发生:依赖DCL1和/或DCL3,其中DCL3依赖的nat-siRNAs同时依赖RDR2和PolIV,DCL1依赖的nat-siRNAs部分不依赖RDR/PolIV;
- 功能:可顺式调控NAT转录本的表达(如At1g51402在dcl1突变体中上调6倍,n=3,P<0.05),且部分nat-siRNAs的积累受胁迫调控(如拟南芥80对cis-NATs的nat-siRNAs在至少一种胁迫下变化≥2倍),提示其作为植物胁迫响应分子标记的潜力。
本研究首次全基因组解析了植物nat-siRNAs的特性,为其作为基因表达调控因子的角色提供了系统证据,推进了对植物小分子RNA调控网络的理解。
