1. 领域背景与文献引入
文献英文标题:Beyond the Hox complex;发表期刊:Genome Biology;影响因子:未公开;研究领域:发育生物学(同源框基因演化与基因组结构研究)
同源框基因是一类编码转录因子的保守基因家族,1984年其核心同源框序列首次被鉴定,开启了发育调控基因演化的研究领域。90年代以来,Hox基因簇的保守性研究取得关键突破,1994年Garcia-Fernandez和Holland发现文昌鱼Hox基因簇的原型结构,证实其在动物轴向模式形成中的核心作用。当前领域研究热点聚焦于基因组共线性(synteny)与基因簇演化的功能关联,未解决的核心问题包括脊椎动物祖先基因组中同源框基因的整体组织形式、不同同源框基因家族的演化关联性,以及驱动基因簇形成与分化的分子机制。现有研究多局限于单一同源框基因家族的分析,缺乏跨物种、多家族的系统整合研究,无法全面揭示祖先基因组的结构特征。Pollard和Holland的研究正是针对这一空白,通过整合多物种的基因连锁数据,提出脊椎动物祖先存在包含多类同源框基因的大型簇状结构,为理解同源框基因的演化历程提供了全新框架。
2. 文献综述解析
作者以同源框基因的演化脉络为核心评述逻辑,从祖先基因的起源、现存物种的基因分布、演化驱动力三个维度,系统梳理了领域内的研究进展与不足。
现有研究已充分验证Hox基因簇的高度保守性,在从果蝇到人类的几乎所有动物类群中,Hox基因均以簇状形式存在并参与身体轴向的模式形成,技术上主要依赖序列比对、单物种基因组测序及基因表达分析,这些方法在揭示单一基因家族的演化规律上具有高特异性,但存在明显局限性:多数研究仅聚焦于Hox基因簇本身,对NK、engrailed等其他同源框基因家族的基因组组织演化关注不足;跨物种分析多局限于两两物种的比较,缺乏多物种数据的系统整合来推断祖先基因组结构;对基因簇演化的功能约束机制研究尚不深入,未明确祖先基因簇的形成原因及维持动力。
本研究突破了现有研究的局限,首次通过整合人类、小鼠、果蝇等多个物种的基因连锁数据,采用“拼图式”共线性分析方法,提出脊椎动物祖先基因组中存在包含EHGBox、Extended Hox、NKL三大类同源框基因的大型连锁簇,将同源框基因的演化研究从单一Hox复合体拓展至更广泛的基因家族网络,为理解脊椎动物基因组结构的演化起源提供了关键证据。
3. 研究思路总结与详细解析
本研究的核心目标是推断脊椎动物祖先基因组中同源框基因的组织形式,核心科学问题是同源框基因在脊椎动物祖先中是否形成超越Hox复合体的更大规模连锁簇,技术路线遵循“跨物种数据整合→概率模型验证→演化场景构建”的闭环逻辑,通过多物种共线性分析与统计建模,系统还原了同源框基因簇的演化历程。
3.1 跨物种基因连锁数据整合
实验目的是通过多物种间的基因连锁信息,识别演化保守的基因区域,为推断祖先基因组结构提供基础。方法细节是收集人类、小鼠、果蝇等物种中同源框基因(包括NK、engrailed、Hox家族)及连锁功能基因(如角蛋白、hedgehog蛋白、FGF受体)的染色体定位数据,采用“拼图式”比对方法,将不同物种中存在连锁关系的基因进行匹配,识别演化过程中保留的共线性区域。结果解读:分析发现多个同源框基因家族在不同物种中存在保守连锁,例如人类中多个NK类基因集中分布于同一染色体带,其果蝇同源基因也位于同一染色体区域,这一结果支持这些基因在脊椎动物祖先中处于同一连锁簇的假设。产品关联:文献未提及具体实验产品,领域常规使用基因组数据库检索工具(如Ensembl、NCBI Map Viewer)及序列比对软件(如BLAST)开展相关分析。
3.2 演化概率模型构建
实验目的是验证跨物种共线性分析的可靠性,明确祖先基因在现存物种中保留连锁的概率。方法细节基于已发表的染色体重排频率数据,包括人类与小鼠基因组之间约180次染色体重排、河豚与人类同源基因的连锁保留概率(40-50%),结合脊椎动物祖先与现存物种的演化时间(约6亿年),构建基因连锁保留的概率模型。结果解读:模型计算得出,脊椎动物祖先中相邻的两个基因,在现存物种中仍保持连锁的概率约为50%,这一结果为跨物种共线性分析推断祖先基因组结构的合理性提供了统计学支持。产品关联:文献未提及具体实验产品,领域常规使用统计分析软件(如R、Python)进行概率模型构建与数据分析。
3.3 祖先基因簇演化场景推演
实验目的是基于共线性数据与概率模型,构建同源框基因簇从脊椎动物祖先到现存物种的完整演化路径。方法细节整合基因串联重复、全染色体复制、基因丢失及染色体重排的现有研究数据,结合Hox基因簇的已知演化历程,提出包含多阶段事件的演化场景。结果解读:研究提出,脊椎动物祖先的单一染色体上存在包含EHGBox、Extended Hox、NKL三大类同源框基因的大型连锁簇(至少包含30个基因),而ParaHox基因簇虽同时存在但未与上述簇连锁;后续脊椎动物支系发生全染色体复制,伴随部分基因丢失,随机染色体重排逐渐打破大型连锁簇,形成现存人类、小鼠等物种中同源框基因的分散分布模式。该演化场景通过图1直观展示,清晰呈现了基因簇从整合到分散的过程。
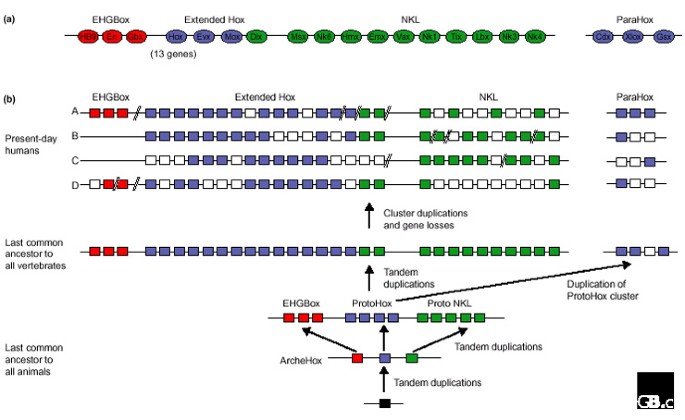
产品关联:文献未提及具体实验产品,领域常规使用基因组可视化工具(如Circos、IGV)展示演化场景与基因连锁关系。
4. Biomarker研究及发现成果解析
本文聚焦于基因组结构演化研究,涉及的分子标记为同源框基因的保守连锁区域(属于演化类基因组结构Biomarker),其核心价值在于作为推断脊椎动物祖先基因组结构的关键依据。
Biomarker定位:该类Biomarker属于基因组结构标记,筛选与验证逻辑为“跨物种共线性分析→概率模型验证→演化场景推演”,通过多物种间基因连锁的保守性,反向推断祖先基因组的基因组织形式。研究过程详述:标记来源为人类、小鼠、果蝇等多个物种的公共基因组数据库,验证方法为跨物种共线性比对与统计概率分析,特异性表现为不同物种中同源基因的连锁保留模式符合演化时间对应的概率(如河豚与人类同源基因的连锁保留概率为40-50%),敏感性数据文献未明确提供。核心成果提炼:该基因组结构标记首次揭示了脊椎动物祖先存在包含至少30个同源框基因的大型簇状结构,将Hox复合体纳入更广泛的同源框基因演化网络,为研究基因簇演化的功能约束提供了全新视角;研究的统计学支持为基因连锁保留概率约50%(基于6亿年演化时间,文献未明确样本量),该发现填补了脊椎动物祖先基因组结构研究的空白,为后续探索基因簇演化的功能意义奠定了基础。
