1. 领域背景与文献引入
文献英文标题:Drought-responsive dynamics of H3K9ac-marked 3D chromatin interactions are integrated by OsbZIP23-associated super-enhancer-like promoter regions in rice;发表期刊:Genome Biology;影响因子:17.906(2023年);研究领域:植物逆境生物学(水稻干旱胁迫响应)
水稻作为全球首要主粮作物,同时也是对干旱高度敏感的作物,过去数十年已成为植物抗旱研究与育种的核心模式物种。领域发展关键节点清晰:早期研究揭示了干旱响应基因的复杂共表达网络,但多基因聚合等育种策略培育的抗旱材料效果仍未达预期;组学技术兴起后,组蛋白修饰(如H3K4me3)与干旱响应的关联被发现,但H3K4me3的干旱特异性修饰区域仅占1.6%,与基因表达的相关性较弱;3D基因组研究层面,植物缺乏哺乳动物中的CTCF蛋白,以染色质互作域(CIDs)作为核心空间互作单位,此前仅发现昼夜节律相关的启动子-启动子互作(PPIs),但干旱胁迫下水稻3D染色质互作的动态变化及其调控基因共表达的机制仍属空白,这一核心问题严重制约了水稻抗旱育种的精准性。
针对上述研究空白,本研究以H3K9ac这一响应环境胁迫的活性组蛋白修饰为标记,通过高分辨率ChIA-PET技术绘制水稻干旱及复水条件下的3D基因组图谱,揭示超级启动子区域(SPRs)整合启动子-启动子互作的超级增强子样功能,并明确OsbZIP23作为关键调控因子介导干旱特异性3D染色质互作的机制,为水稻抗旱育种提供了全新的表观遗传调控靶点。
2. 文献综述解析
作者从组蛋白修饰调控干旱响应、3D基因组结构与基因表达调控两个核心维度对现有研究进行系统评述,梳理了领域内的研究进展与局限性。
组蛋白修饰研究方面,现有研究已证实H3K4me3在干旱胁迫下会发生重塑,但该修饰的干旱特异性区域占比极低,且与基因表达的相关性较弱,无法全面解释干旱响应基因的表达调控;H3K9ac在拟南芥、玉米等植物中被发现响应干旱胁迫,但水稻中缺乏直接证据支持其参与干旱响应的调控。3D基因组研究方面,现有研究明确了植物以染色质互作域(CIDs)替代哺乳动物的拓扑关联域(TADs)作为核心空间互作单位,且发现了昼夜节律相关的启动子-启动子互作,但干旱胁迫下水稻3D染色质互作的动态变化及调控因子尚未明确;同时,基于RNAPII的ChIA-PET数据因RNAPII的管家基因属性,无法解析胁迫特异性的3D基因组调控机制。
现有研究的核心局限性在于:缺乏干旱胁迫下水稻高分辨率3D基因组图谱,未明确染色质互作调控干旱响应基因共表达的具体分子机制,尤其是转录因子如何参与3D染色质互作的形成与动态调控。本研究的创新价值在于:首次绘制水稻干旱及复水条件下H3K9ac标记的高分辨率3D基因组图谱,发现超级启动子区域(SPRs)这一新型超级增强子样元件,明确OsbZIP23作为关键整合因子介导干旱特异性启动子-启动子互作的形成,填补了干旱胁迫下植物3D基因组调控机制的空白,为表观遗传调控作物抗逆性提供了新的理论框架。
3. 研究思路总结与详细解析
本研究的核心目标是解析干旱胁迫及复水条件下水稻H3K9ac标记的3D染色质互作动态变化,揭示其调控干旱响应基因共表达的分子机制,明确关键调控因子。核心科学问题包括:干旱胁迫如何重塑水稻3D染色质结构?超级启动子区域如何整合启动子-启动子互作调控基因共表达?OsbZIP23在3D染色质互作动态调控中的作用机制是什么?技术路线遵循“样本处理→组学分析→机制解析→功能验证”的闭环逻辑:通过设置正常供水、干旱胁迫、复水三个处理组,结合ChIP-seq、ChIA-PET、RNA-seq等组学技术解析组蛋白修饰与3D基因组动态,通过突变体分析、ChIP-seq验证转录因子结合位点,最终通过原生质体实验验证功能,系统阐明干旱响应的3D基因组调控机制。
3.1 水稻H3K9ac修饰响应干旱胁迫的动态特征分析
实验目的:明确H3K9ac修饰在干旱及复水条件下的动态变化规律,及其与基因表达的相关性。
方法细节:选用明恢63(MH63)水稻幼苗,设置正常供水(NC)、干旱胁迫(DS,相对土壤含水量维持~5%)、复水(RE,恢复供水4天)三个处理组,每个处理设置2个生物学重复;采用增强型染色质免疫沉淀(eChIP)结合高通量测序(ChIP-seq)技术分析H3K9ac修饰的基因组分布,通过IDR(0.05)筛选高可信度修饰峰;同时通过RNA-seq分析对应处理组的基因表达谱,关联H3K9ac修饰强度与基因表达的变化。
结果解读:H3K9ac修饰峰主要分布在转录起始位点(TSS)近端启动子区域(-1000~+100bp),占比超70%(图1b);干旱胁迫下H3K9ac修饰峰的数量较正常条件增加,但平均跨度显著变窄(n=2,P<0.05),部分正常条件下的宽峰分裂为多个干旱特异性窄峰(图1d);H3K9ac修饰强度的变化与基因表达的Pearson相关系数在干旱胁迫下达到0.6以上(n=2,P<0.001),表明H3K9ac是响应水分变化的活性染色质标记(图1g)。
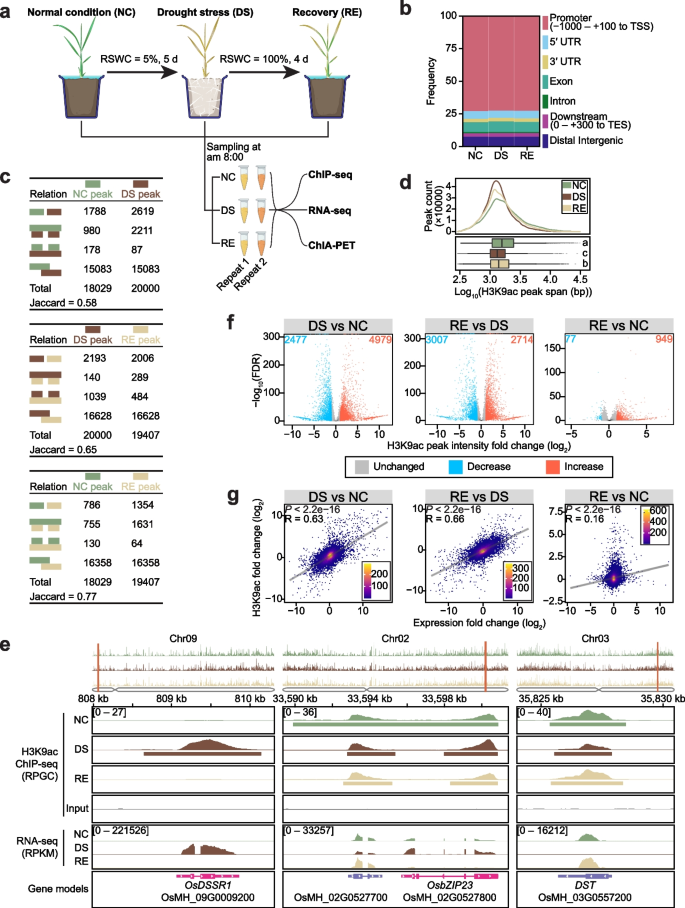
实验所用关键产品:抗H3K9ac抗体(Abcam, ab10812)、RNeasy Plant Mini Kit(QIAGEN, 74904)、TruSeq RNA kit(Illumina, 20020599)。
3.2 干旱及复水条件下H3K9ac标记的3D基因组动态解析
实验目的:解析干旱及复水条件下水稻3D染色质互作的动态变化特征,明确染色质互作与水分胁迫的关联。
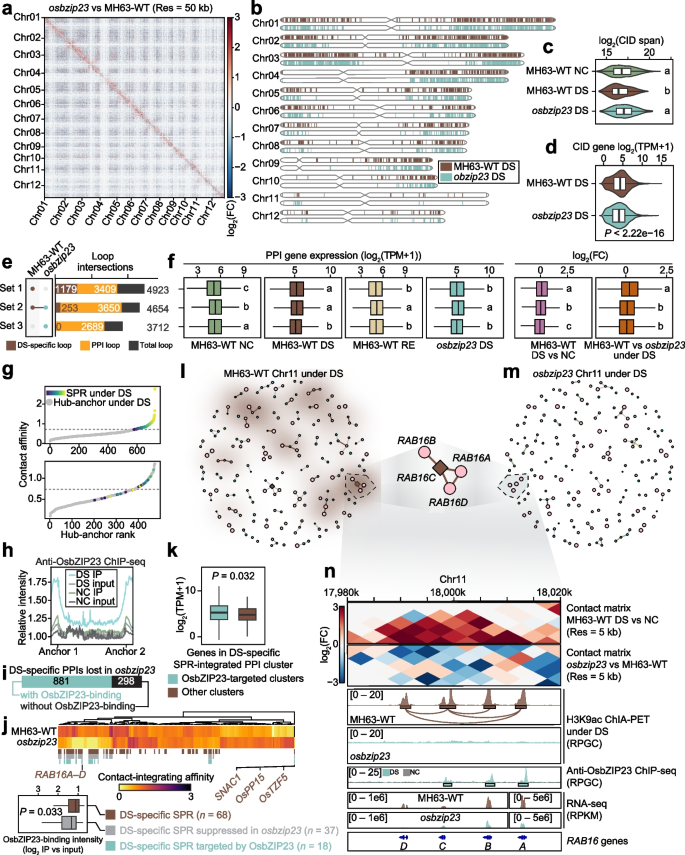
方法细节:采用长读长ChIA-PET技术,对三个处理组的H3K9ac标记的染色质互作进行高通量测序,以i-PET计数>6、FDR<0.05为标准筛选可信染色质环;基于连续连接的局部环构建染色质互作域(CIDs),分析染色质环数量、CIDs的基因组覆盖度与跨度、染色体间互作的变化;通过BL-Hi-C技术验证染色质环的可靠性,采用荧光原位杂交(FISH)技术验证染色体间互作的动态变化。
结果解读:正常条件下共鉴定到24235个H3K9ac标记的染色质环,干旱胁迫下仅存9577个,超10000个环在干旱胁迫下消失,复水后恢复至10626个(图2a、e);染色体间互作在干旱胁迫下大幅减少,超90%的染色体间环消失,复水后仅部分恢复(图2g、k);CIDs的总基因组覆盖度和平均跨度在干旱胁迫下显著降低(n=2,P<0.01),表明干旱胁迫导致染色质互作发生去浓缩(图2d、e)。
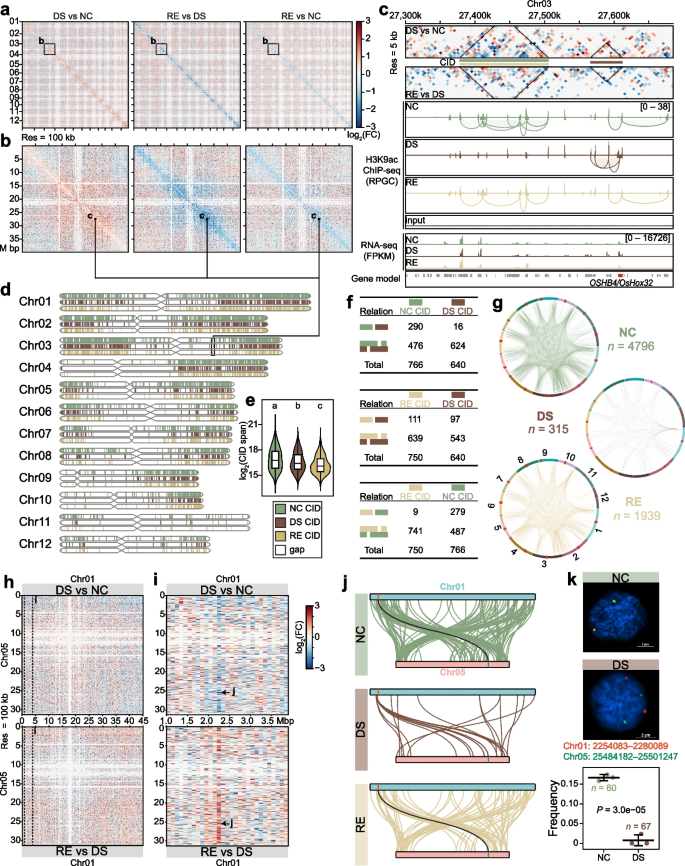
实验所用关键产品:抗H3K9ac抗体(Abcam, ab10812)、Dynabeads protein G beads(Life Technologies, 10003D)。
3.3 启动子-启动子互作(PPIs)调控干旱响应基因共表达的机制分析
实验目的:明确启动子-启动子互作在干旱响应基因表达调控中的作用,尤其是条件特异性互作的功能。
方法细节:将染色质环按锚定区域类型划分为启动子-启动子互作(PPIs)等类别,分析PPIs锚定基因的表达水平与仅H3K9ac修饰、无H3K9ac修饰基因的差异;通过Mfuzz聚类分析基因共表达模块,关联条件特异性PPIs与共表达模块的相关性;构建PPI网络,分析网络节点的连接度特征。
结果解读:PPIs锚定基因的表达水平显著高于仅H3K9ac修饰(非PPIs)和无H3K9ac修饰的基因(n=2,P<0.001),表明启动子-启动子互作能进一步增强H3K9ac修饰介导的基因激活(图3d);干旱特异性PPIs连接的基因显著富集于干旱响应共表达模块(模块2),该模块基因主要参与非生物胁迫响应(图3g);PPI网络中存在连接度显著高于其他节点的枢纽锚定区域,提示其在整合PPIs调控基因共表达中的核心作用(图3h)。
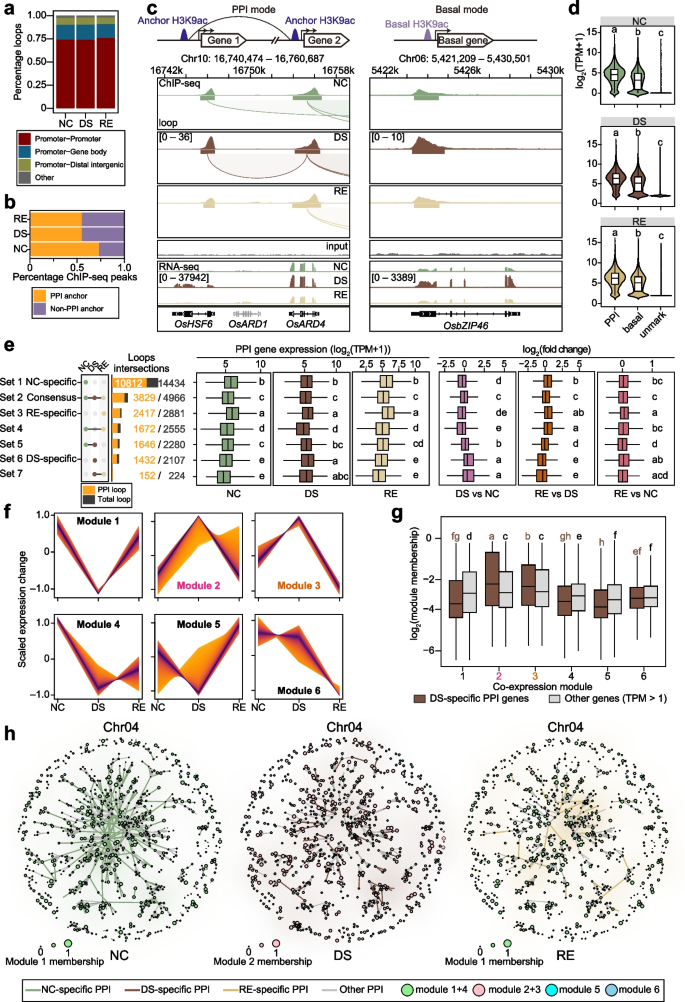
3.4 超级启动子区域(SPRs)的鉴定及功能分析
实验目的:鉴定整合启动子-启动子互作的超级启动子区域,明确其在干旱响应中的调控功能。
方法细节:采用随机游走算法将PPI网络划分为紧密连接的互作簇,每个簇中连接度最高的锚定区域定义为枢纽锚定区;计算枢纽锚定区的接触整合亲和力(CIA)值以消除锚定区域跨度的影响,将CIA值排名前20%的枢纽锚定区定义为超级启动子区域(SPRs);分析条件特异性SPRs的特征,并通过基序富集分析预测结合的转录因子。
结果解读:干旱条件下共鉴定到68个特异性SPRs,其整合的PPI簇中干旱特异性PPIs的比例及对应基因的表达变化幅度显著高于非特异性枢纽锚定区(n=2,P<0.05)(图4e);基序富集分析发现干旱特异性SPRs中存在bZIP、WRKY等干旱响应转录因子的结合位点(图4c),提示转录因子参与SPRs介导的PPIs调控。
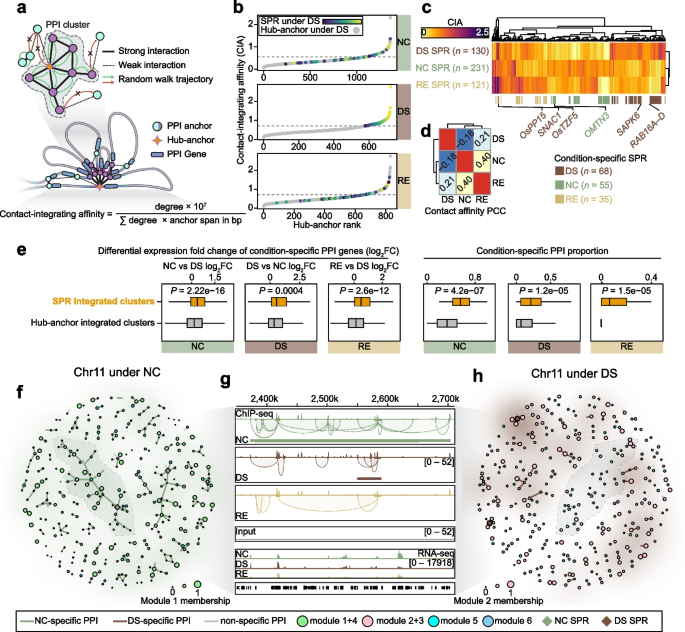
3.5 OsbZIP23调控干旱特异性PPIs的机制验证
实验目的:明确OsbZIP23在干旱特异性启动子-启动子互作形成中的作用及分子机制。
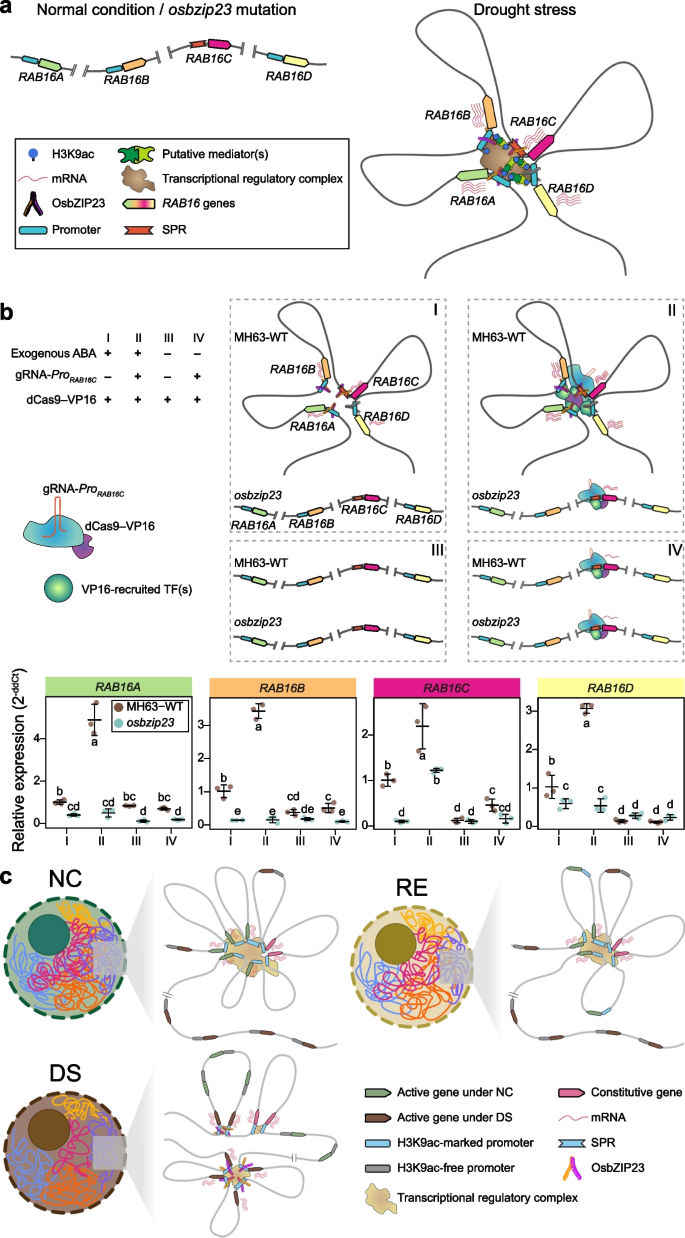
方法细节:对OsbZIP23 T-DNA突变体进行干旱胁迫处理,采用ChIA-PET技术分析其3D染色质互作图谱,并与野生型比较;通过ChIP-seq技术分析OsbZIP23在干旱胁迫下的基因组结合位点;采用dCas9-VP16激活系统在水稻原生质体中验证OsbZIP23对RAB16基因簇PPI簇形成及共表达的调控作用。
结果解读:OsbZIP23突变导致超80%的干旱特异性PPIs解离,对应基因的表达水平显著降低(n=2,P<0.01)(图5e、f);OsbZIP23在干旱胁迫下特异性结合于干旱PPIs的锚定区域,超75%的解离PPIs锚定区存在OsbZIP23的结合位点(图5h、i);原生质体实验显示,仅在OsbZIP23存在且ABA处理(模拟干旱信号)时,靶向RAB16C SPR的dCas9-VP16才能同时激活四个RAB16基因的表达,表明OsbZIP23介导的PPI簇是基因共表达的结构基础(图6b)。
实验所用关键产品:抗OsbZIP23多克隆抗体(前期实验验证)、dCas9-VP16载体系统。
4. Biomarker研究及发现成果
Biomarker定位
本研究中鉴定的超级启动子区域(SPRs)属于表观遗传及3D基因组层面的调控型Biomarker,其筛选与验证逻辑为:基于H3K9ac标记的启动子-启动子互作网络,通过随机游走算法划分紧密连接的互作簇,计算枢纽锚定区的接触整合亲和力(CIA)值,筛选CIA值排名前20%的区域定义为SPRs;通过与正常、复水条件比较,筛选出CIA值在干旱下较其他条件高1.5倍以上的干旱特异性SPRs;最终通过ChIP-seq验证转录因子结合位点、原生质体实验验证其调控基因共表达的功能。
研究过程详述
SPRs的分子来源为水稻幼苗染色质互作数据中的启动子区域锚定区,验证方法包括:BL-Hi-C技术验证其整合的PPI簇的可靠性,ChIP-seq技术验证干旱响应转录因子(如OsbZIP23)的结合位点,dCas9-VP16激活系统验证其调控基因共表达的功能;干旱特异性SPRs的特异性表现为:其CIA值在干旱胁迫下较正常和复水条件显著升高1.5倍以上,对应的PPI簇中干旱特异性PPIs的比例显著高于非特异性SPRs(n=2,P<0.05),且富集干旱响应转录因子的结合位点。
核心成果提炼
超级启动子区域(SPRs)作为新型超级增强子样元件,能够整合干旱响应基因的启动子-启动子互作形成簇状结构,调控基因的共表达;OsbZIP23作为核心调控因子,特异性结合于干旱特异性SPRs,介导干旱特异性PPI簇的形成,是干旱胁迫下3D染色质互作动态及基因共表达的关键整合因子。其中,RAB16C SPR介导的PPI簇调控RAB16A-D四个脱水素基因的共表达,是水稻干旱响应的关键功能模块,OsbZIP23突变后该PPI簇完全解离,四个基因的表达水平显著降低(n=2,P<0.01)。本研究鉴定的SPRs及OsbZIP23为水稻抗旱育种提供了全新的表观遗传调控靶点,为精准调控作物抗逆性奠定了理论基础。
