1. 领域背景与文献引入
文献英文标题:Nanobiomotors of archaeal DNA repair machineries: current research status and application potential;发表期刊:Cell & Bioscience;影响因子:未公开;研究领域:古菌分子生物学、DNA修复纳米生物马达
领域共识:纳米生物马达是细胞内关键的“分子机器”,通过ATP结合与水解将化学能转化为机械运动,参与DNA修复、复制、重组等核心生命过程,其能量转化机制是分子生物学的核心研究方向之一;古菌作为三域生物中的特殊类群,与真核生物在信息加工机制(包括DNA修复、转录、翻译)上具有高度保守性,且超嗜热古菌编码的蛋白具有极端环境稳定性,是研究纳米生物马达的理想模型。当前领域研究热点集中于解析纳米生物马达的分子运动机制,以及开发其在纳米医学(如药物递送)中的应用潜力,但仍存在未解决的核心问题:古菌部分DNA修复纳米马达的生理功能存在争议(如XPB/XPD是否真的参与核苷酸切除修复),HerA的详细运动机制、RadA旁系同源物的具体调控机制尚未明确,且马达在纳米医学中的应用研究仅停留在理论探讨阶段。针对这些研究空白,本文系统综述了古菌DNA修复领域各类纳米生物马达的结构特征、分子运动机制,并探讨了其在纳米生物技术中的应用潜力,为该领域的基础研究与应用开发提供了全面的指导框架。
2. 文献综述解析
本文以纳米生物马达的类型为核心分类维度,系统梳理了古菌DNA修复领域中SF2家族解旋酶、FtsK-HerA超家族HerA解旋酶、RecA/RadA家族重组酶及RadA旁系同源物的研究进展,明确了各类马达的结构特征、分子机制及应用前景。
现有研究已通过结构生物学与生化实验结合,揭示了SF2家族XPB、XPD、Hjm解旋酶的晶体结构与ATP驱动的构象变化模式,RecA/RadA家族重组酶的多构象变化与链交换机制,以及RadA旁系同源物RadB、RadC的初步结构与功能;HerA被证实形成六聚体环结构,与Mre11-Rad50、NurA协同参与DNA双链断裂末端加工,但详细运动机制尚未明确。现有研究的优势在于从分子层面揭示了纳米生物马达“化学能-机械能”转化的核心基础,为理解古菌DNA修复机制提供了关键线索;局限性在于部分马达的生理功能存在争议,HerA的详细运动机制、RadA旁系同源物的具体调控机制仍不清晰,且应用方面的研究仅停留在理论探讨阶段,缺乏实际应用验证。
本文的创新价值在于首次系统整合了古菌DNA修复领域各类纳米生物马达的研究进展,填补了该领域系统综述的空白;同时将马达的结构机制与纳米医学应用潜力相结合,提出利用古菌纳米马达的高稳定性与主动运动能力开发药物递送载体的设想,为后续的基础研究与应用开发提供了全面的指导框架。
3. 研究思路总结与详细解析
本文为综述性研究,核心目标是系统总结古菌DNA修复纳米生物马达的结构特征、分子运动机制及应用潜力,核心科学问题是阐明这些马达如何通过ATP结合与水解实现构象变化,进而驱动DNA修复相关的机械运动,技术路线为按马达类型分类梳理已发表的结构生物学、生化实验及遗传学研究结果,结合分子模型探讨运动机制,最后展望其在纳米生物技术中的应用前景。
3.1 SF2家族DNA修复解旋酶的结构与机制解析
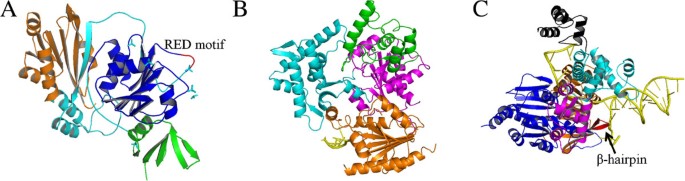
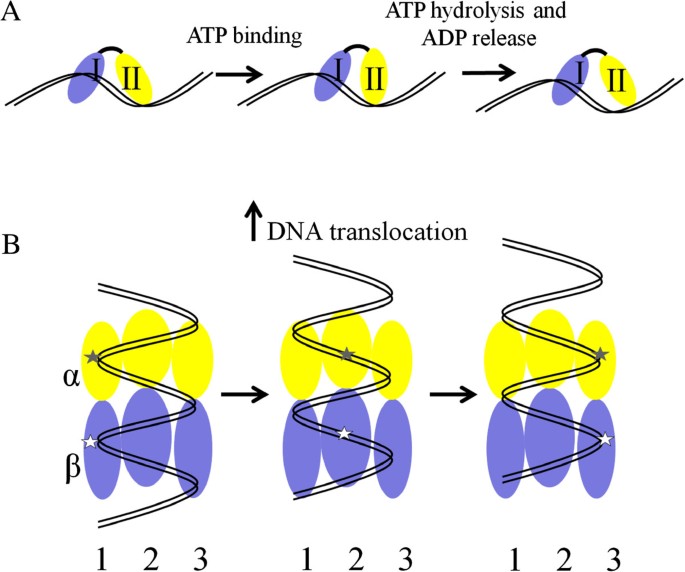
实验目的是明确古菌SF2家族解旋酶(XPB、XPD、Hjm)的结构特征、DNA结合与解旋的分子机制。方法细节包括采用X射线晶体衍射技术解析解旋酶的晶体结构(如嗜热古菌AfuXPB、TaXPD的晶体结构),通过生化实验检测ATP酶活性、DNA解旋活性,结合分子动力学模拟探讨构象变化与DNA运动的关联。结果解读:AfuXPB包含两个RecA折叠的ATPase结构域(HD1、HD2)及DNA损伤识别域(DRD),ATP结合后两个ATPase结构域形成闭合构象,水解ATP驱动结构域旋转,RED基序插入双链DNA实现解旋(
),该过程符合旋转运动模型(
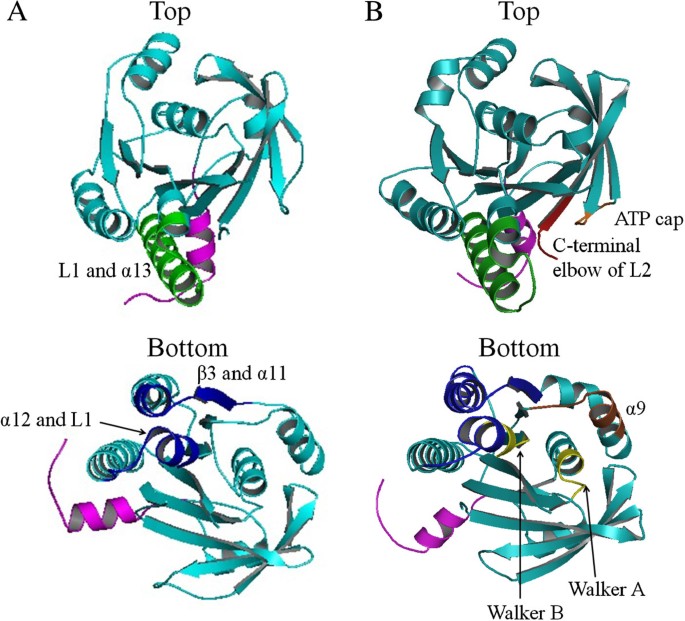
);TaXPD包含4FeS和Arch结构域,结合单链DNA但解旋位点未明确,且遗传学研究显示古菌XPD可能不参与经典的核苷酸切除修复,其生理功能有待进一步验证;Hjm解旋酶能通过ATP驱动的构象变化解旋Holliday junction,还能结合3"单链DNA重启停滞的复制叉,其运动机制同样依赖ATP结合与水解的构象变化。产品关联:文献未提及具体实验产品,领域常规使用X射线晶体衍射仪、重组蛋白表达纯化试剂盒、ATP酶活性检测试剂盒、DNA底物合成试剂等。
3.2 HerA解旋酶的结构与功能分析
实验目的是探讨HerA在古菌DNA双链断裂末端加工中的作用机制及运动模式。方法细节包括采用生化实验分析HerA与Mre11-Rad50、NurA的相互作用,通过电镜观察HerA的多聚体结构,结合同源建模推测其运动机制。结果解读:HerA属于FtsK-HerA超家族,形成六聚体环结构,与Mre11-Rad50、NurA协同作用,参与DNA双链断裂末端的加工,将双链DNA转化为3"突出端;推测:基于与细菌FtsK的同源性,推测HerA可能采用旋转-尺蠖运动模型(

),但目前尚未解析其晶体结构,详细运动机制仍不明确。产品关联:文献未提及具体实验产品,领域常规使用透射电子显微镜、蛋白-蛋白相互作用检测试剂盒、DNA末端加工活性检测体系等。
3.3 RadA重组酶的构象变化与链交换机制
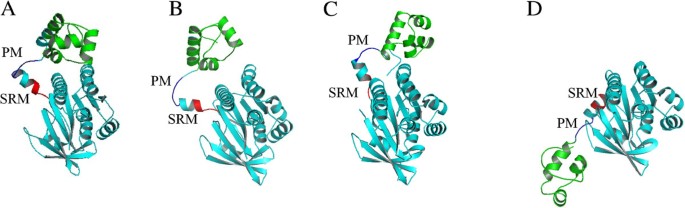
实验目的是揭示RadA介导同源重组的分子机制,明确ATP结合水解与构象变化的关联。方法细节包括采用X射线晶体衍射技术解析不同核苷酸结合状态下RadA的多聚体结构(环、右手丝状体、过旋右手丝状体、左手丝状体),通过生化实验检测ATP酶活性、DNA链交换活性,分析金属离子对RadA构象与活性的调控作用。结果解读:RadA在无ATP时形成环结构(用于储存以减少ATP消耗),ATP结合后形成右手丝状体,结合单链DNA形成核蛋白丝;ATP水解驱动亚基间发生约30°的旋转,通过亚基旋转基序(SRM)的构象变化实现丝状体的构象转换(
),进而介导链交换形成D-loop结构;金属离子(Mg²+、Ca²+、K+)通过稳定ATP结合位点或DNA结合环(L1、L2)的构象,调控RadA的ATP酶活性与丝状体稳定性(
)。产品关联:文献未提及具体实验产品,领域常规使用X射线晶体衍射仪、DNA链交换检测试剂盒、金属离子浓度梯度实验体系等。
3.4 RadA旁系同源物的功能分析
实验目的是明确RadB、RadC等RadA旁系同源物在古菌DNA修复中的作用机制。方法细节包括采用X射线晶体衍射技术解析RadB、RadC的晶体结构,通过生化实验检测其ATP酶活性、DNA结合能力,以及与RadA的相互作用,结合遗传学实验分析其在DNA修复中的功能。结果解读:RadB形成二聚体结构,ATP结合后构象变化增强其DNA结合活性,但ATP酶活性较弱;RadC形成六聚体环结构,部分RadC成员(如Sulfolobus的RadC1)能增强RadA的ATP酶活性与链入侵活性,RadC2则与RadA和Holliday junction resolvase Hjc相互作用,参与同源重组过程;遗传学实验显示RadC1、RadC2在古菌DNA损伤修复中发挥不同的调控作用。产品关联:文献未提及具体实验产品,领域常规使用基因敲除技术、蛋白相互作用分析试剂盒、DNA损伤修复检测体系等。
4. Biomarker研究及发现成果解析
本文未涉及疾病诊断、预后或治疗相关的传统生物标志物(Biomarker)研究,而是聚焦于古菌DNA修复纳米生物马达的结构与功能特征,这些马达可作为纳米生物技术中的功能分子靶点,其核心结构域与活性位点可视为“功能Biomarker”,用于指导纳米材料的设计与开发。
Biomarker定位
本文中的功能分子靶点包括纳米生物马达的RecA折叠ATPase结构域、DNA结合域(如XPB的DRD域、RadA的L1/L2环),筛选逻辑基于古菌与真核生物DNA修复机制的保守性,以及古菌蛋白的高稳定性(超嗜热古菌的蛋白在极端条件下仍保持活性),通过结构生物学解析核心功能域,结合生化实验验证其“化学能-机械能”转化的核心活性,形成“保守性筛选-结构解析-功能验证”的完整逻辑链条。
研究过程详述
这些功能分子靶点来源于古菌细胞内的DNA修复蛋白,通过基因克隆、重组表达获得纯化蛋白,采用X射线晶体衍射技术解析其三维结构,明确核心功能域的空间构象;通过生化实验检测ATP酶活性、DNA结合/解旋/链交换活性,验证其功能活性;特异性方面,不同类型的纳米生物马达具有独特的结构特征与功能特异性(如XPB识别损伤DNA、HerA加工双链断裂末端),敏感性方面,ATP酶活性检测显示其能响应ATP浓度变化实现构象转换,但具体的量化数据(如Km值、Vmax)因不同马达而异,文献未统一提供。
核心成果提炼
这些功能分子靶点的核心价值在于揭示了古菌纳米生物马达“化学能-机械能”转化的核心分子机制,为纳米医学中的药物递送载体设计提供了理论基础;创新性在于首次系统整合了古菌DNA修复纳米生物马达的结构与功能研究进展,提出利用古菌纳米马达的高稳定性与主动运动能力开发药物递送载体的设想;由于本文为综述性研究,未提供具体的统计学结果(如P值、样本量),相关数据可参考引用的原始研究文献。
