1. 领域背景与文献引入
文献英文标题:Physiological and transcriptome analyses of Opisthopappus taihangensis in response to drought stress;发表期刊:Cell & Bioscience;影响因子:未公开;研究领域:植物逆境生理学(干旱胁迫响应)
干旱是全球范围内影响植物生长发育与农业生产的核心非生物胁迫因子,随着气候变化加剧,水资源短缺问题日益严峻,解析植物的干旱响应机制对培育耐旱作物、保护濒危耐旱植物具有重要战略意义。领域共识:植物通过感知干旱信号,启动多层次的适应机制,包括生理层面积累渗透调节物质维持细胞渗透压、分子层面调控转录因子与功能基因表达,最终实现对胁迫的耐受。当前研究多聚焦于拟南芥、水稻等模式植物的干旱响应机制,已揭示了MYB、bHLH等转录因子家族的调控作用及脯氨酸等渗透调节物质的功能,但针对中国特有濒危耐旱植物的研究较为匮乏。太行菊是仅分布于太行山区的二级保护濒危植物,具有极强的自然耐旱性,但其适应干旱胁迫的生理与分子机制尚未明确,本研究通过生理指标检测与高通量转录组测序结合的方法,系统解析太行菊对不同程度干旱的响应机制,填补了该物种研究的空白,为其保护及耐旱基因资源的挖掘提供了科学依据。
2. 文献综述解析
作者从植物干旱响应的生理调节机制、分子调控网络、转录组学研究技术三个维度对领域现有研究进行系统梳理,明确了模式植物中干旱响应的核心通路,但指出特有濒危耐旱植物研究的关键缺口。现有研究表明,植物在干旱胁迫下会通过积累脯氨酸、甜菜碱等渗透调节物质维持细胞水分平衡,通过MYB、bHLH、WRKY等转录因子家族调控下游功能基因的表达,从而实现对胁迫的适应;转录组学技术已广泛应用于模式植物的干旱响应研究,揭示了大量差异表达基因与调控通路,但这些研究多集中于常见作物或模式物种,针对太行菊这类具有独特生境适应性的濒危植物,尚未开展系统的干旱响应机制研究,缺乏对其特有耐旱基因资源的挖掘。本研究的创新点在于首次将生理表型分析与高通量转录组测序技术结合,针对不同程度干旱胁迫下的太行菊进行系统研究,鉴定出的干旱响应基因与转录因子,不仅填补了太行菊研究的空白,也为濒危植物保护及耐旱作物育种提供了新的基因资源。
3. 研究思路总结与详细解析
本研究以中国特有濒危耐旱植物太行菊为材料,通过PEG6000模拟轻度、重度两种程度的干旱胁迫,结合生理指标检测、转录组测序、功能富集分析及qRT-PCR验证,系统揭示太行菊适应干旱胁迫的生理与分子机制,整体研究遵循“胁迫处理→生理表型分析→转录组测序→功能通路解析→实验验证”的逻辑闭环。
3.1 干旱胁迫处理与生理指标检测
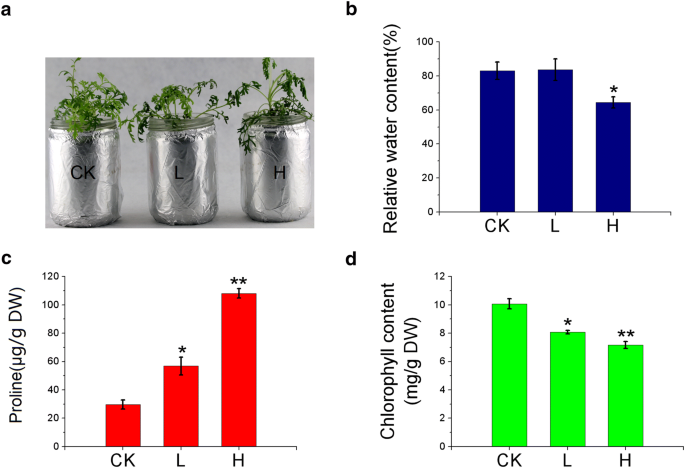
实验目的:明确太行菊对不同程度干旱胁迫的生理响应特征,筛选核心生理适应指标。方法细节:选取30天苗龄的太行菊植株,分别移栽至含0%(对照组CK)、5%(轻度胁迫组L)、25%(重度胁迫组H)PEG6000的培养基中,每个处理设置3个生物学重复,处理2天后采集新鲜叶片样本,采用烘干称重法检测叶片相对含水量(RWC)、比色法检测脯氨酸含量、乙醇-丙酮混合液提取后分光光度法检测叶绿素含量。结果解读:重度胁迫下太行菊植株出现明显萎蔫(图1a),轻度胁迫下植株形态与对照组无显著差异;相对含水量在轻度胁迫下与对照组无显著差异,重度胁迫下显著降低16.7%(n=3,P<0.01)(图1b);脯氨酸含量随胁迫程度增加显著积累,重度胁迫下含量为对照组的3.2倍(n=3,P<0.01)(图1c);叶绿素含量随胁迫程度增加略有下降,但无统计学差异(n=3,P>0.05)(图1d),表明太行菊通过积累脯氨酸维持细胞渗透压,在轻度干旱下可保持正常水分状态以适应胁迫。实验所用关键产品:OMEGA的Plant RNA Kit、Shimadzu分光光度计、Roche的LightCycler Multiplex DNA Master Kit等。
3.2 转录组测序与差异表达基因分析
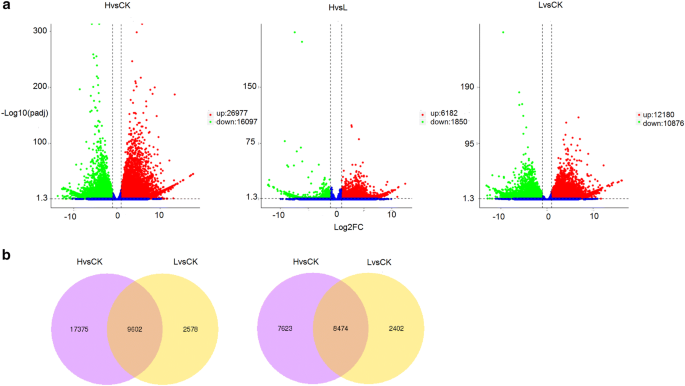
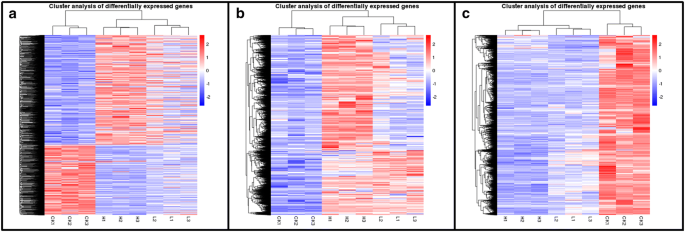
实验目的:鉴定太行菊响应不同程度干旱胁迫的差异表达基因(DEGs),明确分子层面的响应特征。方法细节:提取对照组、轻度胁迫组、重度胁迫组叶片的总RNA,构建RNA-seq文库后采用Illumina HiSeq 2500平台测序,以拟南芥基因组为参考序列进行比对,使用DESeq R包进行差异表达分析,筛选阈值为|log2(倍数变化)|>1且校正后P值<0.005。结果解读:对照组与轻度胁迫组间共鉴定出23056个DEGs,其中12180个上调、10876个下调;轻度胁迫组与重度胁迫组间共鉴定出8032个DEGs,其中6182个上调、1850个下调;对照组与重度胁迫组间共鉴定出43074个DEGs,其中26977个上调、16097个下调(图2);层次聚类分析显示,差异基因的表达水平随胁迫程度加剧呈现显著的梯度变化,上调与下调基因的聚类特征均与胁迫程度高度相关(图3),表明太行菊通过调控大量基因的表达水平,实现对不同程度干旱胁迫的精准适应。
3.3 差异基因功能富集分析
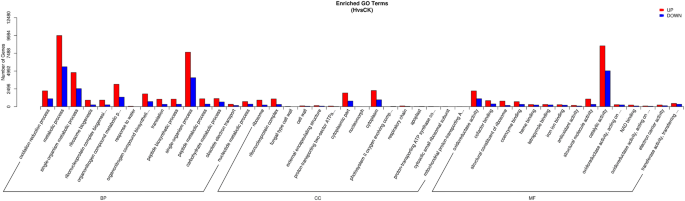
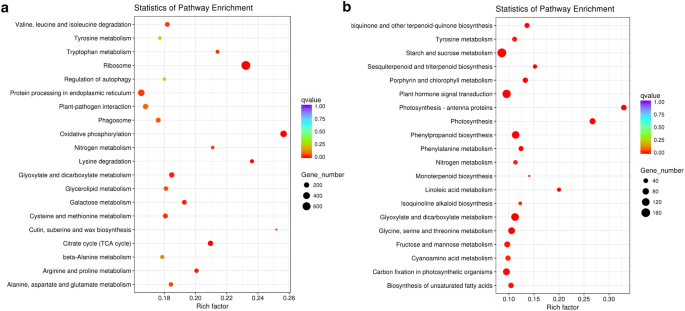
实验目的:解析差异表达基因参与的生物学过程与代谢通路,明确太行菊干旱响应的核心分子通路。方法细节:选取对照组与重度胁迫组间的DEGs进行GO(基因本体)和KEGG(京都基因与基因组百科全书)富集分析,GO富集使用GOseq R包基于非中心超几何分布计算,KEGG富集基于KEGG数据库的基因注释信息。结果解读:GO富集显示,DEGs主要富集在“代谢过程”“催化活性”“单生物过程”等功能条目,且上调基因数量显著多于下调基因(图4);KEGG富集显示,上调DEGs主要富集在氨基酸代谢通路,包括半胱氨酸和甲硫氨酸代谢、精氨酸和脯氨酸代谢等,下调DEGs主要富集在淀粉和蔗糖代谢通路(图5),表明太行菊通过增强氨基酸代谢(尤其是脯氨酸合成相关通路)调节细胞渗透压,同时下调碳水化合物代谢以减少能量消耗,从而适应干旱胁迫。
3.4 转录因子鉴定与分析
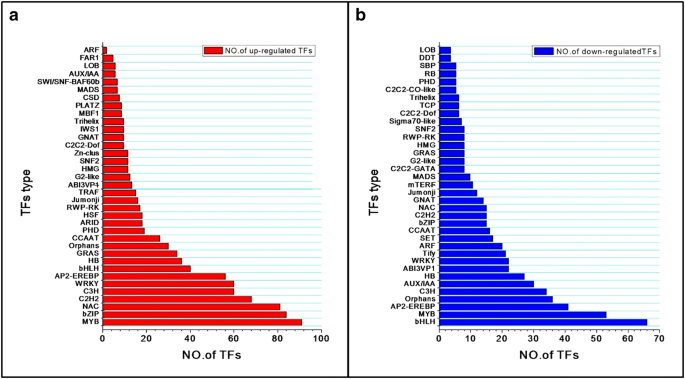
实验目的:鉴定参与太行菊干旱响应的转录因子(TFs),明确核心调控因子家族及表达模式。方法细节:将差异表达基因与拟南芥转录因子数据库进行比对,筛选出差异表达的转录因子,分析其家族分布及随胁迫程度的表达变化模式。结果解读:对照组与重度胁迫组间共鉴定出931个上调转录因子,分属36个家族,其中MYB、bZIP、NAC家族占比最高;614个下调转录因子分属36个家族,其中bHLH、MYB、AP2-EREBP家族占比最高(图6);进一步分析发现,39个转录因子随干旱胁迫程度增加持续上调,13个转录因子持续下调,这些转录因子可能是太行菊干旱响应的核心调控因子,其中MYB、bHLH等家族成员的表达模式与模式植物中的干旱响应调控机制一致。
3.5 qRT-PCR验证转录组结果
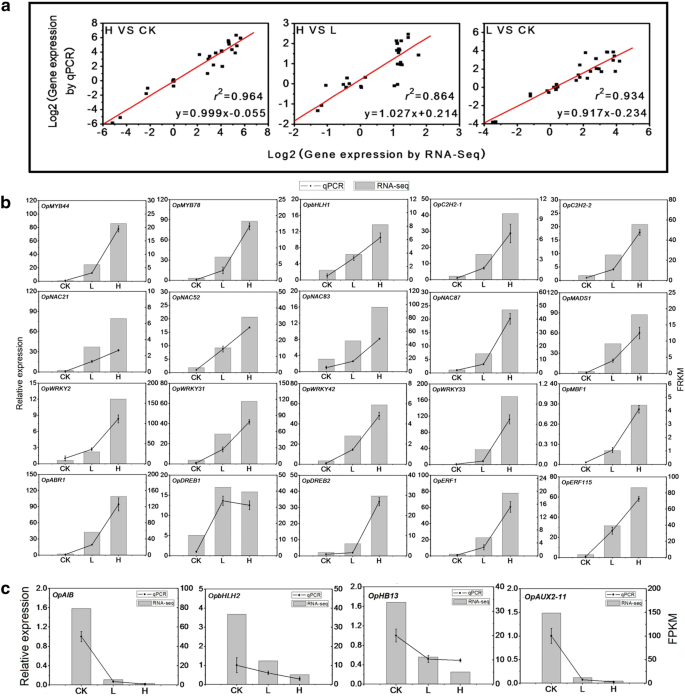
实验目的:验证转录组测序结果的可靠性,确认核心干旱响应基因的表达模式。方法细节:选取32个差异表达的核心基因(包括MYB、bHLH、C2H2等家族成员),使用Primer 5软件设计特异性引物,采用Roche的LightCycler 480实时荧光定量PCR系统进行qRT-PCR检测,每个样本设置3个生物学重复,采用2^-△△CT法计算相对表达量。结果解读:qRT-PCR结果与转录组数据的相关性系数在对照组vs重度胁迫组、重度胁迫组vs轻度胁迫组、轻度胁迫组vs对照组分别为0.964、0.864、0.934(图7a),表明转录组数据具有较高的可靠性;多数转录因子(如OpMYB44、OpbHLH1)随胁迫程度增加显著上调,少数基因(如OpDREB1)在重度胁迫下略有下调(图7b、c),进一步验证了转录组分析的结果。
4. Biomarker研究及发现成果
本研究鉴定的干旱响应Biomarker涵盖生理与分子两个层面,通过“生理表型筛选→转录组测序鉴定→qRT-PCR验证”的完整逻辑链条,明确了这些Biomarker在太行菊干旱响应中的功能与调控作用,为干旱胁迫的检测及耐旱基因资源的挖掘提供了依据。
Biomarker定位:生理层面的核心Biomarker为脯氨酸,分子层面的核心Biomarker为39个持续上调的转录因子及氨基酸代谢通路中的关键基因;筛选逻辑为:首先通过生理指标检测发现脯氨酸含量随干旱胁迫显著积累,可作为胁迫程度的生理指示物;然后通过转录组测序从全基因组水平鉴定出差异表达的基因与转录因子,筛选出随胁迫程度持续变化的核心分子Biomarker;最后通过qRT-PCR验证核心Biomarker的表达模式,确认其可靠性。
研究过程详述:脯氨酸来源于太行菊叶片样本,采用比色法定量检测,结果显示其含量随PEG浓度增加显著升高,重度胁迫下含量为对照组的3.2倍(n=3,P<0.01);分子Biomarker通过RNA-seq从叶片转录组中筛选,qRT-PCR验证显示,OpMYB44在轻度和重度胁迫下的相对表达量分别为对照组的2.1倍和4.5倍(n=3,P<0.01),OpbHLH1分别为1.8倍和3.7倍(n=3,P<0.01);原文未提及ROC曲线分析等特异性与敏感性相关数据,故无对应结果。
核心成果提炼:脯氨酸作为渗透调节型Biomarker,可有效反映太行菊的干旱胁迫程度,其积累与精氨酸和脯氨酸代谢通路中关键基因(如Arg1、OAT)的上调表达直接相关;39个持续上调的转录因子(如MYB、bHLH家族成员)作为调控型Biomarker,参与太行菊干旱响应的信号转导与基因表达调控,是干旱胁迫响应的核心调控节点;本研究首次在太行菊中鉴定出这些干旱响应Biomarker,揭示了其独特的干旱适应机制,为濒危植物的保护及耐旱作物的分子育种提供了重要的基因资源,原文未报道额外的统计学结果(如风险比HR等)。
