1. 领域背景与文献引入
文献英文标题:Rational design of promoter editing confers multipathogen resistance in rice;发表期刊:Genome Biology;影响因子:17.906(2024年);研究领域:作物分子生物学、植物免疫与抗病育种。
水稻作为全球超过半数人口的主粮,其产量稳定直接关系到粮食安全,但稻瘟病、白叶枯病、纹枯病等病害每年造成巨大产量损失,仅稻瘟病每年减产即可供养6000万人。传统抗病育种策略如R基因导入、基因堆叠虽能获得抗性,但病原菌的快速进化易导致抗性失效;病斑模拟突变体(Lesion Mimic Mutants, LMM)如rbl1等可激活植物基础免疫,实现广谱抗病,但往往伴随生长抑制和产量损失,难以直接应用于生产。近年来,启动子编辑作为精准调控基因表达的技术,为解决性状权衡问题提供了新途径,但由于启动子顺式调控元件的复杂性,编辑结果的可预测性差,缺乏系统性的优化框架。针对这一空白,本研究以水稻LMM基因RBL1为靶标,通过整合染色质可及性分析、生物信息学预测和功能验证,建立可预测的启动子编辑体系,精准调控RBL1表达水平,成功打破免疫-生长权衡,获得广谱抗病且无产量损失的水稻材料,为作物抗病育种提供了新的技术范式。
2. 文献综述解析
作者的综述逻辑以“水稻病害防控需求→现有策略的局限→启动子编辑的潜力与挑战→RBL1基因的研究基础”为脉络,层层递进地引出研究问题。现有研究可分为三类:一是传统R基因抗病育种,其优势是直接获得对特定病原菌的抗性,技术成熟度高,但缺陷是抗性易因病原菌的进化突变而快速丧失,且难以实现广谱抗病;二是LMM突变体的应用,这类材料通过激活植物基础免疫实现广谱抗病,但普遍存在免疫过度激活导致的生长抑制和产量损失,如rbl1突变体虽能抗多种病害,但植株生长缓慢、结实率降低;三是启动子编辑技术的应用,该技术通过精准修饰基因启动子区域实现基因表达的精细调控,已在水稻粒形、番茄心室数等性状改良中取得成功,但由于启动子调控元件的复杂性,编辑结果难以预测,缺乏可重复的系统性设计方法。
本研究的创新价值在于,首次将染色质可及性分析、生物信息学预测与原生质体功能验证相结合,建立了可预测的启动子编辑框架,解决了LMM基因应用中免疫-生长权衡的核心问题。与传统的随机启动子编辑相比,本研究通过提前鉴定关键调控区域,大幅减少了筛选工作量;与直接编辑编码区获得的RBL1Δ12等位基因相比,本研究获得的PRO1等位基因仅调控基因表达水平,保留了完整的蛋白序列,避免了蛋白截短可能带来的未知功能影响,为LMM基因的精准应用提供了新的思路。
3. 研究思路总结与详细解析
本研究的整体目标是通过可预测的启动子编辑技术,精准调控水稻LMM基因RBL1的表达水平,打破免疫-生长权衡,获得广谱抗病且产量不受影响的水稻材料;核心科学问题是如何系统鉴定RBL1启动子的关键调控区域,实现基因表达的精细调控;技术路线遵循“预测→验证→编辑→表型→机制”的闭环逻辑:首先通过生物信息学分析预测RBL1启动子的核心调控区域,然后在水稻原生质体中验证不同区域的调控功能,接着利用CRISPR-Cas9技术获得不同表达水平的启动子编辑株系,随后进行抗病性和农艺性状鉴定筛选最优株系,最后通过多组学分析解析其抗病机制,并在田间试验和不同品种中验证策略的适用性。
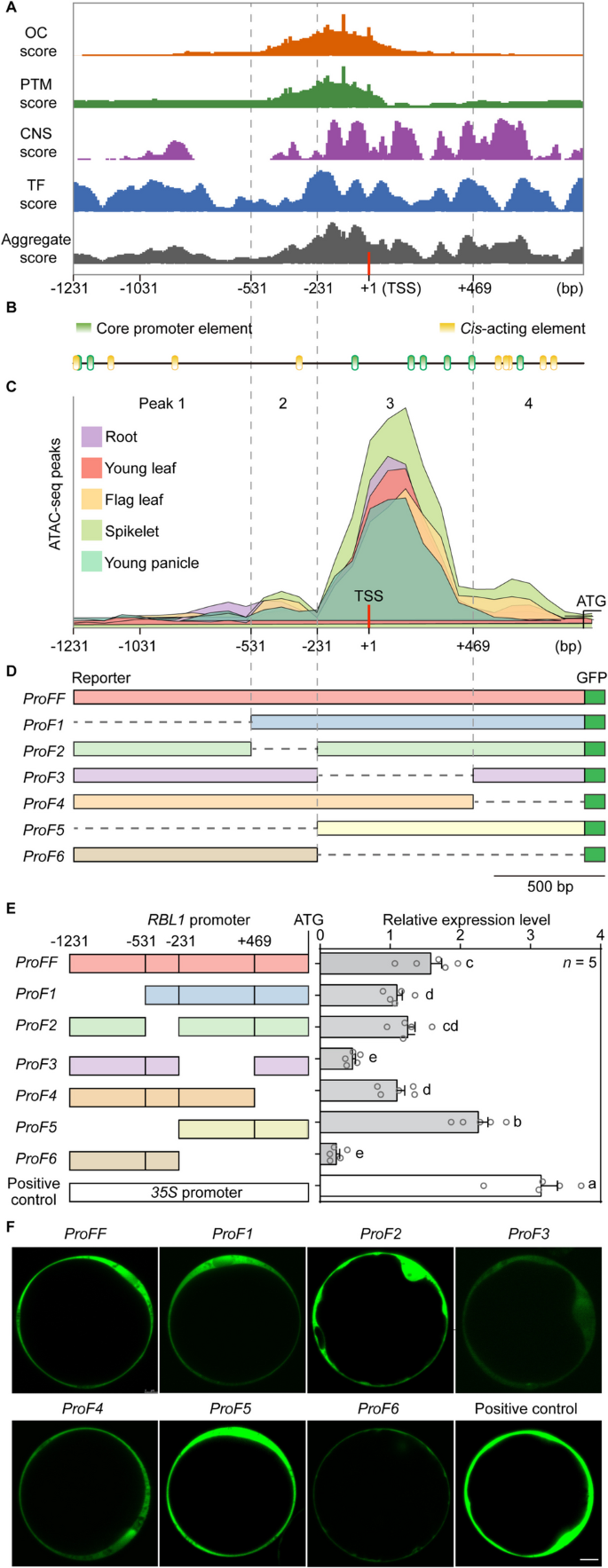
3.1 RBL1启动子顺式调控区域的生物信息学预测
实验目的:系统鉴定RBL1启动子中调控基因表达的关键顺式调控区域,为后续启动子编辑提供靶点。
方法细节:通过5" RACE技术确定RBL1的转录起始位点(TSS),整合ATAC-seq、DNase-seq、ChIP-seq等公共数据库的染色质可及性数据,结合PlantDeepSEA、PlantCARE等生物信息学工具,分析启动子区域的开放染色质峰、转录因子结合位点、核心启动子元件和顺式作用元件,最终将RBL1的2.2kb启动子区域划分为4个峰区域。
结果解读:分析结果显示,TSS附近的峰3(-231至+468 bp)是核心调控区域,该区域具有最高的染色质可及性,富集了大量转录因子结合位点和保守非编码序列,PlantDeepSEA的饱和突变分析显示该区域的突变会显著影响启动子活性;PlantCARE分析也证实该区域包含核心启动子元件和多个顺式作用元件。
产品关联:文献未提及具体实验产品,领域常规使用MACS2、FIMO、PlantDeepSEA等生物信息学工具进行染色质数据分析与顺式元件预测。
3.2 水稻原生质体中启动子区域调控功能验证
实验目的:在原生质体系统中验证不同启动子区域对RBL1表达的调控作用,验证生物信息学预测结果的可靠性。
方法细节:构建包含不同启动子区域缺失的GFP报告载体(ProF1-Pro6,分别缺失峰1-峰4的不同组合),以全长启动子(ProFF)为对照,通过PEG介导的转化方法将载体导入水稻原生质体,培养18h后通过荧光显微镜观察GFP荧光信号,同时利用实时荧光定量PCR(qRT-PCR)定量GFP的表达水平。
结果解读:荧光信号和qRT-PCR结果显示,缺失峰3的ProF3载体的GFP表达水平较ProFF降低70.7%(n=5,P<0.05),缺失峰3+峰4的ProF6载体降低85.3%(n=5,P<0.05),而缺失峰1+峰2的ProF5载体的GFP表达水平升高43.1%(n=5,P<0.05),证明峰3是RBL1启动子的关键正调控区域,峰1和峰2可能包含负调控元件。
产品关联:文献未提及具体实验产品,领域常规使用pM999-eGFP等报告载体,PEG介导的原生质体转化体系,qRT-PCR试剂如ChamQ Universal SYBR qPCR Master Mix等。
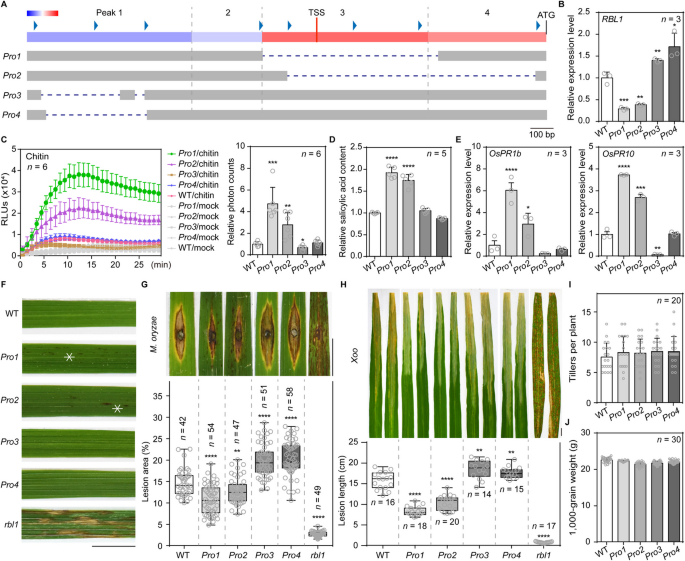
3.3 CRISPR-Cas9介导的RBL1启动子编辑水稻株系构建
实验目的:获得不同RBL1表达水平的启动子编辑水稻株系,为后续表型鉴定提供材料。
方法细节:设计8个靶向RBL1启动子4个峰区域的向导RNA(gRNA),构建两个CRISPR-Cas9载体(每个载体包含4个gRNA),通过农杆菌介导的转化方法导入水稻品种Kitaake,获得T0代植株后,通过Sanger测序鉴定编辑类型,筛选无转基因的纯合T1株系,并利用qRT-PCR测定RBL1的表达水平。
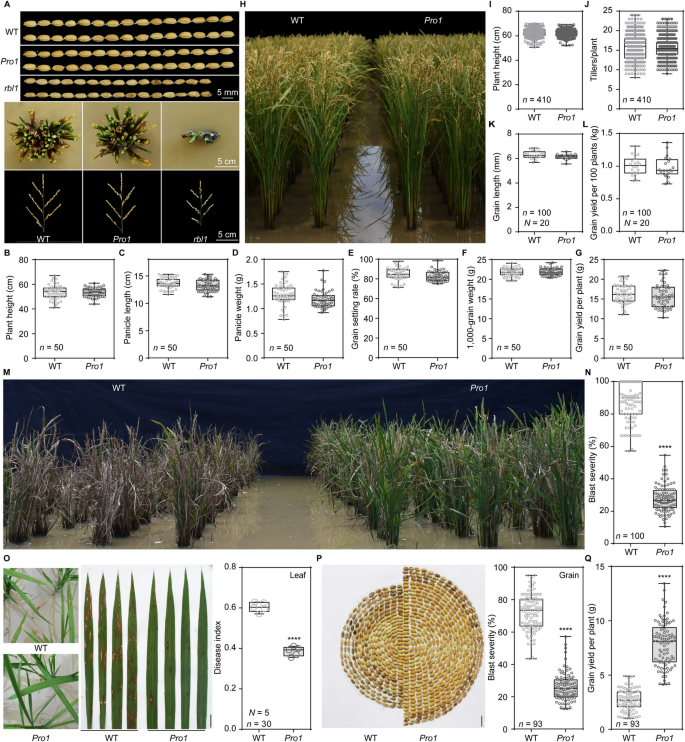
结果解读:共获得5个纯合编辑株系(Pro1-Pro5),其中Pro1株系在峰3和峰4区域存在763bp的缺失,RBL1表达水平较野生型降低71.0%(n=3,P<0.001);Pro2株系存在1051bp的缺失(覆盖峰3和峰4),RBL1表达水平降低61.7%(n=3,P<0.001);Pro3和Pro4株系在峰1区域存在缺失,RBL1表达水平分别升高1.7倍和1.4倍(n=3,P<0.05);Pro5株系因结实率低未进行后续研究。
产品关联:实验所用关键产品:CRISPR载体pRGEB32,qRT-PCR试剂如HiScript II 1st Strand cDNA Synthesis Kit、ChamQ Universal SYBR qPCR Master Mix。
3.4 启动子编辑株系的抗病性与农艺性状鉴定
实验目的:鉴定不同启动子编辑株系的抗病性和农艺性状,筛选出广谱抗病且产量不受影响的最优株系。
方法细节:对6周龄的水稻植株进行病原菌接种:采用打孔接种法接种稻瘟病菌(M. oryzae),剪叶接种法接种白叶枯病菌(Xoo),打孔接种法接种纹枯病菌(R. solani),分别在接种后14天(稻瘟病、白叶枯病)和5天(纹枯病)统计病斑面积或长度;同时测定株高、分蘖数、千粒重、单株产量等农艺性状。
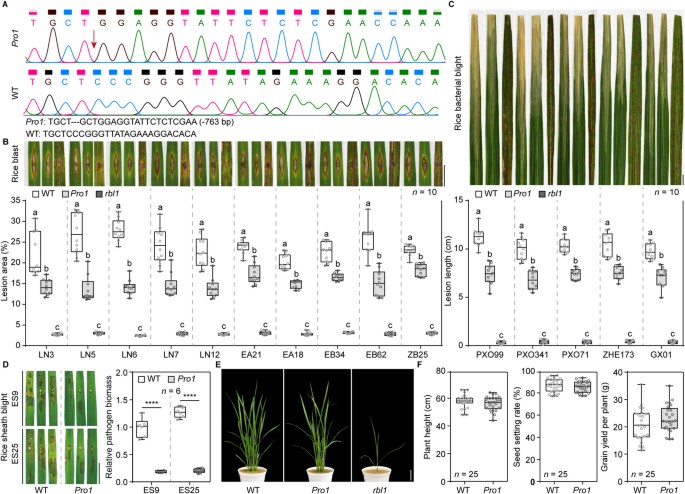
结果解读:Pro1和Pro2株系对稻瘟病的抗性显著增强,病斑面积较野生型分别减少26.7%和13.4%(n=42-58,P<0.001);对白叶枯病的病斑长度分别缩短至7.7cm和9.6cm(野生型为14.9cm,n=14-20,P<0.001);对纹枯病的病斑面积也显著小于野生型(n=6,P<0.0001)。同时,Pro1和Pro2的农艺性状与野生型无显著差异,单株产量分别为23.1g和22.5g(野生型为21.1g,n=10,P>0.05);而Pro3和Pro4株系的抗病性显著降低,农艺性状无显著变化。进一步验证显示,Pro1对10个稻瘟病菌株、5个白叶枯病菌株均具有广谱抗性。
产品关联:文献未提及具体实验产品,领域常规使用病原菌接种的标准方法,农艺性状测定使用常规农学统计方法。
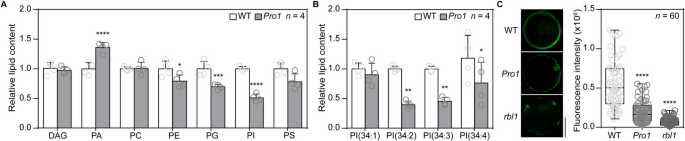
3.5 Pro1株系的抗病机制解析
实验目的:解析Pro1株系广谱抗病的分子机制,阐明启动子编辑调控抗病性的内在逻辑。
方法细节:采用脂质组学分析Pro1与野生型植株的磷脂代谢差异,通过转录组和代谢组分析基因表达和代谢通路的变化,测定活性氧(ROS)、水杨酸(SA)含量和病程相关基因(PR)的表达水平,同时利用PtdIns(4,5)P2生物传感器观察细胞膜上该磷脂的分布变化。
结果解读:脂质组学结果显示,Pro1株系中磷脂酸(PA)含量较野生型升高12.0%(n=4,P<0.05),磷脂酰肌醇(PI)含量降低13.1%(n=4,P<0.05);细胞膜上PtdIns(4,5)P2的荧光信号降低62.7%(n=60,P<0.0001)。生理指标测定显示,Pro1株系在几丁质处理后的ROS水平较野生型升高4.7倍(n=6,P<0.001),SA含量升高1.9倍(n=5,P<0.001),PR基因OsPR1b和OsPR10的表达水平分别升高5.0倍和1.7倍(n=3,P<0.001)。转录组分析显示,13个防御相关通路显著上调,代谢组分析显示苯丙氨酸代谢通路(SA合成的前体通路)显著激活。
产品关联:实验所用关键产品:脂质组学使用HPLC-MS平台,转录组使用BGISEQ-500测序平台,ROS测定使用luminol和辣根过氧化物酶,SA测定使用LC-MS,qRT-PCR试剂如ChamQ Universal SYBR qPCR Master Mix。
3.6 田间试验与品种适用性验证
实验目的:验证Pro1株系在田间自然条件下的抗病性和农艺性状,同时验证该启动子编辑策略在其他水稻品种中的适用性。
方法细节:在湖北、海南的正常田间条件下种植Pro1株系,测定株高、分蘖数、千粒重、单株产量等农艺性状;在湖北恩施的稻瘟病重发区进行田间抗病性鉴定,统计穗瘟、叶瘟的病级和病粒率;同时在水稻品种Nipponbare中编辑相同的RBL1启动子区域,获得nip-8株系并鉴定其抗病性。
结果解读:在正常田间条件下,Pro1的农艺性状与野生型无显著差异;在稻瘟病重发区,Pro1的穗瘟病 severity为28.0%(野生型为75.1%,n=93,P<0.0001),叶瘟病 severity为38.6%(野生型为60.5%,n=150,P<0.0001),病粒率为26.1%(野生型为72.4%,n=93,P<0.0001),单株产量为7.9g(野生型为2.7g,n=93,P<0.0001)。在Nipponbare品种中获得的nip-8株系,RBL1表达水平降低,PR基因表达升高,对稻瘟病的抗性显著增强,证明该策略具有广泛的品种适用性。
产品关联:文献未提及具体实验产品,领域常规使用田间试验的标准设计方法。
4. Biomarker研究及发现成果解析
Biomarker定位与筛选逻辑
本研究涉及两类Biomarker:一是PRO1等位基因(RBL1启动子的763bp缺失区域),作为广谱抗病性状的功能分子标记;二是磷脂代谢物(PA、PI、PtdIns(4,5)P2),作为抗病性的潜在生理标记。PRO1等位基因的筛选逻辑为:生物信息学预测RBL1启动子的核心调控区域→原生质体验证调控功能→CRISPR-Cas9编辑获得株系→表型鉴定与互补实验验证功能;磷脂代谢物的筛选逻辑为:基于RBL1的功能(CDP-DAG合酶,参与磷脂代谢)→脂质组学分析Pro1与野生型的磷脂差异→验证磷脂变化与抗病性的关联。
研究过程详述
PRO1等位基因的来源是CRISPR-Cas9介导的RBL1启动子编辑,验证方法包括Sanger测序、Southern blotting和互补实验(将包含完整启动子的RBL1基因导入Pro1株系,其抗病性恢复为野生型水平),该标记的特异性表现为仅在启动子编辑的抗病株系中存在,与广谱抗病性状完全共分离。磷脂代谢物的来源是水稻叶片组织,验证方法为HPLC-MS脂质组学分析,其中PA的含量在Pro1中升高12.0%(n=4,P<0.05),PI的含量降低13.1%(n=4,P<0.05),细胞膜上PtdIns(4,5)P2的荧光信号降低62.7%(n=60,P<0.0001),这些磷脂的变化与Pro1的抗病性显著相关。
核心成果提炼
PRO1等位基因的核心功能是通过降低RBL1的表达水平,调控磷脂代谢,激活植物基础免疫通路,从而实现广谱抗病且不影响产量,其创新性在于首次通过可预测的启动子编辑获得LMM基因的最优表达水平,打破了免疫-生长权衡的核心瓶颈,该标记可直接用于分子标记辅助育种,快速筛选抗病材料。磷脂代谢物作为潜在的Biomarker,为水稻抗病性的早期鉴定提供了新的指标,其中PA的积累和PI、PtdIns(4,5)P2的降低可作为抗病性的生理标记,用于辅助筛选抗病材料。此外,本研究建立的可预测启动子编辑框架,也为其他LMM基因的精准应用提供了可复制的技术方案。
