1. 领域背景与文献引入
文献英文标题:Transcriptional slippage in bacteria: distribution in sequenced genomes and utilization in IS element gene expression;发表期刊:Genome Biology;影响因子:未公开;研究领域:细菌分子生物学(转录调控与基因表达)
转录滑移是RNA聚合酶在转录均聚核苷酸序列时发生的滑动现象,导致mRNA序列出现非模板编码的核苷酸插入或缺失,进而产生异质性mRNA群体,最终翻译出多种蛋白产物。该现象最早于1962年通过体外实验被发现,后续体内研究证实其在少数细菌基因的表达调控中发挥作用,比如大肠杆菌pyrBI和codBA操纵子的转录起始调控。早期研究多聚焦于单个基因或特定物种,仅揭示了转录滑移的基本机制及有限的功能场景,但随着高通量测序技术的发展,大量细菌基因组序列的公布为全基因组范围分析转录滑移位点提供了可能。当时的研究尚未系统揭示不同细菌中对转录滑移易感序列的选择压力差异,也未明确其在移动遗传元件基因表达中的利用模式,因此本研究通过多基因组比较分析,填补了这一领域空白,为全面理解细菌转录调控的多样性提供了新视角。
2. 文献综述解析
作者对现有研究的分类维度主要包括转录滑移的发生机制、功能应用场景(病毒基因、细菌基因)以及研究技术体系(体外实验、体内验证)。现有研究的关键结论显示,转录滑移可导致mRNA异质性,进而产生功能不同的蛋白产物;在少数细菌基因(如嗜热菌Thermus thermophilus的dnaX基因)和病毒基因(如副黏病毒P基因)中,转录滑移被主动用于产生多种功能蛋白;由于多数情况下转录滑移会产生有害的异常蛋白,其易感序列在编码区通常受到负选择。现有研究的技术方法优势在于,早期体外实验明确了转录滑移的核心机制,体内实验验证了其在特定基因中的调控功能,但局限性也较为明显:研究多集中于单个基因或少数物种,缺乏全基因组范围的系统分析;对不同细菌中选择压力的差异及转录滑移在移动遗传元件中的利用情况研究不足,也未关注到其对基因组注释的影响。
本研究的创新价值在于,首次对108个细菌基因组进行全基因组范围的转录滑移易感序列(9个连续A/T,即9A/9T)分布分析,揭示了不同细菌中负选择作用的序列长度阈值差异;通过比较基因组学分析,明确了转录滑移在插入序列(IS元件)基因表达中的广泛利用,证实其可作为核糖体移码的替代机制产生转座酶融合蛋白;此外还发现了“假假基因”现象,即转录滑移可恢复移码突变的开放阅读框(ORF),纠正了传统基因组注释中将这类基因错误归类为假基因的偏差,为基因组注释提供了新的参考依据。
3. 研究思路总结与详细解析
本研究的整体框架为:以明确细菌基因组中长A/T均聚序列的分布规律及转录滑移的功能利用为核心目标,围绕“不同细菌中对转录滑移易感序列的选择压力差异”“转录滑移在基因表达中的功能场景”两大科学问题,采用“全基因组序列统计→随机基因组对照验证→功能基因筛选→比较基因组学分析→机制拓展”的闭环技术路线,系统揭示了转录滑移的分布特征与功能价值。
3.1 细菌基因组中A/T均聚序列的分布分析
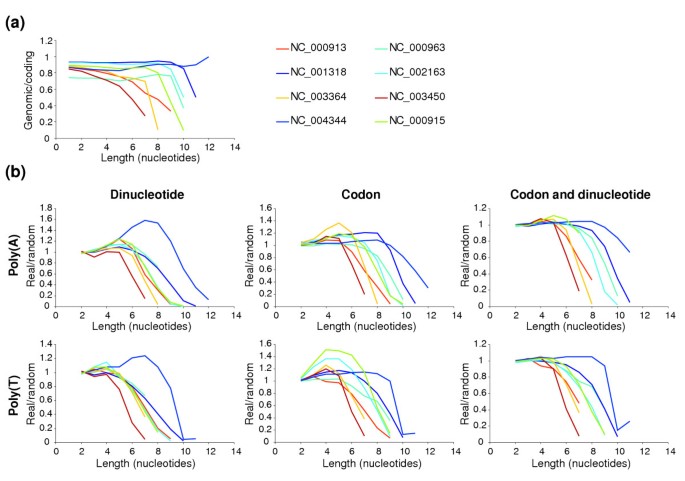
实验目的:明确不同细菌基因组中编码区与全基因组中不同长度A/T均聚序列的分布差异,确定负选择作用的序列长度阈值。
方法细节:下载108个已测序细菌的全基因组序列及编码区序列,统计不同长度的A/T均聚序列在编码区(Pc)与全基因组(Pg)中的数量,计算Pc/Pg比值;为排除密码子使用、氨基酸组成等因素的干扰,为每个代表性基因组生成1000个随机基因组(保留原蛋白序列,随机化密码子并考虑密码子使用偏好),比较真实基因组与随机基因组中A/T均聚序列的分布差异。
结果解读:在AT富集的细菌基因组中,当A/T均聚序列长度达到9个核苷酸时,Pc/Pg比值显著下降,表明该长度的序列在编码区受到明显的负选择;而在GC富集的基因组中,长A/T序列本身出现概率极低,未观察到显著的Pc/Pg比值变化;部分AT富集基因组(如伯氏疏螺旋体Borrelia burgdorferi)中各长度A/T序列的Pc/Pg比值无显著变化,推测其可能存在抑制转录滑移的特殊机制。
产品关联:文献未提及具体实验产品,领域常规使用基因组序列分析脚本、CodonDiShuffle等工具进行序列统计与随机化分析。
3.2 含9A/9T序列的功能基因筛选与比较分析
实验目的:筛选细菌基因组中含9A/9T序列的编码基因,通过比较基因组学分析其转录滑移的功能利用情况。
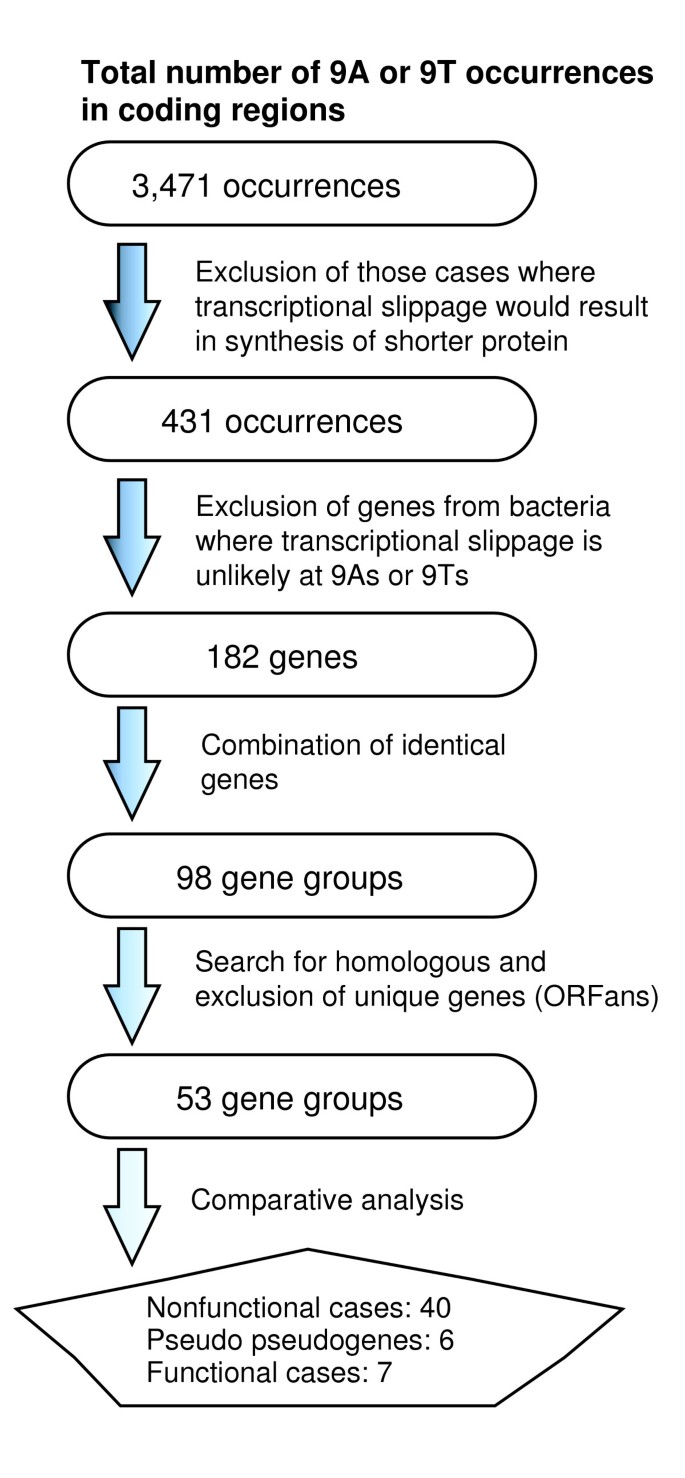
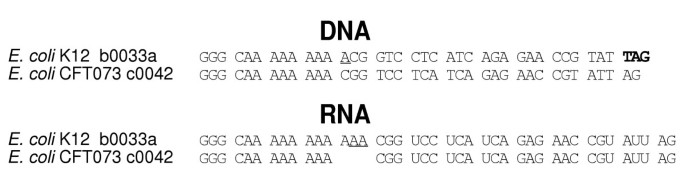
方法细节:在108个细菌基因组的编码区中搜索9A/9T序列,筛选出转录滑移可产生更长ORF的基因(即滑移后ORF长度超过原序列);排除那些基因组中9A/9T序列数量超过20且无负选择证据的细菌(推测其转录滑移不活跃);对筛选出的基因进行去冗余处理,合并同源基因;通过tBLASTn比对寻找同源基因,排除ORFan基因(无同源序列,不适合比较分析);对剩余基因进行比较基因组学分析,若至少两个非同源但序列相似的基因中,转录滑移融合的ORF结构保守,则认为该转录滑移具有功能。
结果解读:共筛选出98个基因群,其中53个找到同源基因;40个基因的转录滑移未发现明确功能,推测其转录滑移无显著有害或有益作用;6个基因的转录滑移可恢复移码突变的ORF,产生与其他物种中完整ORF同源的蛋白,这类基因被称为“假假基因”;7个基因(其中6个来自IS元件)的9A/9T序列及ORF融合结构在同源基因中保守,表明转录滑移被功能利用,尤其是在IS元件中,转录滑移替代核糖体移码产生转座酶融合蛋白(ORFA-ORFB),为IS元件的转座提供必要功能。

产品关联:文献未提及具体实验产品,领域常规使用BLAST序列比对工具、Clustal多序列比对软件进行同源分析与序列比对。
3.3 转录滑移功能机制的验证与拓展分析
实验目的:验证IS元件中转录滑移的功能,并探讨转录滑移的潜在机制与其他可能的应用场景。
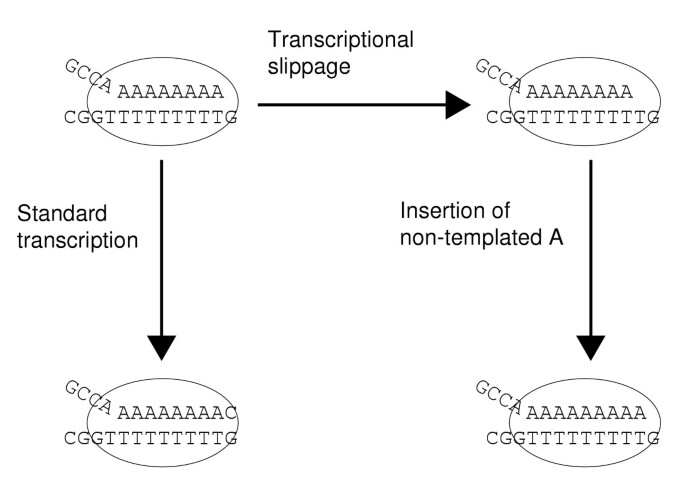
方法细节:通过质谱分析耐辐射球菌Deinococcus radiodurans的蛋白组,寻找IS元件基因通过转录滑移产生的融合蛋白;结合现有研究成果,分析转录滑移的分子机制,并推测其他低复杂度重复序列引发转录滑移的可能性。
结果解读:质谱分析初步发现了IS元件基因通过转录滑移产生的融合蛋白产物(未发表数据);提出转录滑移的分子模型:RNA聚合酶在均聚序列处,RNA链与模板解离,重新结合时发生滑动,导致核苷酸插入或缺失;推测(AT)n等重复序列也可能引发转录滑移,且在病原菌毒力基因中,简单序列重复可能通过转录滑移产生多种蛋白产物,帮助病原菌逃避宿主免疫应答。
产品关联:文献未提及具体实验产品,领域常规使用质谱分析系统(如FITC质谱)进行蛋白组分析。
4. Biomarker研究及发现成果解析
本研究中涉及的Biomarker为转录滑移易感序列(9A/9T均聚序列),属于转录调控类功能元件。其筛选与验证逻辑为:首先通过全基因组序列统计筛选出编码区中的9A/9T序列,结合随机基因组对照验证其在AT富集细菌中受到负选择;再通过比较基因组学分析,验证其在同源基因中的保守性及功能利用情况,形成“全基因组筛选→选择压力验证→功能保守性验证”的完整逻辑链条。
研究过程详述:该Biomarker来源于细菌基因组编码区序列,验证方法包括全基因组序列统计分析、随机基因组对照分析、比较基因组学比对及蛋白组质谱分析。在AT富集细菌中,9A/9T序列在编码区的丰度显著低于全基因组(Pc/Pg比值显著降低),但文献未明确提供其特异性与敏感性的量化数据(文献未明确提供该数据,基于图表趋势推测)。在IS元件中,9A/9T序列保守存在,转录滑移可将两个重叠的ORF融合为一个完整的ORF,产生转座酶融合蛋白;在“假假基因”中,9A/9T序列由移码突变产生,转录滑移可恢复原ORF,产生与其他物种中完整ORF同源的功能蛋白。
核心成果提炼:该Biomarker在IS元件基因表达中作为替代核糖体移码的机制,产生转座酶融合蛋白,是IS元件转座所必需的功能元件,创新性在于首次系统揭示了转录滑移在细菌移动遗传元件中的广泛利用;在“假假基因”中,该Biomarker可恢复移码突变的ORF,纠正了传统基因组注释中对这类基因的错误归类,为基因组注释提供了新的参考。统计学结果显示,在108个细菌基因组中,共发现27个含9A/9T序列的IS元件(n=27,未提及P值),6个“假假基因”通过转录滑移恢复ORF(n=6,未提及P值)。
