1. 领域背景与文献引入
文献英文标题:BART-Seq: cost-effective massively parallelized targeted sequencing for genomics, transcriptomics, and single-cell analysis;发表期刊:Genome Biology;影响因子:约14.03(2019年);研究领域:靶向测序技术、单细胞转录组学、基因组学。
下一代测序(NGS)技术的发展推动了生命科学研究的规模化,其中通过DNA条形码对NGS文库进行索引(即多路复用)是降低成本、提高样本通量的关键。在单细胞转录组学领域,现有技术如Drop-Seq、10× Genomics可实现数千个单细胞的同时分析,但基因覆盖度较低,仅适用于细胞类型的分类;SMART-Seq2技术可实现高覆盖度的单细胞转录组分析,但成本高昂,样本通量有限。在靶向测序领域,基于捕获的方法(如Illumina Targeted RNA Expression)需要大量起始材料,且碎片化、杂交等中间步骤导致定量动态范围较差;基于聚合酶链式反应(PCR)的方法操作相对简单,但引物兼容性差,易出现非特异性扩增;微流控设备(如Fluidigm Biomark)和探针杂交技术(如Nanostring nCounter)定量准确性高,但设备昂贵、操作复杂,难以实现大规模样本的分析。领域共识:目前缺乏一种兼具低成本、高灵敏度、宽动态范围且可同时适用于基因组和转录组分析的靶向测序技术,尤其是在大规模单细胞研究中的应用存在空白。
针对上述领域痛点,本研究开发了一种名为条形码组装靶向测序(Barcode Assembly foR Targeted Sequencing, BART-Seq)的新技术,通过先索引后扩增的创新策略,无需中间纯化步骤,实现了低成本、高灵敏度的大规模样本靶向测序,同时适用于基因组DNA(gDNA)和RNA的分析,为生命科学研究提供了一种高效的技术工具。
2. 文献综述解析
作者按技术原理将现有靶向测序和单细胞转录组技术分为四类:基于捕获的靶向测序技术、基于PCR/连接的靶向测序技术、微流控/探针杂交技术、单细胞全局转录组技术,系统评述了各类技术的优势与局限性,明确了当前领域的核心需求。
基于捕获的靶向测序技术可特异性靶向基因组或转录组的特定区域,适用于大样本量的批量分析,但需要微克级的起始材料,碎片化、磁珠捕获、杂交等中间步骤会导致样本损失和定量偏差,动态范围较窄;基于PCR/连接的靶向测序技术操作相对简便,起始材料需求较低,但引物设计难度大,多重PCR中引物兼容性差,易形成引物二聚体和非特异性扩增,定量准确性不足;微流控设备和探针杂交技术可实现高精度的定量分析,部分技术可实现空间转录组分析,但设备和试剂成本高昂,操作流程复杂,样本通量有限,难以满足数千甚至数万个样本的大规模分析需求;单细胞全局转录组技术可实现大规模单细胞的转录组分析,推动了细胞异质性的研究,但基因覆盖度低,仅能检测高表达基因,适用于细胞分型,无法深入解析基因表达的细微变化和分子机制。
与现有技术相比,BART-Seq技术具有三大核心创新点:一是采用先索引后扩增的策略,不同于现有先扩增后索引的靶向测序方法,避免了中间纯化步骤导致的样本损失,提高了灵敏度和定量准确性;二是通过生物信息学优化设计条形码和引物,确保了多重PCR的特异性和引物兼容性,可同时靶向数十个基因区域;三是成本极低,单个样本的分析成本约为1美元,远低于现有靶向测序和单细胞转录组技术,且可同时适用于基因组和转录组分析,是首个满足低成本、高灵敏度、规模化需求的靶向测序技术,填补了领域空白。
3. 研究思路总结与详细解析
本研究的核心目标是开发并验证BART-Seq技术,实现低成本、高灵敏度、可规模化的靶向测序,同时适用于基因组和转录组的大规模样本(包括单细胞)分析;核心科学问题是解决先索引后扩增策略中的引物兼容性、样本识别准确性、定量准确性等关键技术难题;技术路线为:生物信息学优化设计条形码和引物→构建条形码-引物复合物→PCR靶向扩增→NGS测序→数据处理与分析,分别在基因组BRCA突变检测和转录组干细胞分化机制研究中验证技术性能。
3.1 条形码与引物的生物信息学设计及优化
实验目的:设计具有高兼容性、低非特异性的条形码和引物,确保多重PCR的特异性和效率,减少样本识别过程中的错误。
方法细节:首先筛选GC含量为50%-60%的8-mer条形码和10-mer接头序列,去除含连续重复碱基的序列;通过模拟退火算法进行全局优化,选择两两比对得分最低的序列,降低序列相似性和二级结构形成的概率;针对每个靶标区域,使用Primer3软件设计最多5对引物,确保引物3"端为胸腺嘧啶(T),以适配Klenow片段的模板无关A尾添加特性;通过模拟退火算法优化引物组合,计算所有引物对的最小自由能(mfe),选择mfe最高的组合以减少引物二聚体的形成;最后通过BLAST比对排除与人类基因组或转录组同源的序列,开发在线工具(http://icb-bar.helmholtz-muenchen.de)供科研人员设计引物和条形码。
结果解读:凝胶电泳结果显示,条形码-引物复合物的构建过程稳定,中间产物清晰可见(图1c);当多重PCR的引物数量从1增加到10时,扩增效率未受到显著影响(图1e),说明设计的引物具有良好的兼容性,可用于多重靶向扩增实验。
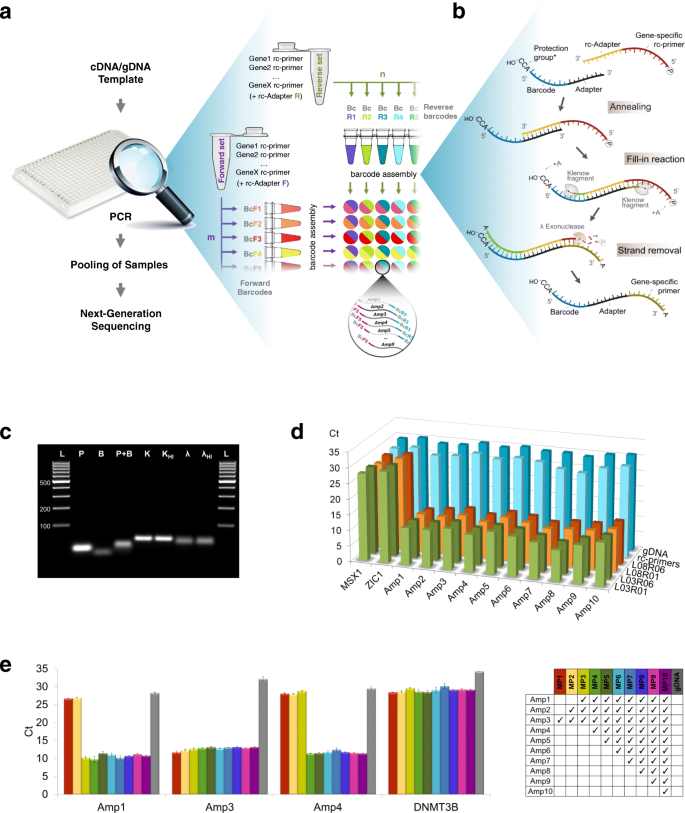
产品关联:文献未提及具体实验产品,领域常规使用引物设计软件(如Primer3)、DNA序列分析工具(如Biopython、ViennaRNA)等。
3.2 gBART-Seq技术体系构建与BRCA突变检测验证
实验目的:构建适用于基因组分析的BART-Seq技术(gBART-Seq),验证其在临床样本中检测BRCA突变的准确性和特异性。
方法细节:通过Klenow片段的双向填充反应将条形码与接头-引物复合物连接,再通过λ核酸外切酶(λ-exo)去除反向互补链,生成单链条形码-引物复合物;以人类BRCA1和BRCA2基因的10个区域为靶标,使用4种条形码组合进行多重PCR扩增,通过嵌套qPCR验证扩增特异性;收集96名德系犹太裔乳腺癌患者的gDNA样本,使用12个正向和8个反向条形码进行靶向扩增,构建NGS文库后用Illumina MiSeq测序,分析BRCA founder突变的等位基因频率,对比临床检测结果;为减少λ-exo对条形码5"端的修剪,在条形码5"端添加三核苷酸保护基(5"CCA),再次进行突变检测验证。
结果解读:嵌套qPCR结果显示,所有10个靶标区域均被特异性富集,循环阈值(Ct)显著低于非扩增样本、非靶标区域和非条形码引物对照组(图1d);初始实验中,92/96(n=96,约96%)的样本检测结果与临床一致(图2c);添加5"CCA保护基后,95/96(n=96,99%)的样本检测结果与临床一致,仅1例样本可能因交叉污染出现误判(图2d),证明gBART-Seq具有极高的准确性和特异性,可用于临床样本的基因突变检测。
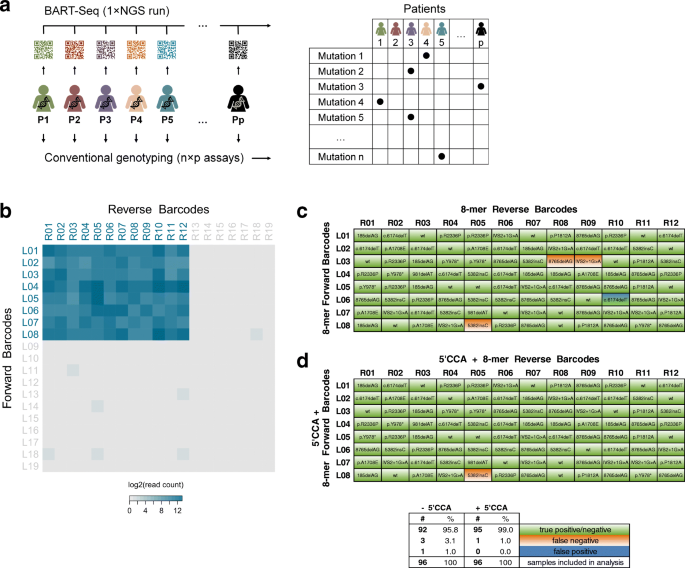
产品关联:实验所用关键产品:GeneRuler™ 100 bp DNA Ladder(Thermo Fisher Scientific)、Illumina MiSeq测序平台、λ核酸外切酶(New England Biolabs)等。
3.3 rBART-Seq技术体系构建与转录组定量准确性验证
实验目的:构建适用于RNA分析的BART-Seq技术(rBART-Seq),验证其在转录组定量分析中的准确性和动态范围。
方法细节:设计靶向11个人类多能性和管家基因转录本(其中5个为跨外显子引物)及4个外源RNA spike-in的引物,通过嵌套qPCR验证引物特异性;制备人类多能干细胞(hPSCs)的总RNA稀释系列(4-256 pg),添加固定浓度的RNA spike-in,反转录为互补DNA(cDNA)后,使用不同条形码组合进行PCR扩增,构建NGS文库后用Illumina NextSeq测序;分析基因读数与RNA模板浓度的相关性,对比rBART-Seq与现有全局转录组技术的定量准确性。
结果解读:嵌套qPCR验证显示引物特异性良好;RNA稀释系列实验中,不同条形码标记的等摩尔样本的变异系数低于25%,基因读数与模板RNA浓度的线性回归决定系数(R²)平均高于0.96,斜率接近1(图3c);与现有全局转录组技术相比,rBART-Seq的线性回归R²中位数为0.98,定量准确性更优(图3d),证明rBART-Seq具有良好的定量准确性和宽动态范围,可用于低起始量RNA样本的定量分析。
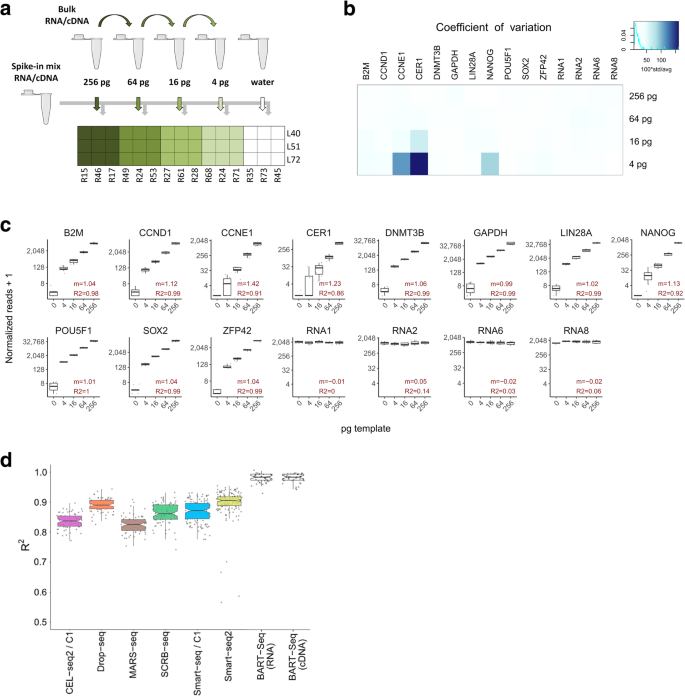
产品关联:实验所用关键产品:SuperScript™ III First-Strand Synthesis System(Invitrogen)、RNeasy Mini Kit(QIAGEN)、Illumina NextSeq测序平台等。
3.4 rBART-Seq在单细胞转录组分析中的应用:不同培养基对hPSCs多能性的影响
实验目的:验证rBART-Seq在单细胞转录组分析中的可行性,探究不同培养基对人类胚胎干细胞(hESCs)多能性状态的调节作用。
方法细节:将培养在mTeSR™1、KSR-bFGF、E8三种培养基中的hESCs及BJ成纤维细胞通过荧光激活细胞分选(FACS)技术分选到含RNA spike-in的反转录反应体系中,共分选超过4500个孔;通过rBART-Seq技术进行单细胞转录组测序,使用RNA spike-in进行数据归一化,去除低信号的空孔样本;通过均匀流形近似和投影(UMAP)分析单细胞转录组数据,比较不同培养基中hESCs的基因表达差异。
结果解读:单细胞数据显示,每个孔中的细胞数量与基因读数数量高度相关(图4b),hESCs与成纤维细胞的基因表达谱存在显著差异(图4c);UMAP分析显示,mTeSR™1培养基培养的hESCs主要处于始发态(primed-like),表现为LIN28A、DNMT3B高表达,而E8培养基培养的hESCs主要处于基态(ground state-like),表现为NANOG、ZFP42高表达(图4d),说明不同培养基可调节hESCs的多能性状态,为干细胞培养和分化研究提供了参考。
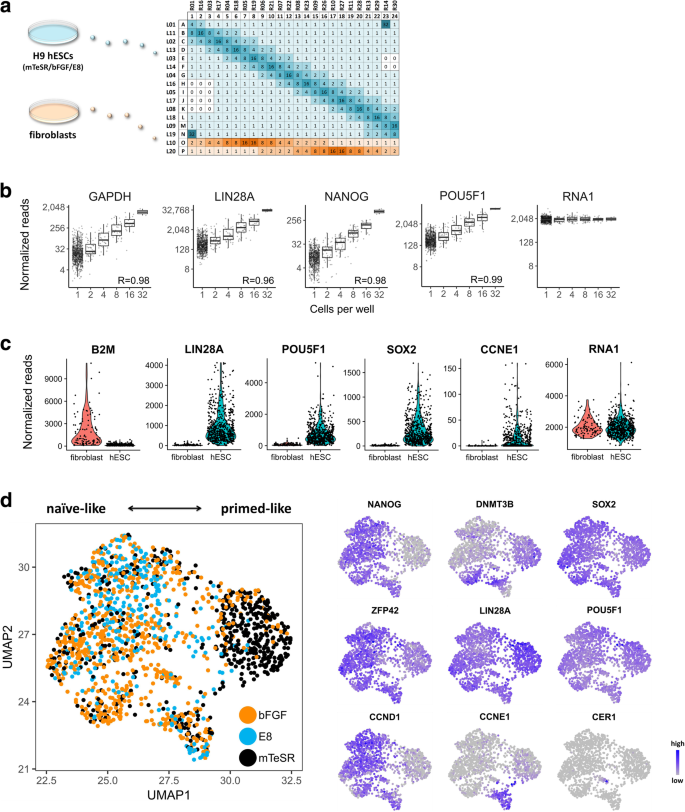
产品关联:实验所用关键产品:mTeSR™1培养基(Stem Cell Technologies)、Accutase(Sigma)、BD Aria III分选仪等。
3.5 rBART-Seq在Wnt/β-catenin通路激活机制研究中的应用
实验目的:利用rBART-Seq技术探究Wnt/β-catenin通路不同激活方式对hESCs分化的影响,解析通路激活的分子机制。
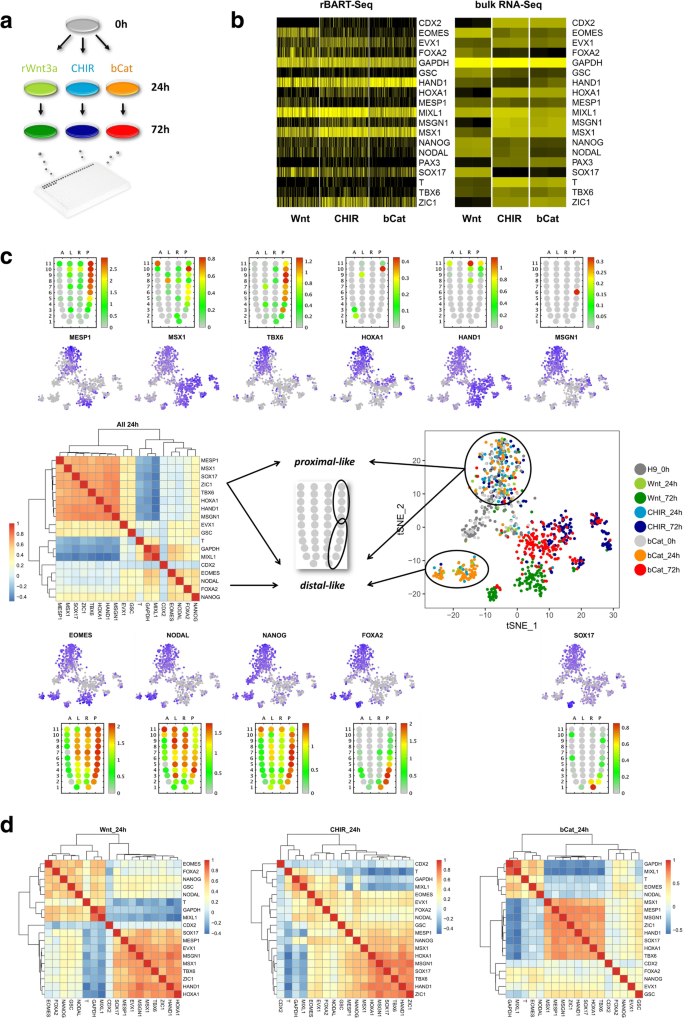
方法细节:用重组Wnt3a(rWnt3a)、GSK3抑制剂CHIR99021处理hESCs,同时用强力霉素(Dox)诱导表达组成型激活的β-catenin(ΔN90);在处理0、24、72小时后,通过FACS分选单细胞,使用rBART-Seq技术分析22个早期原肠胚标记物、管家基因及3个RNA spike-in的表达;对比rBART-Seq与全局RNA-Seq的结果,通过t分布邻域嵌入(tSNE)和基因相关性分析解析细胞亚群的特征。
结果解读:rBART-Seq的单细胞转录组结果与全局RNA-Seq结果高度相似,证明rBART-Seq可准确反映基因表达的变化(图5b);处理24小时后,基因相关性分析显示两个细胞亚群,分别对应胚胎发育中的近端和远端区域(图5c);处理72小时后,rWnt3a处理组产生 definitive endoderm-like(FOXA2、SOX17高表达)和lateral plate mesoderm-like(HAND1、MESP1高表达)两种细胞亚群,而CHIR99021处理组和ΔN90诱导组的细胞亚群特征相似,与rWnt3a处理组存在显著差异(图5d),说明CHIR99021主要通过稳定β-catenin激活通路,但与rWnt3a的激活效果存在差异,为Wnt通路的研究提供了新的见解。
产品关联:实验所用关键产品:CHIR99021(Tocris)、Y-27632(R&D Systems)、强力霉素(Clontech)等。
4. Biomarker研究及发现成果解析
本研究涉及两类生物标志物(Biomarker):一类是BRCA1/2的遗传突变标志物,用于乳腺癌的遗传易感筛查;另一类是干细胞多能性和分化的基因表达标志物,用于解析干细胞的状态和分化机制。
Biomarker定位:BRCA突变标志物为德系犹太裔人群中BRCA1/2的10个founder突变,筛选逻辑基于已报道的该人群遗传易感突变,验证逻辑为:gBART-Seq靶向扩增突变区域→NGS测序分析等位基因频率→与临床检测结果对比;干细胞标志物包括多能性标志物(NANOG、ZFP42、LIN28A、DNMT3B)和早期原肠胚分化标志物(FOXA2、SOX17、HAND1、MESP1),筛选逻辑基于已报道的干细胞状态和胚胎发育标记物,验证逻辑为:rBART-Seq单细胞转录组分析→基因表达与细胞状态的关联分析。
研究过程详述:BRCA突变标志物的来源为96名德系犹太裔乳腺癌患者的gDNA样本,验证方法为gBART-Seq靶向扩增突变区域,NGS测序后计算突变等位基因与野生型等位基因的比值,当比值>0.20时判定为突变;特异性和敏感性:添加5"CCA保护基后,99%的样本与临床检测结果一致,文献未明确提供ROC曲线数据,但高一致性证明其特异性和敏感性优异;干细胞标志物的来源为hPSCs的单细胞转录组样本,验证方法为rBART-Seq定量基因表达,通过UMAP和tSNE分析细胞亚群的基因表达特征;多能性标志物的特异性表现为NANOG、ZFP42在基态hPSCs中高表达,LIN28A、DNMT3B在始发态hPSCs中高表达,与已知细胞状态的标记物表达模式完全一致;分化标志物的特异性表现为FOXA2、SOX17在 definitive endoderm-like细胞中高表达,HAND1、MESP1在 lateral plate mesoderm-like细胞中高表达,与胚胎发育的分子特征一致。
核心成果提炼:BRCA突变标志物的功能关联为乳腺癌的遗传易感标志物,本研究证明gBART-Seq可高效、准确地检测这些突变,可用于大规模人群的遗传筛查;多能性标志物的创新性在于首次通过大规模单细胞分析明确了不同培养基对hPSCs多能性状态的调节,NANOG、ZFP42可作为基态hPSCs的特异性标志物,LIN28A、DNMT3B可作为始发态hPSCs的特异性标志物;早期原肠胚分化标志物的创新性在于揭示了CHIR99021与rWnt3a激活Wnt通路的差异,FOXA2、SOX17和HAND1、MESP1可分别作为 definitive endoderm和lateral plate mesoderm的特异性标志物,为干细胞分化机制的研究提供了关键分子标记。文献未提供上述标志物的风险比(HR)等统计学数据,但高一致性的实验结果证明了其可靠性。
