1. 领域背景与文献引入
文献英文标题:An integrative pan-cancer-wide analysis of epigenetic enzymes reveals universal patterns of epigenomic deregulation in cancer;发表期刊:Genome Biology;影响因子:未明确提供;研究领域:肿瘤表观遗传学。
领域共识:2011年Hanahan等提出表观遗传失调是癌症的核心标志之一,后续研究逐步揭示表观遗传酶(epigenetic enzymes, EEs)的遗传突变可导致全基因组DNA甲基化(DNA methylation, DNAm)模式异常,如DNMT3A突变在急性单核细胞白血病中引发甲基化紊乱,IDH1突变在胶质瘤中建立高甲基化表型。当前肿瘤表观遗传学的研究热点集中在表观遗传酶作为癌症驱动因子和潜在治疗靶点的挖掘,已发现EZH2、UHRF1等多个表观遗传酶在特定癌种中存在表达失调或功能异常。但现有研究多聚焦单一癌种的局部调控机制,缺乏泛癌层面的系统整合分析,尚未明确是否存在跨癌种的普遍表观基因组失调模式,也未系统解析表观遗传酶对全基因组DNA甲基化的调控网络,这一空白限制了对癌症表观调控共性机制的理解。
本研究针对上述核心问题,通过整合TCGA数据库10种癌种的RNA-seq和DNA甲基化数据,开展泛癌层面的表观遗传酶表达失调分析及甲基化调控关联研究,旨在识别跨癌种普遍失调的表观遗传酶及其对全基因组DNA甲基化的调控作用,为癌症表观遗传治疗提供泛癌通用的靶点和理论依据。
2. 文献综述解析
作者以表观遗传酶的功能分类(writers、readers、editors、erasers)为维度,系统梳理了现有研究中表观遗传酶在癌症中的突变、表达失调及调控作用,明确了单一癌种研究的局限性,进而提出泛癌整合分析的必要性。
现有研究已证实部分表观遗传酶在特定癌种中具有驱动作用,如EZH2作为组蛋白甲基转移酶,在多种癌症中过表达并促进肿瘤进展;ZBTB4作为DNA甲基化阅读器,其表达水平与乳腺癌无复发生存显著相关。技术方法上,现有研究多采用单一癌种的组学数据进行分析,能精准揭示癌种特异性的表观调控机制,但缺乏跨癌种的整合对比,无法识别表观遗传失调的普遍规律。局限性方面,多数研究聚焦表观遗传酶的突变分析,而表观遗传酶的功能失调还可由基因扩增、缺失或自身DNA甲基化修饰导致,且单一癌种中表观遗传酶的突变频率通常较低,难以反映其在癌症发生中的整体调控作用。
通过对比现有研究的未解决问题,本研究的创新价值凸显:首次通过泛癌整合分析,系统识别出18个跨癌种一致失调的表观遗传酶,其中11个为潜在癌基因、7个为潜在抑癌基因;明确了全基因组DNA高甲基化和低甲基化是两个独立的调控过程,由不同的表观遗传酶网络调控;填补了泛癌层面表观遗传酶调控全基因组甲基化模式的研究空白,为理解癌症表观调控的共性机制提供了新视角。
3. 研究思路总结与详细解析
本研究的整体框架为“表观遗传酶基因集构建→泛癌差异表达筛选→DNA甲基化不稳定指数量化→表达与甲基化关联分析→因果网络建模识别关键调控因子→跨癌种调控位点验证”的闭环逻辑;研究目标是识别跨癌种普遍失调的表观遗传酶及其对全基因组DNA甲基化的调控作用;核心科学问题是癌症中是否存在跨癌种的表观基因组失调普遍模式,以及哪些表观遗传酶是全基因组DNA甲基化的核心调控因子。
3.1 表观遗传酶基因集构建与泛癌差异表达分析
实验目的:构建全面覆盖表观遗传调控功能的基因集,筛选在跨癌种中一致失调的表观遗传酶。
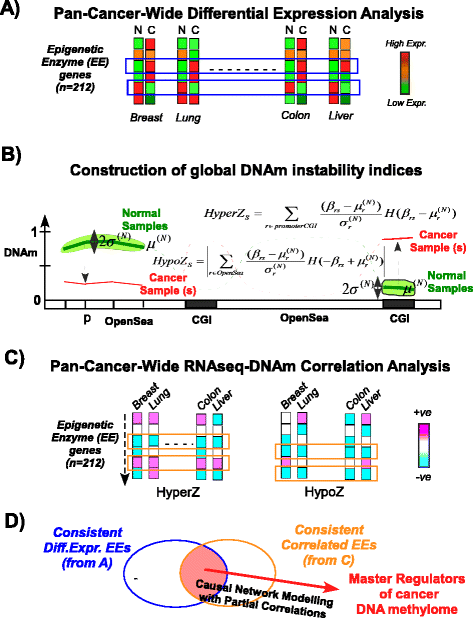
方法细节:从近期权威综述和文献检索中收集212个表观遗传酶基因,涵盖DNA甲基转移酶、组蛋白修饰酶、染色质重塑酶等所有核心表观调控功能类别;下载TCGA数据库中10种癌种(乳腺癌、膀胱癌等)的RNA-seq V2水平3数据,经过零值替换、对数转换、批次效应校正等质量控制步骤后,采用校正t检验分析正常与癌组织的基因表达差异;筛选在至少8种癌种中呈现一致上调或下调的基因,并通过二项式模型分析结果的统计学显著性。
结果解读:热图显示62个表观遗传酶在至少8种癌种中一致失调,其中35个基因在癌组织中显著上调(潜在癌基因),27个基因显著下调(潜在抑癌基因);统计学模拟显示,随机选取212个基因时,仅预期0.54个基因上调、0.89个基因下调,本研究结果的P值分别小于10^-50和10^-30,具有极高的统计学显著性;上调基因包括EZH2、DNMT1/3A/3B、UHRF1等已知癌基因,下调基因包括ZBTB4、CBX7等已报道的抑癌基因。
产品关联:文献未提及具体实验产品,领域常规使用R语言的limma包进行差异表达分析,sva包进行批次效应校正,TCGA数据库提供标准化组学数据。
3.2 DNA甲基化不稳定指数(HyperZ/HypoZ)构建
实验目的:量化肿瘤样本的全基因组DNA高甲基化和低甲基化水平,明确二者是否为独立的调控过程。
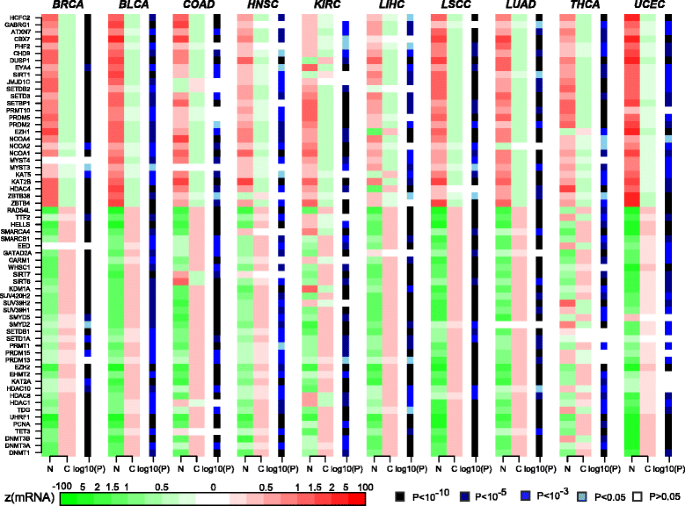
方法细节:将Illumina 450K芯片的CpG探针分为启动子CpG岛(CGI)和开放海区域,采用bumphunter工具将相邻CpG探针聚类为区域;计算每个肿瘤样本中各区域相对于正常样本的Z评分,构建HyperZ指数(反映启动子CGI的异常高甲基化水平)和HypoZ指数(反映开放海区域的异常低甲基化水平);通过二维密度图和Spearman相关分析,评估10种癌种中两个指数的相关性。
结果解读:二维密度图显示,在所有10种癌种中,HyperZ与HypoZ的相关性极弱(R²值约0.1或更低),仅因样本量较大具有统计学显著性,表明全基因组DNA高甲基化和低甲基化是两个独立的调控过程;乳腺癌亚型分析显示,luminal B亚型的HyperZ和HypoZ指数均最高,HER2+亚型的HyperZ指数与luminal B相当,但HypoZ指数显著更低,进一步验证了二者调控机制的独立性。
产品关联:文献未提及具体实验产品,领域常规使用Illumina Infinium HumanMethylation450 BeadChip进行DNA甲基化检测,R语言的minfi包进行数据预处理,bumphunter包进行区域聚类分析。
3.3 表观遗传酶表达与DNA甲基化指数的关联分析
实验目的:识别与全基因组DNA甲基化失调直接相关的表观遗传酶,明确跨癌种的普遍关联模式。
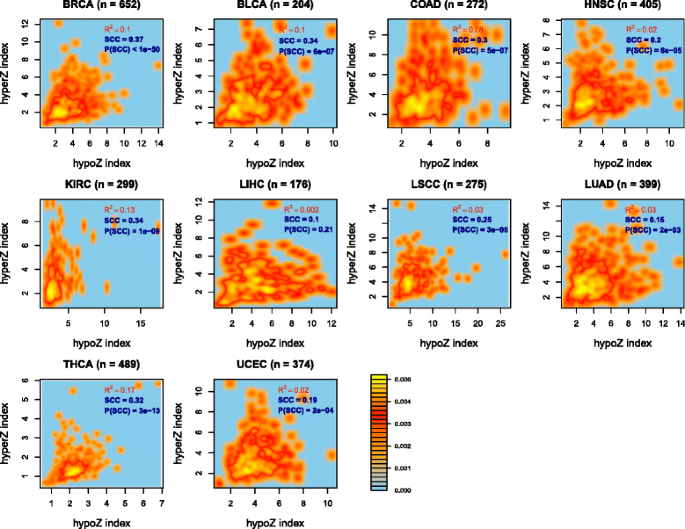
方法细节:利用匹配的肿瘤样本RNA-seq和DNA甲基化数据,计算每个癌种中212个表观遗传酶表达水平与HyperZ、HypoZ指数的Pearson相关系数;将相关系数转换为Fisher Z统计量并计算P值,筛选在至少6种癌种中呈现一致方向(正相关或负相关)且P<0.05的关联;通过二项式模型验证结果的统计学显著性。
结果解读:热图显示,16个表观遗传酶与HyperZ指数存在跨癌种一致关联(5个正相关、11个负相关),33个表观遗传酶与HypoZ指数存在跨癌种一致关联(18个正相关、15个负相关);模拟分析显示,随机基因中仅预期0.54个与HyperZ指数存在跨癌种关联,本研究结果的P值范围为0.001至10^-24,具有显著统计学意义;多数与HyperZ指数关联的基因不与HypoZ指数关联,进一步支持了全基因组高甲基化和低甲基化的独立调控机制。
产品关联:文献未提及具体实验产品,领域常规使用R语言的corrplot包进行相关性可视化,fisherz函数进行统计转换。
3.4 因果网络建模与关键调控因子识别
实验目的:排除间接关联干扰,精准识别全基因组DNA甲基化的核心调控因子。
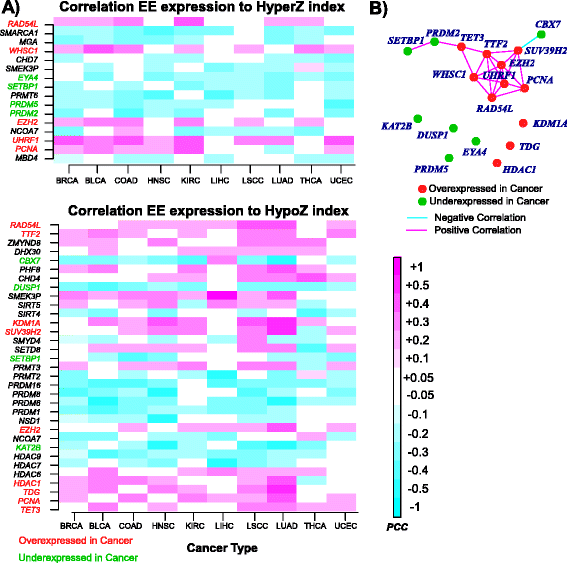
方法细节:筛选同时满足“跨癌种一致失调”和“与DNA甲基化指数一致关联”的18个表观遗传酶,采用偏相关分析构建因果调控网络,控制其他17个表观遗传酶的表达水平及所有18个基因的启动子DNA甲基化水平,评估每个表观遗传酶与HyperZ、HypoZ指数的直接关联;构建10种癌种的共识调控网络,筛选在至少6种癌种中存在直接关联的核心调控因子。
结果解读:因果网络分析显示,UHRF1和WHSC1的过表达与全基因组启动子高甲基化直接相关,CBX7的低表达与开放海区域低甲基化直接相关,三者为泛癌层面全基因组DNA甲基化的核心调控因子;表达相关性分析显示,UHRF1、EZH2、WHSC1等癌基因形成正相关核心簇,而KDM1A、HDAC1等表观遗传酶的调控作用相对独立,不与核心簇基因显著相关。
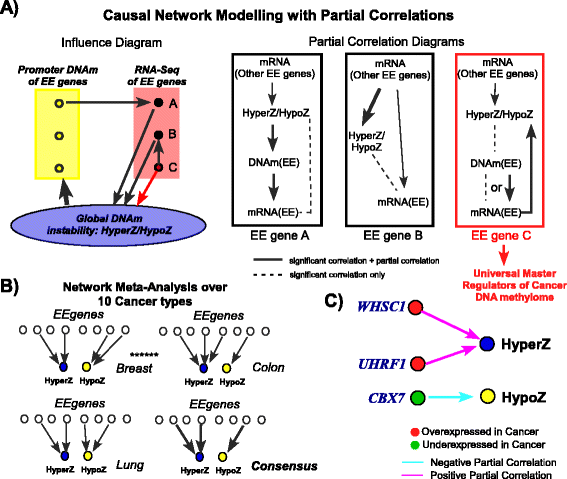
产品关联:文献未提及具体实验产品,领域常规使用R语言的ppcor包进行偏相关分析,igraph包进行网络可视化。
3.5 跨癌种调控位点一致性验证
实验目的:验证核心调控因子对DNA甲基化的调控位点是否具有跨癌种普遍性。
方法细节:计算核心调控因子(UHRF1、WHSC1、CBX7)表达水平与每个基因组区域DNA甲基化水平的Pearson相关系数,在乳腺癌中按相关性强度对基因组区域排序;将该排序与其他9种癌种的排序进行Spearman秩相关分析,评估调控位点的跨癌种一致性。
结果解读:热图显示,与UHRF1、WHSC1、CBX7表达相关性最高的基因组区域,在10种癌种中的排序高度一致,Spearman秩相关的P值均小于0.001,表明这些核心调控因子的作用位点具有泛癌普遍性;进一步分析显示,这些高关联位点在肿瘤与正常组织中的甲基化水平差异显著,具有明确的生物学意义。
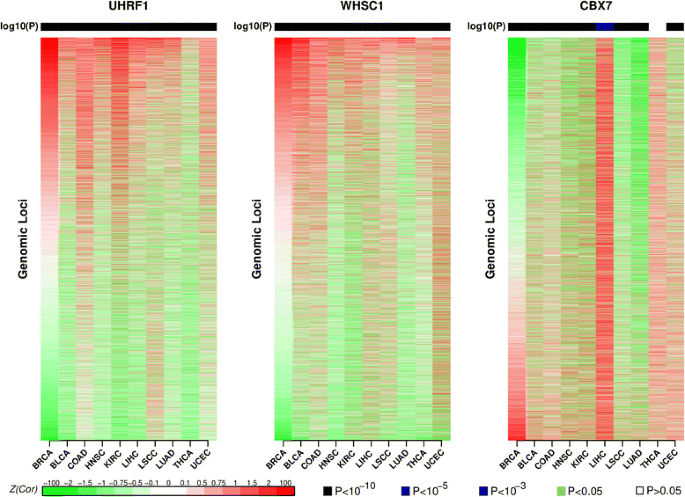
产品关联:文献未提及具体实验产品,领域常规使用R语言的GenomicRanges包进行基因组区域注释,ggplot2包进行热图可视化。
4. Biomarker研究及发现成果
本研究识别的Biomarker为跨癌种一致失调的表观遗传酶,属于功能性Biomarker,筛选逻辑为“泛癌差异表达分析→DNA甲基化指数关联分析→因果网络建模验证”的三级验证体系,涵盖11个潜在癌基因和7个潜在抑癌基因。
这些Biomarker来源于TCGA数据库10种癌种的肿瘤组织样本,验证方法包括泛癌差异表达分析(校正t检验)、表达与甲基化指数的相关性分析(Pearson相关)、因果网络建模(偏相关分析);特异性方面,18个表观遗传酶在至少8种癌种中呈现一致的表达失调模式,与DNA甲基化指数的关联在至少6种癌种中方向一致;敏感性方面,未直接提供ROC曲线数据,但差异表达的统计学显著性极高(P<10^-30),表明其能有效区分正常与癌组织。
核心成果提炼:UHRF1作为核心癌基因,其过表达与全基因组启动子高甲基化显著正相关,跨癌种调控位点高度一致,已被其他研究证实为多种癌症的预后不良标志物(风险比HR值未在本研究中提及);WHSC1作为组蛋白甲基转移酶,首次在泛癌层面揭示其过表达与启动子高甲基化的关联;CBX7作为组蛋白阅读器,首次明确其低表达与开放海区域低甲基化的调控关系;统计学结果显示,18个表观遗传酶的差异表达P值小于10^-30,与DNA甲基化指数的关联P值范围为0.001至10^-24,具有极高的可靠性。这些Biomarker为泛癌通用的表观遗传治疗靶点提供了理论依据,也为癌症的表观遗传诊断提供了潜在的泛癌标志物。
