1. 领域背景与文献引入
文献英文标题:Hi-D: nanoscale mapping of nuclear dynamics in single living cells;发表期刊:Genome Biology;影响因子:未公开;研究领域:核动态学(染色质动力学)。
染色质的空间组织与动态变化是调控基因表达、DNA复制及细胞命运决定的核心机制。早期研究通过“染色体 territories”模型揭示了染色体在核内的分隔模式,Hi-C等基于接触频率的技术进一步解析了染色质的层级结构(如拓扑相关结构域TADs、compartment),但这些方法主要反映静态结构,难以捕捉动态变化。传统的单基因座追踪(如CRISPR标记的单粒子追踪,SPT)虽能解析局部动态,但稀疏的标记无法反映全核范围的动态异质性;早期的bulk动态分析(如intensity-based MSD,iMSD)虽覆盖全局,但分辨率(~μm级)不足以解析亚微米级的局部动态模式。此前研究虽发现染色质动态的空间相干性(如微米级的关联运动),但未能在高分辨率下关联动态参数与核结构或功能。针对这一空白,本研究开发了Hi-D(high-resolution diffusion mapping)方法,结合密集光流(dense optical flow)轨迹重建与贝叶斯模型选择,实现活细胞内全核范围的单像素分辨率动态mapping,旨在揭示染色质动态的空间模式及其与转录活性、染色质压缩的关系。
2. 文献综述解析
作者通过“方法类型”与“科学问题未解决”两个维度对现有研究进行评述:其一,按方法分类,现有研究可分为三类——单基因座追踪(SPT)、基于接触频率的结构分析(Hi-C)、早期bulk动态分析;单基因座追踪的关键结论是染色质动态的异质性(如局部约束运动),但局限性在于稀疏标记导致全局信息缺失;Hi-C等结构分析揭示了染色质的层级结构,但无法反映动态变化;早期bulk动态分析(如correlation spectroscopy)虽能解析全局动态,但分辨率低,无法关联结构与动态。其二,现有研究的未解决问题包括:染色质动态的空间模式(如亚微米级的异质性)、动态与转录活性的因果关系、染色质压缩是否直接调控动态。
本研究的创新点在于:首次将密集光流与贝叶斯推断结合,实现单像素分辨率的全核动态mapping,解决了“全局覆盖”与“高分辨率”的矛盾;通过高斯混合模型(GMM)解卷积动态亚群,首次在高分辨率下关联了动态模式与核功能(如转录活性、核膜结合)。
3. 研究思路总结与详细解析
本研究的整体目标是开发Hi-D方法并解析染色质动态的空间模式与调控机制,核心科学问题包括:① 染色质动态的空间异质性如何分布;② 转录活性如何调控染色质与RNA Pol II动态;③ 染色质压缩与动态是否存在直接关联。技术路线遵循“方法开发→验证→应用→关联”的闭环:首先开发Hi-D方法(密集光流轨迹重建+贝叶斯模型选择),随后通过模拟数据与实验验证方法的准确性与鲁棒性,再将方法应用于不同转录状态(血清饥饿/刺激)、转录抑制(DRB处理)的细胞模型,最后关联动态参数与染色质压缩状态。
3.1 Hi-D方法开发与参数提取
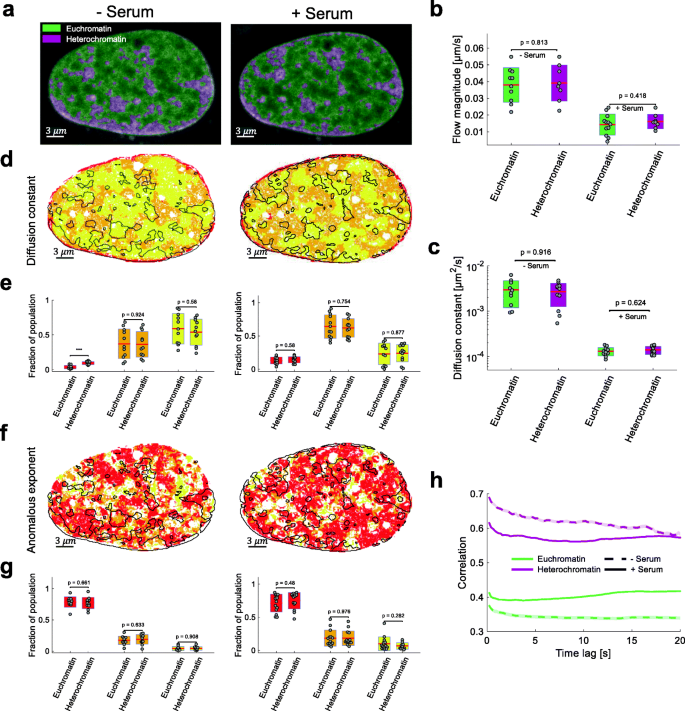
实验目的是开发能解析单像素分辨率染色质动态的方法。方法细节包括两步:首先,对SiR-DNA标记的活细胞共聚焦图像序列(150帧,5 fps)进行密集光流分析,重建每个像素的轨迹(空间分辨率65 nm);随后,对每个像素的均方位移(MSD)曲线,通过贝叶斯推断从5种扩散模型(自由扩散、异常扩散、定向运动、异常扩散+定向运动、自由扩散+定向运动)中选择最优模型,提取扩散常数(D)、异常指数(α)等参数。结果解读:轨迹重建实现了亚像素精度的动态解析,贝叶斯模型选择揭示异常扩散是染色质的主要动态模式(α≈0.3-0.7),参数映射显示动态的空间异质性——扩散常数与异常指数呈马赛克样分布(图1)。实验所用关键产品:SiR-DNA染料(Spirochrome AG)、共聚焦显微镜(Leica DMI8)。
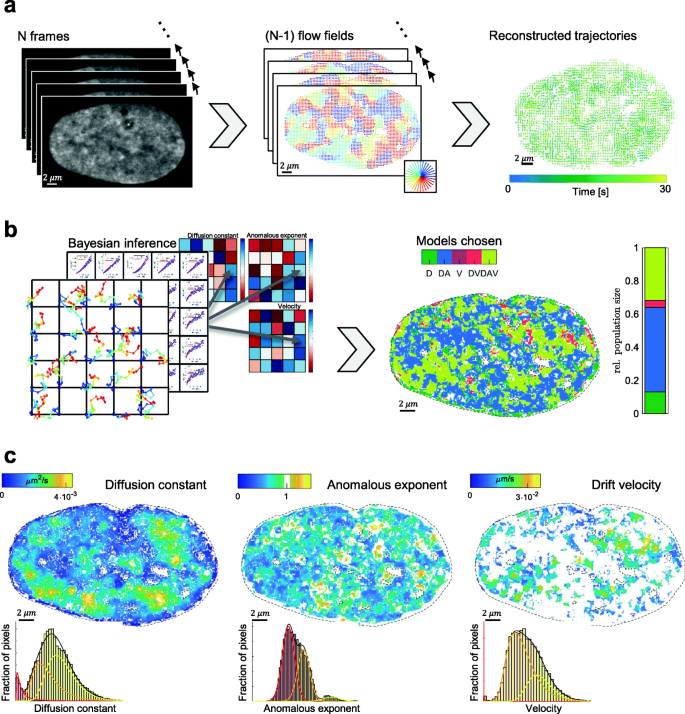
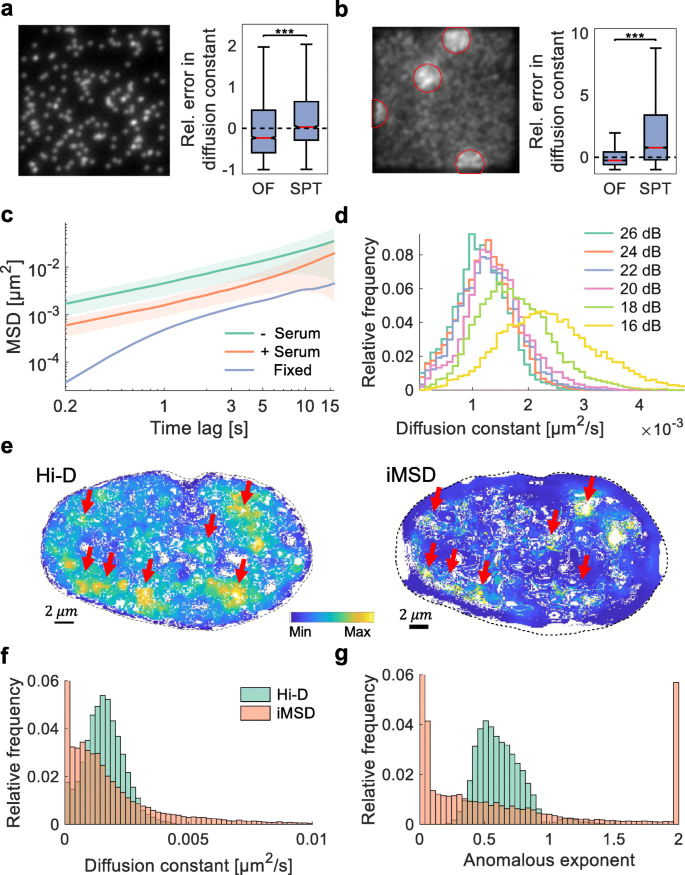
3.2 方法验证与鲁棒性测试
实验目的是验证Hi-D方法的准确性与抗噪声能力。方法细节包括三部分:① 模拟数据对比:生成低密度(0.001/px³)与高密度(0.02/px³)标记的模拟图像,分别用Hi-D与动态多目标追踪(SPT)分析,计算扩散常数的相对误差;② 实验验证:分析甲醛固定细胞(无动态)与活细胞(血清饥饿/刺激)的MSD曲线,计算扩散常数;③ 噪声鲁棒性:向图像添加不同信噪比(SNR=16-26 dB)的噪声,分析参数稳定性。结果解读:模拟数据显示,高密度标记下Hi-D的扩散常数误差显著低于SPT(p<0.001),而低密度标记下SPT更优(图2a-b);活细胞的扩散常数比固定细胞高两个数量级(活细胞D≈10⁻³ μm²/s,固定细胞D≈10⁻⁵ μm²/s,n=13),证实动态解析高于噪声背景(图2c);SNR≥20 dB时,扩散常数分布保持稳定(图2d)。实验所用关键产品:甲醛(固定细胞)、SiR-DNA染料。
3.3 转录活性对染色质与RNA Pol II动态的调控
实验目的是解析转录活性对染色质与RNA Pol II动态的影响。方法细节包括:① 细胞处理:血清饥饿(24 h,抑制转录)、血清刺激(10-15 min,激活转录)、DRB处理(100 μM,抑制RNA Pol II延伸);② 动态分析:用Hi-D解析染色质(SiR-DNA)与RNA Pol II(RPB1-Dendra2融合蛋白)的动态参数,通过GMM将扩散常数分布解卷积为3个mobility亚群(慢、中、快)。结果解读:血清刺激后,染色质的扩散常数全局降低约一个数量级,快亚群比例减少并重新分类为中间亚群(图3a-c);RNA Pol II的扩散常数在转录激活时升高(快亚群比例增加),DRB处理后慢亚群比例增加(约30%升至40%,n=18),表明RNA Pol II被固定在DNA模板上(图4a-c)。这一结果提示,转录活性通过增加DNA-蛋白结合(如转录因子、RNA Pol II复合物)抑制染色质动态,而RNA Pol II的动态与转录状态正相关。实验所用关键产品:DRB(Sigma-Aldrich)、RPB1-Dendra2稳定细胞系(Maxime Dahan实验室馈赠)。
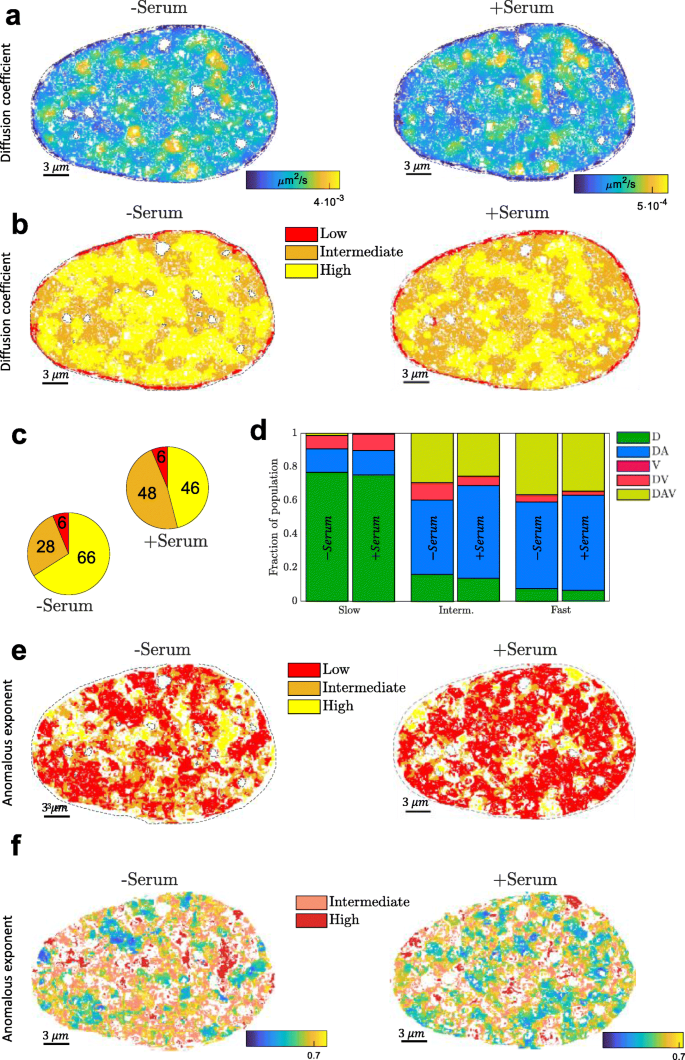
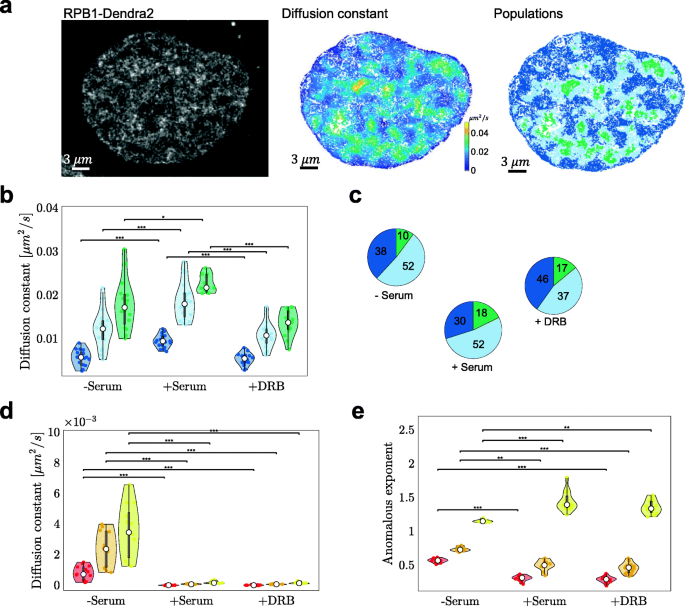
3.4 染色质压缩与动态的关联分析
实验目的是探讨染色质压缩(常染色质/异染色质)与动态的关系。方法细节包括:① 染色质压缩状态鉴定:通过SiR-DNA荧光强度区分常染色质(低强度,开放结构)与异染色质(高强度,压缩结构);② 动态参数关联:分析不同压缩状态下的扩散常数、异常指数及空间自相关性(Moran’s Index)。结果解读:染色质动态与压缩状态无显著相关性——异染色质的扩散常数与常染色质无差异(p>0.05,n=12),但空间自相关性更高(图5b,h)。这表明染色质动态的调控因素是DNA-蛋白相互作用而非压缩程度,而异染色质的高空间相干性可能源于其更密集的自身接触(如DNA环化)。实验所用关键产品:SiR-DNA染料。
4. Biomarker研究及发现成果解析
本研究涉及的Biomarker类型为“染色质动态亚群”(慢、中、快mobility亚群),筛选与验证逻辑为“Hi-D解析全核动态参数→GMM解卷积亚群→空间定位与功能关联”。研究过程:动态亚群来源于活细胞染色质成像数据(SiR-DNA标记),通过GMM对扩散常数分布进行概率解卷积,得到3个具有显著差异的亚群;验证方法包括空间定位(慢亚群集中于核膜区域,对应lamina-associated domains,LADs)、功能关联(血清刺激后快亚群比例降低,与转录抑制相关)。
核心成果:慢亚群可作为核膜结合染色质(LADs)的动态标志物,其比例在转录刺激下保持稳定(约6%核面积,n=12);快亚群与转录活性正相关(血清饥饿时比例高,刺激后降低),可作为转录活跃区域的动态标志物。统计结果:血清饥饿时快亚群比例约为20%,血清刺激后降至10%(n=12,p<0.001);慢亚群比例在两种状态下均约为6%(n=12,p>0.05)。文献未明确提供异常指数的具体统计值,但基于图表趋势推测,异常指数在血清刺激后降低(α≈0.5降至0.3),表明染色质动态更接近缠结聚合物的行为。
综上,本研究开发的Hi-D方法为解析核动态学提供了高分辨率工具,所发现的动态亚群为染色质功能区域的鉴定提供了新型动态标志物,为理解转录调控与核结构的关系奠定了基础。
