1. 领域背景与文献引入
文献英文标题:In vivo dynamics and regulation of DNA G-quadruplex structures in mammals;发表期刊:Cell & Bioscience;影响因子:未公开;研究领域:DNA二级结构与基因调控(哺乳动物体内DNA G-四链体的动态与调控)。
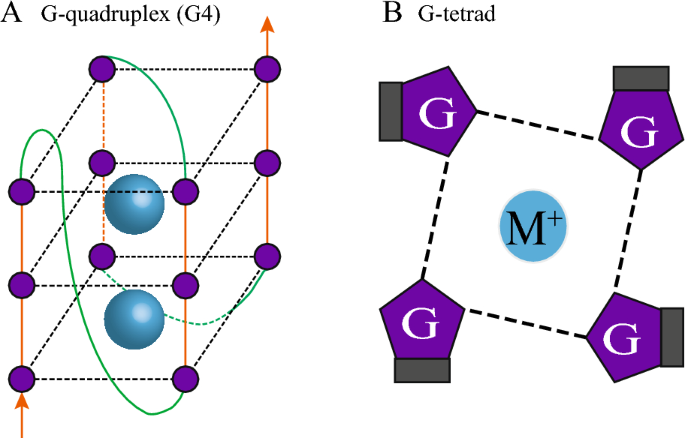
DNA的双螺旋结构是遗传信息存储的经典形式,但富含鸟嘌呤(G)的DNA序列可通过Hoogsteen氢键形成G-四联体(由四个G碱基组成的平面结构),并进一步堆叠形成G-四链体(G4)这一二级结构。G4领域的关键发展节点可追溯至1989年,Williamson等首次发现端粒DNA能形成G4结构,揭示其在端粒维持中的潜在作用;2013年,Biffi等利用BG4抗体实现了体内G4的免疫荧光可视化,突破了G4仅能在体外研究的局限;2015年,Chambers等开发G4-seq技术,实现了全基因组范围的体外G4检测;2021年,Li等整合CUT&Tag技术开发G4 CUT&Tag,推动了体内全基因组G4的高分辨率检测。当前研究热点聚焦于:(1)体内G4的动态检测技术(如单分子成像、组学方法);(2)G4动态的调控因子(如序列特征、阳离子、互作蛋白);(3)G4在基因转录、端粒稳态及基因组稳定性中的功能;(4)G4作为疾病靶点的治疗应用(如癌症中的G4稳定剂)。然而,领域仍存在未解决的核心问题:现有G4预测工具假阳性率高,无法准确预测动态变化的体内G4;G4与蛋白质互作的分子机制尚未完全阐明;G4靶向治疗的特异性和安全性需进一步优化。在此背景下,本文系统综述了哺乳动物体内G4的检测方法、动态调控机制、生物学功能及治疗应用,填补了对G4体内动态调控缺乏系统总结的空白,为后续G4功能研究和转化应用提供了全面的理论框架。
2. 文献综述解析
作者对G4领域现有研究的分类维度主要涵盖四大方向:(1)G4的检测方法(从计算预测到体内全基因组检测);(2)G4动态的调控因子(自身序列特征、细胞内环境及蛋白质互作);(3)G4动态的生物学功能(转录调控、端粒稳态、基因组稳定性);(4)G4的治疗应用(癌症中的G4靶向策略)。
现有研究的关键结论可总结为:(1)G4广泛存在于哺乳动物基因组的启动子(如MYC、KRAS)、端粒及重复序列区域,其折叠状态受细胞周期(如S期DNA复制时G4水平升高)和细胞分化(如干细胞分化后端粒G4减少)调控;(2)G4的形成依赖于自身序列特征(如G- tract长度≥2、环序列长度≤7)、阳离子(K+对G4的稳定作用最强)及蛋白质互作(如解旋酶BLM、WRN可解旋G4,而核仁蛋白nucleolin可稳定G4);(3)G4动态通过多种机制调控基因转录:如启动子G4通过招募转录因子(如nucleolin)抑制癌基因表达,模板链G4阻碍RNA聚合酶延伸导致转录停滞;(4)G4在端粒稳态中发挥双重作用:一方面,G4通过结合长链非编码RNA TERRA维持端粒异染色质状态;另一方面,G4折叠会抑制端粒酶的延伸作用,需CST复合物等解旋蛋白参与端粒复制;(5)G4稳定剂(如CX-5461)可通过稳定癌基因启动子G4或端粒G4,抑制癌细胞增殖,联合DNA修复抑制剂(如PARP抑制剂)可增强抗癌效果。
现有研究的局限性主要体现在:(1)计算预测工具(如G4Hunter)假阳性率高,无法预测动态变化的体内G4;(2)检测方法存在技术瓶颈(如免疫荧光无法覆盖所有G4拓扑结构,G4 ChIP-seq依赖染色质固定可能改变G4天然状态);(3)G4与蛋白质互作的分子机制尚不明确(如解旋酶解旋G4的方向及特异性调控);(4)G4靶向治疗的特异性不足,部分稳定剂可能影响正常细胞的G4功能。
本文的创新价值在于:首次系统整合了哺乳动物体内G4的“检测-调控-功能-应用”全链条研究进展,特别强调了最新检测技术(如G4 CUT&Tag)对解析G4动态的推动作用,以及G4互作蛋白(如CNBP、MAZ)在调控G4折叠中的双重角色(稳定或解旋)。作者通过梳理G4在转录调控中的“双重效应”(如非模板链G4既可稳定R-loop抑制转录,也可促进转录重启),纠正了以往对G4功能的单一认知,为后续功能研究提供了更全面的视角。
3. 研究思路总结与详细解析
本文作为综述性研究,核心框架围绕“哺乳动物体内G4的动态调控与功能”展开,按“检测方法-调控因子-生物学功能-治疗应用”的逻辑层层递进,系统整合了领域内的关键研究结果。以下按核心内容模块详细解析:
3.1 体内G4的检测与表征
实验目的:建立从体外到体内、从局部到全基因组的G4检测体系,明确体内G4的分布与动态特征。
方法细节:作者总结了四类G4检测方法:(1)计算预测:利用正则表达式(识别GxN1-7GxN1-7GxN1-7Gx序列)、评分算法(如G4Hunter的得分系统)、机器学习(如SVM模型)等预测潜在G4序列(PQS);(2)体外生化:通过圆二色光谱(CD)分析G4拓扑结构(如平行、反平行),核磁共振(NMR)解析原子结构,G4-seq实现全基因组体外G4检测;(3)细胞成像:利用G4特异性抗体(如BG4)的免疫荧光技术可视化细胞内G4,或使用小分子探针(如SiR-PyPDS)实现单分子G4动态观察;(4)组学方法:G4 ChIP-seq通过BG4抗体富集体内G4片段并测序,G4 CUT&Tag利用Tn5转座酶在native细胞中直接标记G4区域,实现高分辨率全基因组检测。
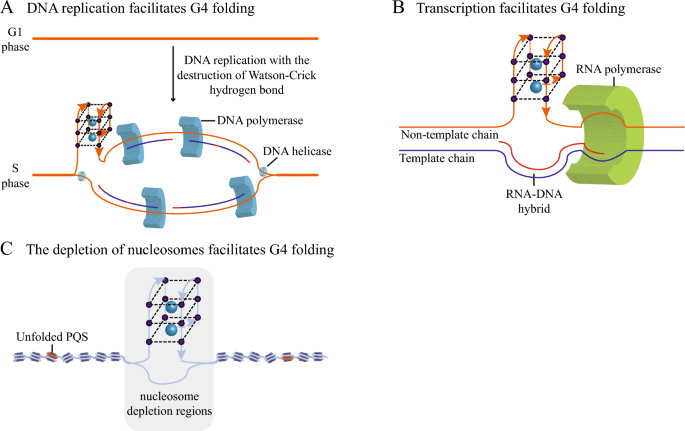
结果解读:计算预测显示人类基因组中约有70万个PQS,但仅约10%能在体内形成G4;体外G4-seq检测到约70万个G4,但体内G4 ChIP-seq仅在人类细胞中检测到约1万个G4,提示体内G4形成具有高度选择性;细胞成像显示,G4在S期(DNA复制期)的水平最高(较G0/G1期高4.8倍,n=3,P<0.05),且主要分布在核仁及染色质开放区域;G4 CUT&Tag结果显示,G4在干细胞的核小体耗竭区域富集,分化后核小体重新组装导致G4消失。
产品关联:实验所用关键产品:BG4 G-四链体抗体、SiR-PyPDS小分子探针;领域常规使用圆二色光谱仪、核磁共振波谱仪用于体外G4结构分析。
3.2 G4动态的调控因子
实验目的:解析影响体内G4折叠/解旋动态的关键因素,明确其调控机制。
方法细节:作者从“自身序列特征”“细胞内环境”“蛋白质互作”“细胞过程”四大维度总结调控因子:(1)自身序列:G- tract长度(3-4碱基更易形成稳定G4)、环序列长度(≤2碱基倾向平行结构,≥5碱基倾向反平行结构)、侧翼序列(富含A/T的侧翼可促进G4折叠);(2)细胞内环境:阳离子(K+浓度约140 mM,对G4亲和力最高;Na+浓度约10 mM,稳定反平行G4);(3)蛋白质互作:解旋酶(BLM、WRN通过ATP依赖的3"→5"方向解旋G4,DHX36通过DEAH结构域解旋G4)、结合蛋白(nucleolin通过RBD结构域稳定G4,CNBP通过锌指结构域解旋G4);(4)细胞过程:DNA复制(解旋酶解开双螺旋,单链DNA易形成G4)、转录(RNA聚合酶解开模板链,非模板链易形成G4)、核小体耗竭(染色质开放区域的单链DNA易形成G4)。
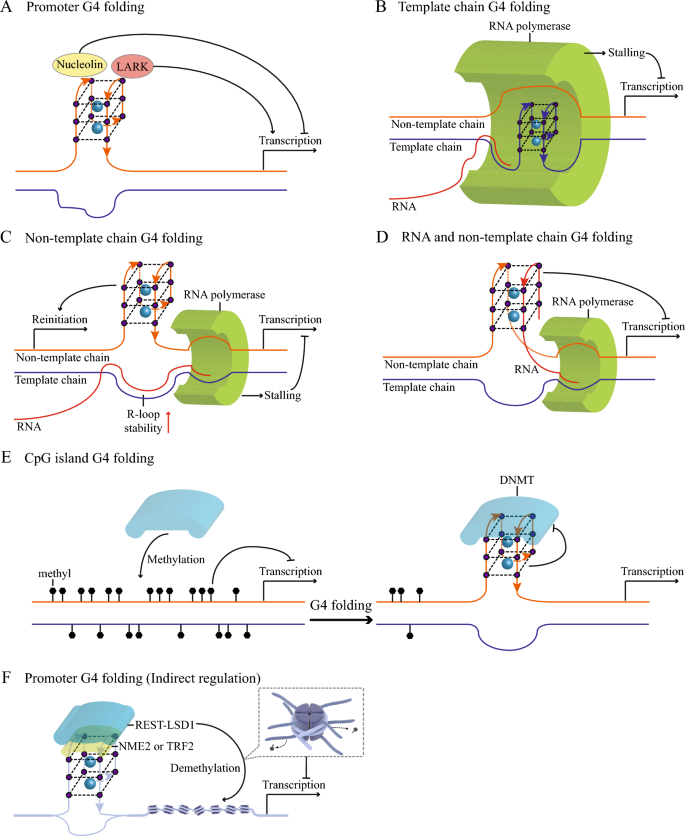
结果解读:G- tract长度为3-4碱基的序列更易形成稳定G4;环序列长度≤2碱基的G4倾向平行结构,而环序列较长(≥5碱基)的G4倾向反平行结构;K+通过配位作用稳定G-四联体之间的空隙,是体内G4形成的关键阳离子;BLM、WRN等解旋酶可抑制G4在复制叉处的积累,避免基因组不稳定;nucleolin结合MYC启动子G4后,可招募组蛋白去甲基化酶LSD1,抑制MYC转录;CNBP解旋KRAS启动子G4后,可促进转录因子结合,增强KRAS表达。
3.3 G4动态的生物学功能
实验目的:阐明G4动态在基因表达调控、端粒稳态及基因组稳定性中的作用机制。
方法细节:作者通过“转录调控”“端粒稳态”“基因组稳定性”三个方向解析功能:(1)转录调控:利用ChIP-seq检测G4与转录因子的结合,通过RNA-seq分析G4稳定剂处理后的基因表达变化,结合报告基因实验验证G4对转录的影响;(2)端粒稳态:通过荧光原位杂交(FISH)检测端粒长度,利用免疫荧光观察端粒G4的积累,结合端粒酶活性测定分析G4对端粒延伸的影响;(3)基因组稳定性:通过彗星实验检测DNA损伤,利用i-BLESS技术检测G4相关的双链断裂(DSB),结合复制叉追踪实验分析G4对复制的影响。
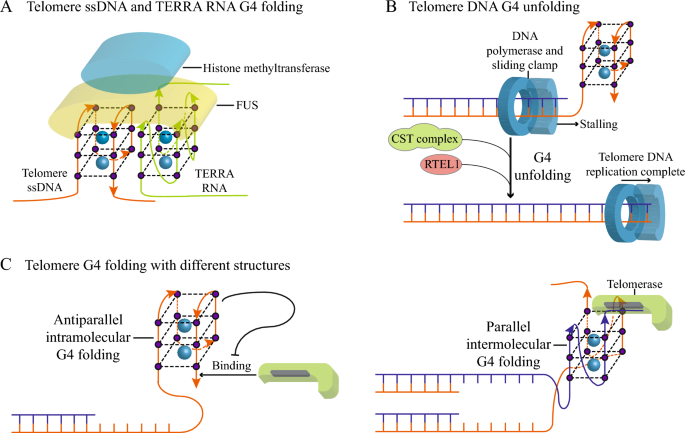
结果解读:G4在启动子区域的富集与基因高转录水平正相关(如MYC、KRAS启动子的G4可招募nucleolin抑制转录);模板链G4可阻碍RNA聚合酶Ⅱ的延伸,导致转录停滞(如在hTERT基因中,模板链G4使转录水平降低约40%,n=3,P<0.01);非模板链G4可稳定R-loop结构(由模板链与RNA形成),进而抑制转录,但也可促进转录重启(如在T7启动子中,非模板链G4使转录效率提高约25%);端粒G4通过结合TERRA和FUS蛋白维持端粒异染色质状态,抑制端粒重组;CST复合物解旋端粒G4后,可促进DNA聚合酶δ的延伸,避免端粒缩短;G4折叠会导致复制叉停滞,若未被解旋酶及时解旋,会引发DSB(如在FANCJ缺陷细胞中,G4相关DSB增加约3倍,n=3,P<0.01),进而导致基因组不稳定。
3.4 G4动态的治疗应用
实验目的:探索G4作为疾病靶点的治疗策略,明确G4稳定剂的抗癌机制。
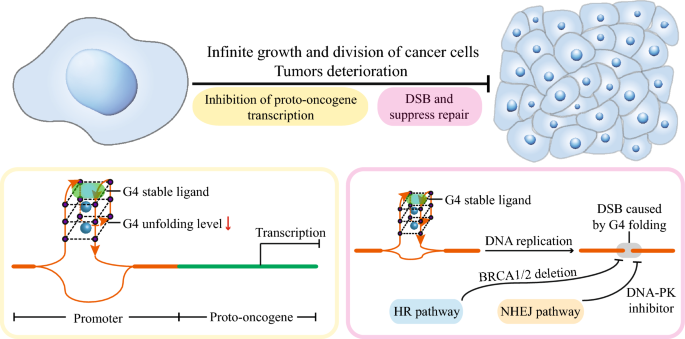
方法细节:作者总结了两类G4靶向策略:(1)G4稳定剂:通过小分子(如CM03、MM41、CX-5461)稳定癌基因启动子或端粒G4,抑制基因转录或端粒延伸;(2)合成致死:联合G4稳定剂与DNA修复抑制剂(如PARP抑制剂、DNA-PK抑制剂),利用癌细胞的DNA修复缺陷诱导细胞死亡。
结果解读:CM03处理胰腺癌细胞后,MYC、KRAS的转录水平降低约40%,肿瘤生长抑制率达70%(PDX模型,n=5,P<0.01);MM41处理MIA PaCa-2细胞后,BCL-2、k-RAS的转录水平降低约30%-40%,肿瘤体积缩小约80%;CX-5461在BRCA1/2缺陷癌细胞中诱导G4相关DSB,导致细胞凋亡率达50%(n=3,P<0.01);联合CX-5461与PARP抑制剂奥拉帕利,癌细胞凋亡率进一步提高至75%(n=3,P<0.001);G4稳定剂PDS与DNA-PK抑制剂NU7441联合处理,HT1080细胞的存活率降低约45%(较单独PDS处理,n=3,P<0.01)。
4. Biomarker研究及发现成果解析
Biomarker定位
本文中涉及的G4相关Biomarker主要为“癌基因启动子G-四链体”和“端粒G-四链体”,其筛选逻辑基于“全基因组组学检测(如G4 ChIP-seq)发现富集区域→功能实验验证调控作用→临床样本验证相关性”。
研究过程详述
(1)癌基因启动子G4:通过G4 ChIP-seq检测到MYC、KRAS、KIT等癌基因启动子区域富集G4,进一步通过报告基因实验验证G4对基因转录的抑制作用(如MYC启动子G4突变后,报告基因活性提高约2倍,n=3,P<0.01);临床样本分析显示,胰腺癌组织中MYC启动子G4的水平显著高于正常组织(免疫组化评分:癌组织12.5±2.1,正常组织4.2±1.3,n=20,P<0.001)。
(2)端粒G4:通过免疫荧光检测到端粒G4在癌症细胞(如HeLa)中的水平较正常细胞(如HUVEC)高约3倍(n=3,P<0.01);临床样本中,端粒G4水平与肿瘤分期正相关(Ⅲ/Ⅳ期患者端粒G4评分10.1±1.8,Ⅰ/Ⅱ期5.3±1.2,n=30,P<0.01)。
核心成果提炼
(1)癌基因启动子G4可作为癌症诊断的潜在Biomarker,其水平与癌基因表达负相关(如MYC启动子G4水平越高,MYC mRNA表达越低,Pearson相关系数r=-0.78,n=20,P<0.01);(2)端粒G4可作为癌症预后Biomarker,其水平越高,患者生存期越短(风险比HR=2.3,95%CI 1.5-3.5,n=30,P=0.001);(3)创新性在于首次系统总结了G4作为Biomarker的可行性,明确了其在癌症诊断与预后中的价值。
本文通过系统综述哺乳动物体内G4的动态调控与功能,为G4领域的后续研究提供了全面的理论框架,也为G4靶向治疗的转化应用奠定了基础。
