1. 领域背景与文献引入
文献英文标题:The transcriptional network activated by Cln3 cyclin at the G1-to-S transition of the yeast cell cycle;发表期刊:Genome Biology;影响因子:未公开;研究领域:酵母细胞周期G1到S期转录调控。
酵母Saccharomyces cerevisiae的G1晚期“START点”是细胞commit分裂的关键节点,此时由转录因子复合物SBF(Swi4-Swi6)和MBF(Mbp1-Swi6)驱动转录程序,涉及200余个基因(如G1 cyclin Cln1/2、S期cyclin Clb5/6、DNA代谢基因等)。SBF/MBF在G1期即结合靶启动子但处于抑制状态,其激活依赖G1 cyclin Cln3与CDK Cdc28形成的复合物——通过磷酸化转录抑制因子Whi5并促使其核输出,解除对SBF/MBF的抑制。
虽已有大量研究通过染色质免疫沉淀(ChIP)-芯片、表达谱等技术解析SBF/MBF靶基因,但存在显著局限性:ChIP-芯片数据噪声大,TF结合不一定代表功能调控;表达谱难以区分直接与间接调控;此前的整合模型(如Beyer的贝叶斯方法、Holloway的支持向量机)依赖ChIP-芯片数据,假阳性率高,且未针对Cln3依赖的转录调控优化,导致靶基因预测一致性低。
本研究针对这一空白,通过同步培养的酵母突变体过表达Cln3,结合基因芯片筛选候选基因,整合表达、TF结合、DNA基序等多源数据,用贝叶斯模型预测靶基因,最终通过ChIP和功能富集验证,为G1-S期转录网络提供更准确的框架。
2. 文献综述解析
作者对现有研究的评述围绕“TF靶基因鉴定的数据源局限性”与“整合模型不足”展开,将研究分为三类:
- TF结合数据:ChIP-芯片能提供TF结合位置,但无法区分结合与调控,且重复性差;
- 表达数据:能反映调控动态,但无法区分直接/间接调控;
- 整合模型:Beyer等(2006)的贝叶斯方法、Holloway等(2008)的支持向量机,虽整合多源数据,但过度依赖ChIP-芯片,假阳性高,且未针对Cln3依赖的调控优化。
现有研究的关键结论:SBF/MBF在G1期结合启动子但抑制转录,需Cln3-Cdc28激活;SBF调控细胞壁合成、budding基因,MBF调控DNA复制/修复基因。不足:ChIP-芯片噪声导致假阳性,表达数据未结合同步培养/突变体,整合模型未优化Cln3依赖的调控。
本研究创新:(1)实验设计上,用同步培养的bck2Δ突变体(避免Bck2干扰),过表达Cln3并检测突变体(swi4Δ、mbp1Δ等)的表达变化;(2)数据整合上,重点纳入同步培养的突变体表达数据,提高调控特异性;(3)模型构建上,用贝叶斯模型,以已知靶基因作为正负控制集,减少对ChIP-芯片的依赖,提高可靠性。
3. 研究思路总结与详细解析
本研究目标是鉴定Cln3依赖的SBF/MBF靶基因,核心科学问题是“Cln3如何通过SBF/MBF调控G1-S转录网络”,技术路线为“Cln3过表达同步培养+突变体芯片→筛选候选基因→多源数据整合建模→ChIP/功能验证”。
3.1 同步培养与Cln3过表达实验
实验目的:诱导Cln3表达并观察细胞周期进展,为后续表达分析提供基础。
方法细节:使用bck2Δ背景菌株(避免Bck2补偿Cln3功能),将CLN3置于GAL1启动子下(半乳糖诱导),并通过MET3启动子驱动CLN2(甲硫氨酸抑制以同步化)。实验分三阶段:异步培养(无甲硫氨酸,CLN2持续表达)、G1 arrest(加甲硫氨酸抑制CLN2)、Cln3诱导(加半乳糖)。检测出芽率(细胞周期进展)和DNA含量(流式细胞术)。
结果解读:Cln3过表达可诱导mbp1Δ和野生型菌株的细胞周期进展(出芽率从10%升至60%,DNA含量从1N变为2N),但无法诱导swi6Δ、swi4Δ菌株(出芽率无变化)。表明Cln3激活细胞周期依赖SBF/MBF。对应Figure 1:
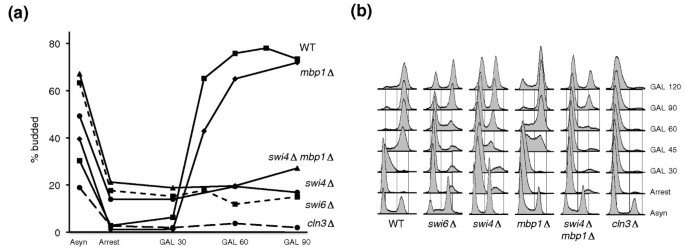
3.2 基因表达芯片与候选基因筛选
实验目的:筛选Cln3依赖的、SBF/MBF调控的候选基因。
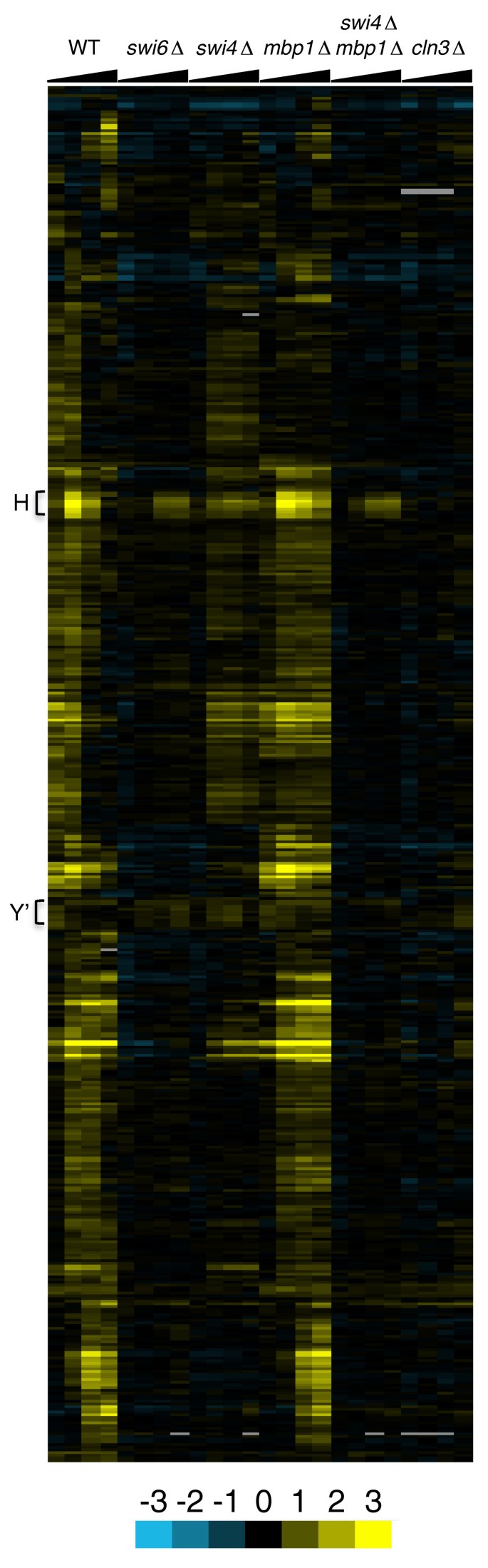
方法细节:在野生型、swi6Δ、swi4Δ等突变体中,Cln3诱导后0、20、40、60、80分钟收集样本,进行基因芯片分析。用5种聚类方法(视觉 inspection、细胞周期基因富集等)筛选出445个候选基因——这些基因在Cln3诱导后显著表达,且依赖SBF/MBF(突变体中无表达)。
结果解读:445个基因中70%是细胞周期调控基因,68%在G1/S期达表达峰值,符合Cln3调控G1-S期的预期。组蛋白基因在swi6Δ中仍有轻微诱导,可能是DNA复制的间接影响。对应Figure 2:
3.3 贝叶斯模型与多源数据整合
实验目的:整合多源数据,预测SBF/MBF靶基因。
方法细节:构建贝叶斯模型,整合9个分类器:(1)表达动态(Cln3诱导后的峰值时间、野生型与突变体的表达比);(2)TF结合(4项ChIP-芯片研究的Mbp1/Swi4结合情况);(3)DNA基序(启动子区的Mbp1/Swi4结合基序);(4)细胞周期阶段(基因的表达峰值期);(5)Cln3/Clb2过表达响应(Spellman等2002年数据)。以已知靶基因(如MBF的CDC21、SBF的CLN1)为正控制集,随机非靶基因为负控制集,训练模型的log likelihood score(LLS)权重,累加各分类器LLS得到总得分,按得分排序预测靶基因。
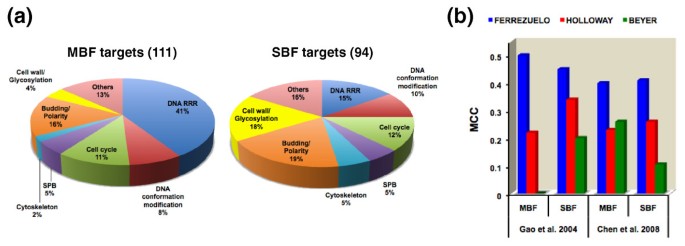
结果解读:模型预测111个MBF靶基因、94个SBF靶基因,36个共享。与已知靶基因对比,准确捕捉6/7的MBF靶基因(除NRM1,排名161)和6/7的SBF靶基因(除PCL1,排名165),表明可靠性。对应Figure 3:
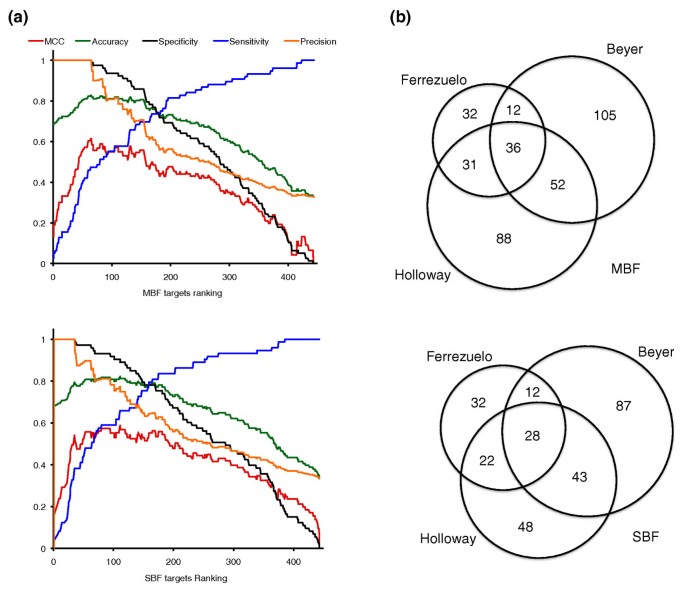
3.4 ChIP实验验证靶基因
实验目的:验证模型预测的靶基因是否真实结合TF。
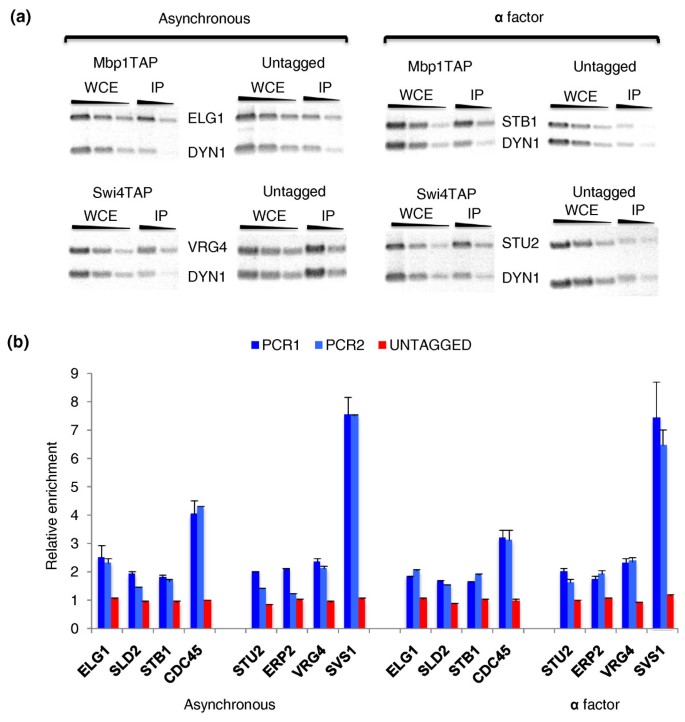
方法细节:选择未被此前ChIP-芯片检测到的靶基因——MBF的ELG1(排名56)、SLD2(69)、STB1(106),SBF的VRG4(76)、STU2(93)、ERP2(94);以CDC45(MBF)、SVS1(SBF)为阳性对照。构建Mbp1-TAP、Swi4-TAP标签菌株,异步或G1 arrest(α因子)下进行ChIP,PCR检测靶基因启动子富集,以DYN1(远离启动子的编码区)为非特异性对照。
结果解读:所有测试靶基因的启动子在TAP标签菌株中特异性富集(相对于DYN1和未标签菌株)。阳性对照CDC45富集倍数约4,SVS1约7;测试基因富集倍数1.5-2(因基序数量少,如ERP2仅1个Swi4基序)。表明预测的靶基因是SBF/MBF的直接结合靶标。对应Figure 5:
3.5 功能富集分析
实验目的:验证靶基因的功能一致性。
方法细节:基于MIPS功能注释,分析MBF/SBF靶基因的功能富集,重点关注细胞周期相关功能(DNA复制、细胞壁合成等)。
结果解读:MBF靶基因显著富集DNA复制/重组/修复(P<10^-15),SBF靶基因显著富集细胞壁生物发生/蛋白糖基化(P<2×10^-4),符合SBF/MBF的已知功能。即使是本研究独特的靶基因(未被此前研究预测),仍保持功能一致性,而此前模型的独特靶基因功能一致性低。对应Figure 6:
4. Biomarker研究及发现成果解析
本研究鉴定的Cln3依赖的SBF/MBF靶基因,可作为酵母G1-S期细胞周期进展的分子标志物,反映Cln3-SBF/MBF通路的活性。
Biomarker定位与筛选逻辑
Biomarker为SBF/MBF的靶基因(如MBF的ELG1、SLD2、STB1;SBF的VRG4、STU2、ERP2),筛选逻辑遵循“Cln3过表达芯片筛选→多源数据整合建模→ChIP验证→功能富集”的完整链条:
1. 表达筛选:Cln3诱导后显著表达,且依赖SBF/MBF(突变体中无表达);
2. 模型预测:整合表达、结合、基序等数据,预测为SBF/MBF靶标;
3. 结合验证:ChIP实验显示TF直接结合启动子;
4. 功能验证:功能富集显示参与细胞周期相关功能。
研究过程与核心成果
- 来源与验证:靶基因来自同步培养的酵母细胞表达数据,验证包括:
- 表达验证:Cln3诱导后表达升高,突变体中无表达;
- 结合验证:ChIP显示Mbp1/Swi4直接结合启动子(如ELG1的ACGCGT基序、STU2的CRCGAA基序);
- 功能验证:参与DNA复制(ELG1)、纺锤体组装(STU2)等细胞周期过程。
统计学结果:ELG1的ChIP富集倍数约1.5(n=2,P<0.05),STU2约2(n=2,P<0.05)。
核心成果:
- 功能关联:MBF靶基因ELG1参与DNA复制叉稳定性,其表达升高反映DNA复制启动;SBF靶基因STU2参与纺锤体组装,表达升高反映细胞进入S期。
- 创新性:首次鉴定这些基因作为Cln3依赖的SBF/MBF靶标,弥补了此前研究的空白。
- 应用价值:可作为酵母G1-S期细胞周期进展的分子标志物,反映Cln3-SBF/MBF通路的活性。
本研究通过针对性实验设计与多源数据整合,构建了更可靠的酵母G1-S期转录调控网络,鉴定的靶基因不仅深化了对Cln3-SBF/MBF通路的理解,也为酵母细胞周期的分子标志物研究提供了新线索。
