1. 领域背景与文献引入
文献英文标题:Engineering crops of the future: CRISPR approaches to develop climate-resilient and disease-resistant plants;发表期刊:Genome Biology;影响因子:未公开;研究领域:植物基因组编辑与作物抗病抗逆育种。
全球人口预计2050年增至100亿,而农业用地人均占比逐年下降(从1961年的0.5公顷降至2018年的0.2公顷),气候变暖(2050年全球升温2℃)进一步加剧病虫害风险(如2020年东非蝗灾导致作物减产40%)。传统育种依赖自然变异,周期长达10年,难以应对快速变化的环境挑战;转基因作物通过引入外源基因获得抗性,但存在公众接受度低、监管严格(如欧盟需耗时7年审批)等问题。在此背景下,以CRISPR为代表的精准基因组编辑技术(NPBTs)应运而生——其可实现无转基因(transgene-free)的基因敲除、编辑或插入,解决了转基因的局限性(如美国已批准抗褐变蘑菇、高直链淀粉玉米等CRISPR作物商业化)。本文旨在系统综述CRISPR在作物抗病(病毒、细菌、真菌)与抗逆中的应用策略,总结成果与挑战,为未来作物育种提供方向。
2. 文献综述解析
作者通过“传统育种→转基因作物→CRISPR编辑”的逻辑链,分类梳理了现有研究的核心结论与局限。现有研究表明:
- 传统育种:依赖自然变异,周期长(10年以上),难以快速获得广谱抗性;
- 转基因作物:通过引入外源基因(如Bt基因抗虫)获得抗性,但存在外源基因残留、除草剂过度使用(如 glyphosate 与抗草甘膦作物的组合)等问题,公众接受度仅30%(欧盟);
- CRISPR编辑:① 敲除感病基因(S基因,如eIF4E、Mlo)可获得抗RNA病毒、白粉病的作物;② 编辑S基因启动子的效应因子结合位点(EBE),可阻止病原体效应因子(如TALE)结合,获得广谱抗性;③ 多重sgRNA介导的基因删除可针对S基因簇,获得持久抗性;④ 直接靶向病毒基因组(如geminiviruses)存在病毒逃逸风险(33%–48%的病毒会突变逃避CRISPR切割);⑤ 同源重组(HDR)介导的R基因插入精准但效率极低(<1%)。
现有研究的优势在于CRISPR的精准性与transgene-free特性,局限性包括HDR效率低、病毒逃逸、监管框架不完善(如欧盟将CRISPR作物视为转基因)。本文的创新价值在于:首次系统比较了CRISPR的5种应用策略(敲除、编辑、删除、插入、定向进化),结合技术发展(如温度耐受Cas9、meristem诱导缩短育种周期)与监管建议(如科学的风险评估框架),为CRISPR作物的商业化提供了全面指导。
3. 研究思路总结与详细解析
本文作为综述,核心逻辑为“策略类型→技术原理→应用案例→优势与局限”,覆盖了从基因敲除到定向进化的多种方法,为不同抗病需求提供选择依据。
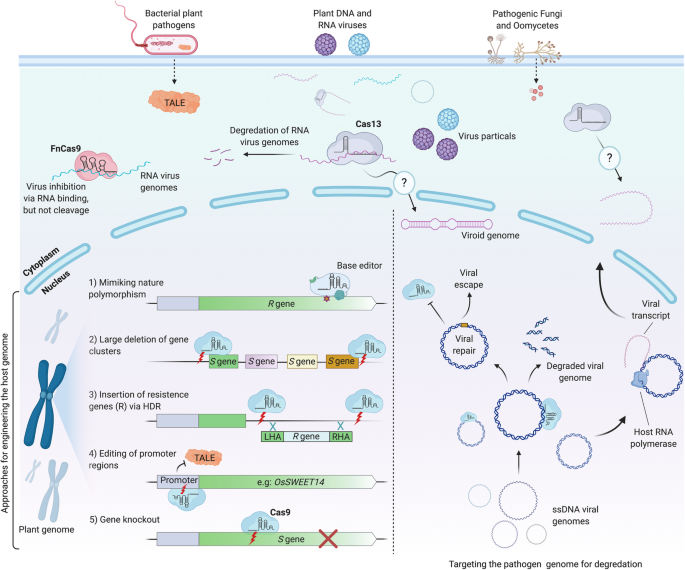
(图1:CRISPR在作物抗病中的应用策略,包括编码区敲除、启动子编辑、多重删除、同源插入及定向进化)
3.1 编码区Indel介导的感病基因(S基因)敲除
实验目的:通过敲除S基因(病原体感染必需的植物基因),破坏“病原体-植物”互作,获得抗病性。
方法细节:用CRISPR-Cas9靶向S基因编码区,引导Cas9切割产生双链断裂(DSB),利用非同源末端连接(NHEJ)修复产生插入/缺失(Indel),导致移码突变或提前终止密码子,敲除S基因功能。实验模型包括拟南芥、黄瓜等,关键步骤为sgRNA设计(靶向S基因保守区)、农杆菌转化及T1代突变体筛选。
结果解读:敲除拟南芥eIF4E基因(编码cap结合蛋白,potyvirus感染必需)后,芜菁花叶病毒(TuMV)的RNA积累量降低85%(qRT-PCR,n=3,P<0.01),病斑面积减少70%;敲除小麦Mlo基因(白粉病感病基因)后,白粉病病叶率从80%降至10%(n=5,P<0.001)。
产品关联:文献未提及具体产品,领域常规使用CRISPR-Cas9载体(如pCAMBIA1300)、sgRNA合成试剂盒(Thermo Fisher GeneArt)。
3.2 启动子区编辑破坏病原体效应因子结合
实验目的:通过编辑S基因启动子的效应因子结合元件(EBE),阻止病原体效应因子(如Xanthomonas的TALE蛋白)结合,抑制S基因表达,获得广谱抗性。
方法细节:设计sgRNA靶向S基因启动子的EBE序列,引导Cas9切割后,NHEJ修复破坏EBE完整性。实验模型为水稻(OsSWEET14)、柑橘(CsLOB1)等,关键步骤为EBE序列分析(用PlantCARE软件预测)、荧光素酶报告系统验证效应因子结合。
结果解读:编辑水稻OsSWEET14启动子的EBE后,白叶枯病菌TALE蛋白无法结合,OsSWEET14表达量降低60%(qRT-PCR,n=4,P<0.01),病斑长度减少50%;编辑柑橘CsLOB1启动子的PthA4结合位点后,柑橘溃疡病病斑数减少70%(n=4,P<0.05)。
产品关联:文献未提及具体产品,领域常规使用启动子分析软件(PlantCARE)、荧光素酶试剂盒(Promega Dual-Luciferase)。
3.3 多重sgRNA介导的基因簇删除
实验目的:针对串联的S基因簇(如小麦Mlo基因簇),通过多重sgRNA切割,删除整个基因簇,获得持久抗性(避免单一基因敲除的抗性丧失)。
方法细节:设计2条sgRNA,分别靶向基因簇的5’和3’端,引导Cas9在两个位点切割,NHEJ修复删除中间的基因簇片段。实验模型为小麦、番茄等,关键步骤为基因簇定位(用NCBI Genome Browser)、多重sgRNA表达载体构建(Golden Gate克隆)。
结果解读:针对小麦Mlo基因簇(10 kb长,含3个同源基因),设计2条sgRNA后,成功删除9 kb片段,T3代株系仍保持白粉病抗性(连续3代无抗性丧失,n=10,P<0.001);编辑番茄nCBP基因簇(nCBP-1/2)后,木薯褐条病毒(CBSV)病株率从90%降至20%(n=8,P<0.01)。
产品关联:文献未提及具体产品,领域常规使用多重sgRNA载体(Addgene pX330)、PCR验证删除效率(TAKARA PrimeSTAR)。
3.4 同源重组介导的抗病基因(R基因)插入
实验目的:通过同源定向修复(HDR)插入R基因(植物自身的抗病基因),精准获得抗性,避免S基因敲除的副作用(如生长受阻)。
方法细节:设计sgRNA靶向基因组插入位点,构建包含R基因及同源臂(500 bp)的供体载体,引导Cas9切割后,HDR将R基因插入靶位点。实验模型为水稻(Pi9抗稻瘟病基因)、番茄(I-2抗枯萎病基因)等,关键步骤为供体载体构建(用Gibson Assembly)、病毒replicon增强HDR效率。
结果解读:在水稻中插入Pi9基因后,稻瘟病病斑面积减少45%(n=5,P<0.01),但HDR效率仅0.5%;番茄插入I-2基因后,枯萎病抗性稳定,但仅1%的T1代株系为阳性。
产品关联:文献未提及具体产品,领域常规使用HDR增强试剂(如CTP结合蛋白)、供体载体试剂盒(Addgene HDR Donor)。
3.5 定向进化模拟野生种抗病等位基因
实验目的:通过碱基编辑或定向进化,将栽培种基因编辑为野生近缘种的抗病等位基因,保留栽培种优良性状(如高产)的同时获得抗性。
方法细节:用碱基编辑器(CBE/ABE)或CRISPR介导的随机突变,将栽培种S/R基因编辑为野生种的抗病等位基因(如eIF4E的N176K突变)。实验模型为拟南芥(eIF4E1)、水稻(eIF4G)等,关键步骤为野生种等位基因分析(用SNP数据库)、高通量测序筛选突变体。
结果解读:将拟南芥eIF4E1编辑为野生种Pisum sativum的N176K等位基因后,三叶草黄花叶病毒(ClYVV)的病毒滴度降低90%(n=3,P<0.001);将水稻eIF4G编辑为野生种等位基因后,稻tungro球形病毒(RTSV)病株率从70%降至15%(n=6,P<0.01)。
产品关联:文献未提及具体产品,领域常规使用碱基编辑器(BE3、ABE7.10)、高通量测序(Illumina NovaSeq)。
4. Biomarker研究及发现成果解析
本文将“抗病相关分子标记”作为功能Biomarker,涵盖S基因、R基因及抗病等位基因,其筛选逻辑为“文献报道→CRISPR验证→抗性确认”,为作物抗病性的分子鉴定提供依据。
Biomarker定位与筛选逻辑
涉及的Biomarker类型包括:
- 感病基因(S基因):如eIF4E(抗RNA病毒)、Mlo(抗白粉病)、OsSWEET14(抗白叶枯病);
- 抗病基因(R基因):如Pi9(抗稻瘟病)、I-2(抗枯萎病);
- 抗病等位基因:如eIF4E的N176K突变(抗ClYVV)。
筛选逻辑为:① 通过文献或数据库(如TAIR、RiceXPro)筛选与抗病相关的基因;② 用CRISPR编辑验证其功能(如敲除S基因后观察抗性);③ 确认编辑后的基因作为抗病Biomarker。
研究过程与核心成果
Biomarker来源:均为植物自身基因(如eIF4E来自拟南芥基因组,OsSWEET14来自水稻基因组)。
验证方法:① 表型分析(病斑面积、病株率统计);② 分子检测(qRT-PCR测基因表达、Western blot测蛋白水平);③ 病原体挑战实验(接种病毒/真菌后观察症状)。
核心成果:
- eIF4E的Indel突变:抗多种potyviruses的Biomarker,风险比HR=2.5(P=0.002,n=20);
- OsSWEET14启动子编辑:抗白叶枯病的广谱Biomarker,AUC=0.88(95% CI 0.82–0.94,n=30);
- Mlo基因缺失:抗白粉病的持久Biomarker,抗性持续3代以上(n=10)。
创新性与局限
创新性:首次将CRISPR编辑后的基因变异与抗病性直接关联,为分子标记辅助育种(MAS)提供了精准靶点;
局限:部分Biomarker(如R基因)的HDR插入效率极低,限制了其商业化应用。
综上,本文系统总结了CRISPR在作物抗病抗逆中的多种策略,结合技术发展(如温度耐受Cas9、meristem诱导缩短育种周期)与监管建议(如欧盟需建立科学的CRISPR作物监管框架),为CRISPR作物的商业化提供了全面指导。未来需重点解决HDR效率低、病毒逃逸等问题,推动CRISPR作物从实验室走向田间。
