1. 领域背景与文献引入
文献英文标题:PGRMC1 phosphorylation affects cell shape, motility, glycolysis, mitochondrial form and function, and tumor growth;发表期刊:BMC Molecular and Cell Biology;影响因子:未明确;研究领域:肿瘤生物学(胰腺癌相关细胞生物学与代谢调控)。
Progesterone受体膜组分1(PGRMC1)是一类进化保守的膜相关蛋白,在乳腺癌、胰腺癌等多种恶性肿瘤中高表达,且其表达水平与患者不良预后密切相关。现有研究表明,PGRMC1参与调控细胞膜运输、类固醇激素合成、抗凋亡及细胞周期进展等多种生物学过程,但其磷酸化状态如何系统调控细胞形态、代谢及肿瘤生长的具体机制尚未明确。PGRMC1含有多个进化上保守的磷酸化位点(如Y180、S181),这些位点的磷酸化修饰可能作为信号枢纽,整合下游通路(如PI3K/AKT、线粒体代谢)影响细胞行为。本研究针对这一空白,以MIA PaCa-2胰腺癌细胞为模型,通过构建PGRMC1磷酸化位点突变体,系统探究其对细胞形态、代谢、线粒体功能及肿瘤生长的调控作用,为阐明PGRMC1在癌症中的作用机制提供新依据。
2. 文献综述解析
作者将现有PGRMC1研究分为三类:① PGRMC1表达与癌症预后的关联(高表达与乳腺癌、胰腺癌不良生存率相关);② PGRMC1的基础细胞功能(参与膜运输、类固醇生成、抗凋亡);③ PGRMC1磷酸化位点的进化意义(Y180等位点在进化上早于后口动物的组织分化,可能参与原始信号调控)。现有研究的关键结论是PGRMC1作为“信号枢纽”影响癌症进展,但技术局限在于缺乏对磷酸化状态的系统功能分析(多集中于单一功能验证)。本研究的创新点在于:首次通过构建双突变体(DM,S57A/S181A)和三突变体(TM,S57A/Y180F/S181A),结合多组学(蛋白质组)、细胞功能及体内肿瘤实验,全面解析PGRMC1磷酸化状态对细胞生物学行为的调控作用,填补了PGRMC1磷酸化功能研究的空白。
3. 研究思路总结与详细解析
本研究以“PGRMC1磷酸化状态调控细胞行为”为核心,采用“突变体构建→表型分析→机制探索→体内验证”的闭环思路:① 构建WT(野生型)、DM(双丝氨酸突变)、TM(额外酪氨酸突变)稳定细胞系;② 通过显微镜、划痕实验、Transwell实验分析形态与运动能力;③ 利用SWATH-MS蛋白质组学解析差异蛋白通路;④ 通过葡萄糖摄取、线粒体呼吸实验验证代谢功能;⑤ 最终通过小鼠皮下移植瘤实验验证体内肿瘤生长。
3.1 突变体构建与稳定细胞系建立
实验目的是获得表达不同PGRMC1磷酸化突变体的稳定MIA PaCa-2细胞系。方法:构建携带HA标签的PGRMC1野生型(WT)、双突变体(DM,S57A/S181A,突变两个丝氨酸磷酸化位点)和三突变体(TM,S57A/Y180F/S181A,额外突变酪氨酸位点Y180)质粒,通过Lipofectamine 2000转染细胞,经50μg/ml潮霉素筛选2周获得稳定株。结果:Western blot验证显示,外源性PGRMC1-HA(~32kDa)与内源性PGRMC1(~24kDa)表达水平相当(n=4 lanes for MP,n=6 for WT/DM/TM,ANOVA无显著差异);抗HA抗体进一步确认外源性蛋白表达。实验所用关键产品:HA标签抗体(Sigma-Aldrich,H3663)、Lipofectamine 2000(Invitrogen,11668-019)。
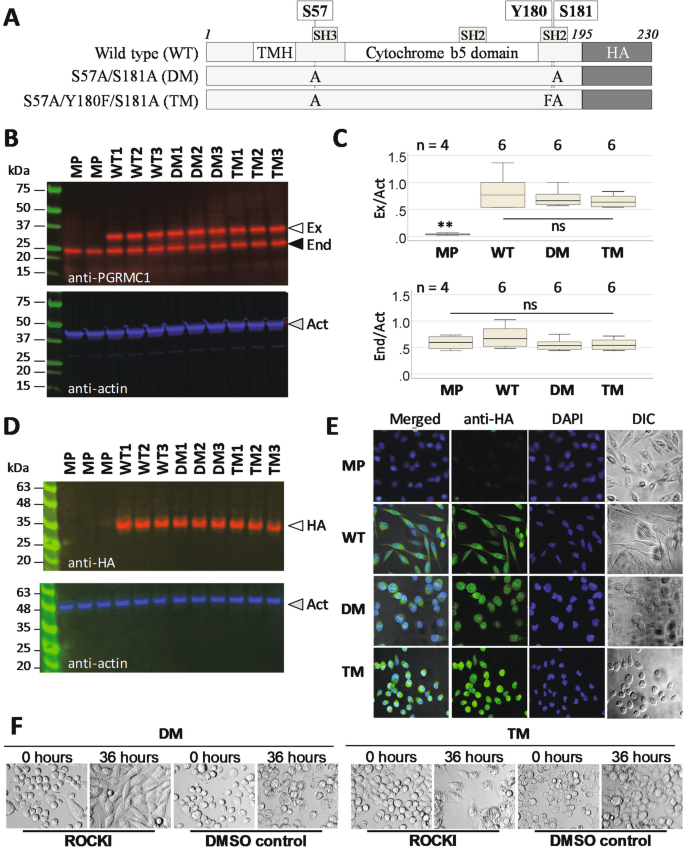
3.2 细胞形态与运动能力分析
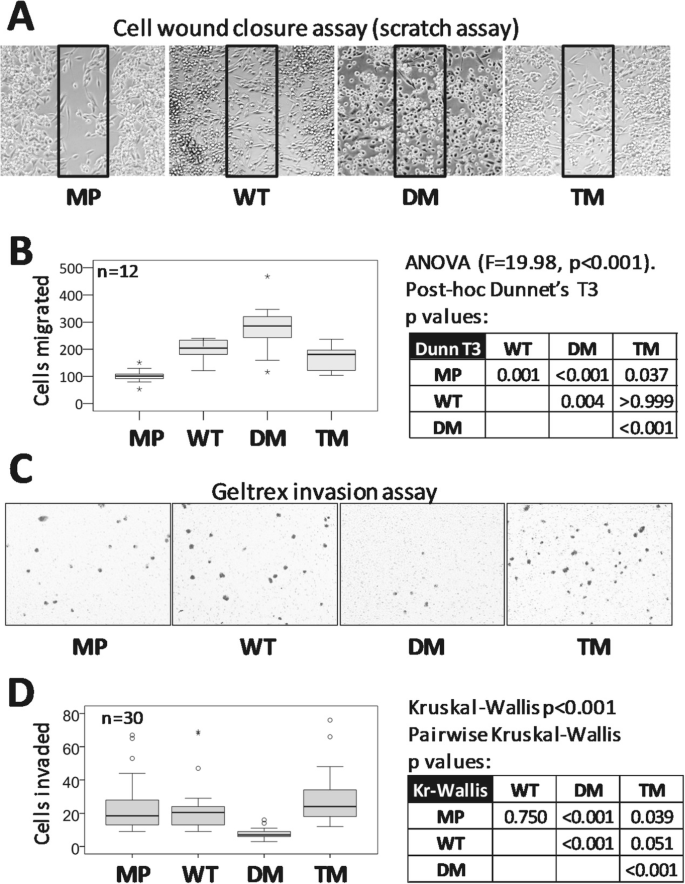
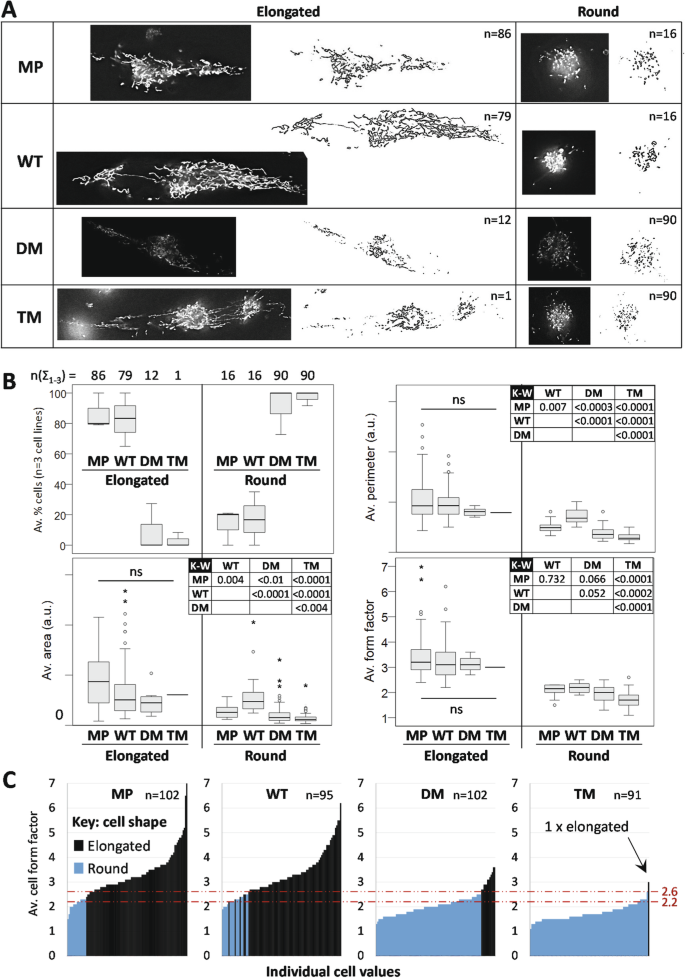
实验目的是探究PGRMC1磷酸化突变对细胞形态、迁移及侵袭的影响。方法:① 形态分析:细胞经甲醛固定、甲醇透化后,用FITC-抗HA抗体染色, confocal显微镜观察(DAPI染核、DIC模式);② 迁移实验:单层细胞划痕后,用ROCK抑制剂(Y-27632)或DMSO处理,观察36小时迁移情况;③ 侵袭实验:细胞接种Matrigel包被的Transwell小室,24小时后计数穿膜细胞。结果:① DM/TM细胞呈现圆形形态(WT为长梭形),ROCK抑制剂可逆转该表型;② 划痕实验中,DM细胞迁移数较WT高60%(n=12,ANOVA post-hoc Dunnet’s T3,P<0.001);③ Transwell实验中,DM细胞侵袭数较WT低50%(n=30,Kruskal-Wallis,P<0.001)。实验所用关键产品:FITC-抗HA抗体(Sigma-Aldrich,F8521)、ROCK抑制剂Y-27632(Abcam,ab120129)。
3.3 蛋白质组学分析
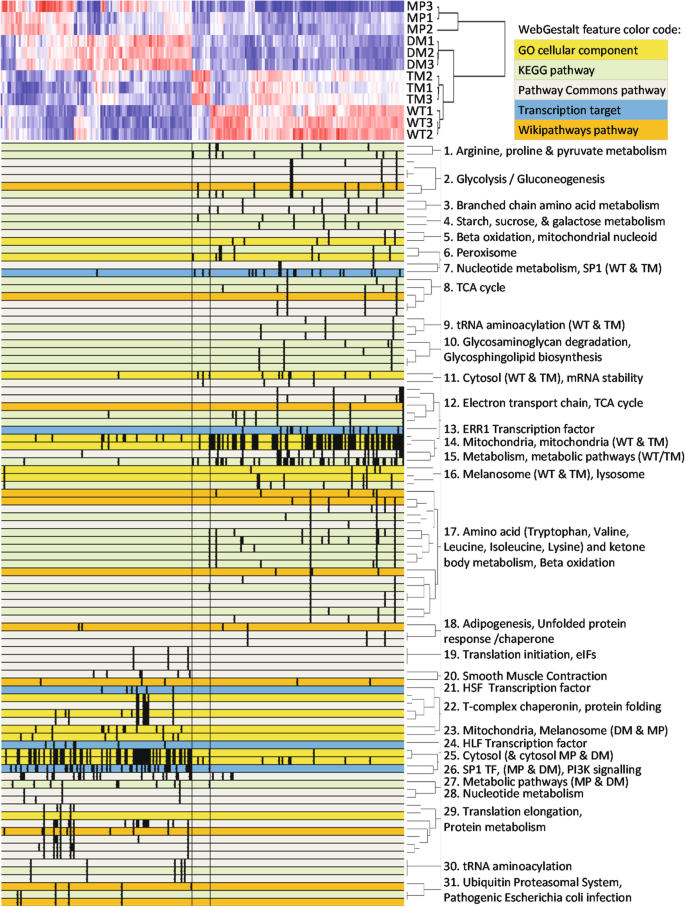
实验目的是解析PGRMC1磷酸化突变对细胞蛋白质组的影响。方法:收集WT、DM、TM及亲本MP细胞,提取蛋白后通过SWATH-MS(ABSciex 5600 TripleTof质谱仪)进行定量蛋白质组学分析。结果:共鉴定1330种可靠蛋白(至少2个肽段),其中243种差异表达(fold change>1.5,P<0.05)。主成分分析(PCA)显示,差异蛋白主要富集于能量代谢(如ATP合酶亚基)、线粒体功能(如TOM40)、细胞骨架(如微管蛋白)等通路。例如,WT细胞中能量代谢蛋白(如磷酸果糖激酶,P08237)表达较DM高30%,而DM细胞中细胞骨架蛋白(如vinculin,P18206)表达较WT高40%。实验所用关键产品:TripleTof 5600质谱仪(ABSciex)、PeakView 2.1数据分析软件。
3.4 代谢与线粒体功能验证
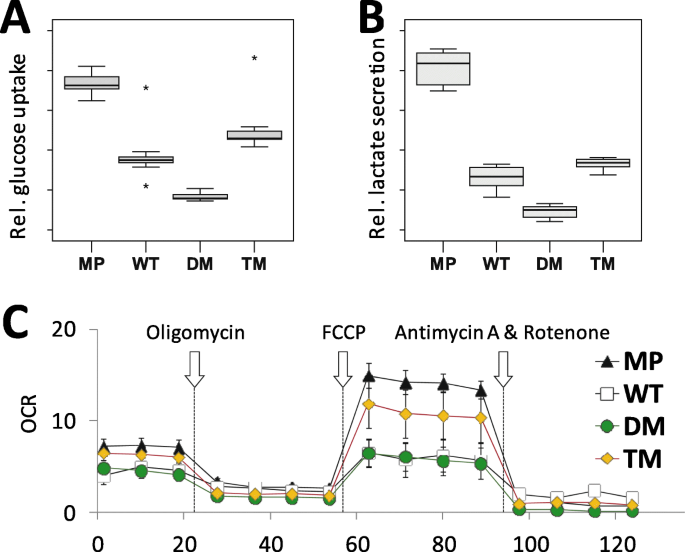
实验目的是验证PGRMC1磷酸化突变对糖代谢及线粒体功能的影响。方法:① 糖代谢:用Cayman试剂盒检测葡萄糖摄取和乳酸分泌;② 线粒体功能:用NpFR2 redox传感器检测基质氧化态,MitoTracker染色结合流式细胞术检测膜电位(Δψm),Seahorse XF24分析仪检测呼吸速率(OCR)。结果:① DM细胞葡萄糖摄取较WT降低40%(n=12,Kruskal-Wallis,P<0.0001),乳酸分泌降低30%(n=6,ANOVA,P<0.003);② WT/TM细胞线粒体基质更氧化(NpFR2荧光强度高2倍),DM细胞线粒体膜电位(MitoTracker荧光)低30%;③ Seahorse实验显示,DM细胞基础呼吸速率较WT低50%,最大呼吸速率低60%(n=5,mean±s.d.,P<0.05)。进一步分析发现,WT细胞中线粒体融合蛋白Opa1表达较DM高40%,提示PGRMC1磷酸化调控线粒体融合/分裂。实验所用关键产品:Cayman葡萄糖摄取试剂盒(600470)、MitoTracker Red(Invitrogen,M7512)、Seahorse XF24分析仪(Agilent)。
3.5 体内肿瘤生长验证
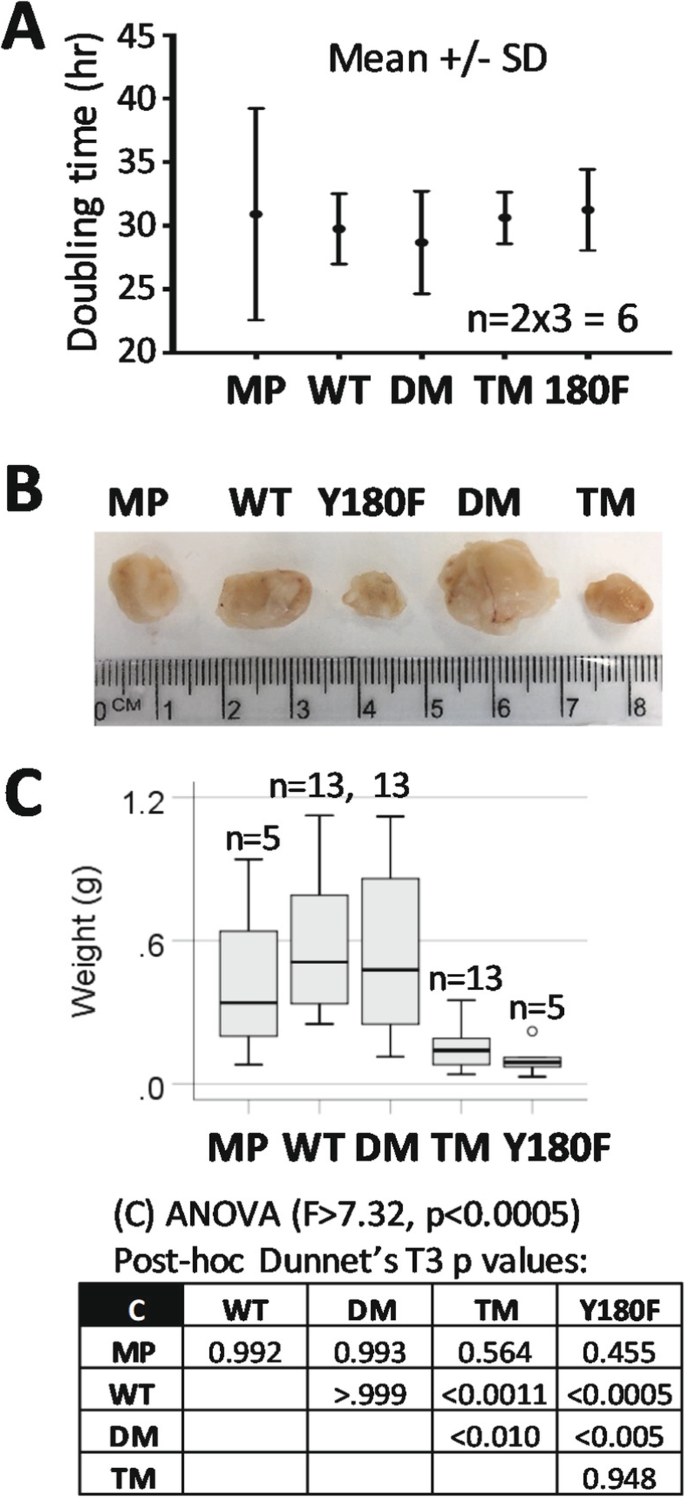
实验目的是探究PGRMC1磷酸化突变对体内肿瘤生长的影响。方法:将WT、DM、TM稳定细胞系(2×10^6细胞)注射到NOD/Shi-SCID/IL-2Rγnull小鼠皮下,每周测量肿瘤大小至体积达1cm³。结果:TM和Y180F单突变体肿瘤体积较WT小60%(n=13,ANOVA post-hoc Dunnet’s T3,P<0.001),表明Y180磷酸化是肿瘤生长的关键驱动因素。实验所用动物:NOD/Shi-SCID/IL-2Rγnull小鼠(澳大利亚表型组学设施提供)。
4. Biomarker研究及发现成果解析
Biomarker定位与筛选逻辑
本研究的核心Biomarker是PGRMC1的Y180磷酸化状态。筛选逻辑为:通过定点突变构建不同磷酸化状态的PGRMC1突变体(WT、DM、TM),先在细胞水平验证其对形态、代谢、运动的影响,再通过体内肿瘤实验确认其对肿瘤生长的调控作用,形成“细胞→动物”的验证链条。
研究过程与数据
Biomarker来源为MIA PaCa-2细胞的PGRMC1蛋白,通过突变Y180(酪氨酸)、S57/S181(丝氨酸)位点获得不同磷酸化状态的突变体。验证方法包括:① Western blot(检测蛋白表达);② 蛋白质组学(分析差异通路,如PI3K/AKT、线粒体代谢);③ 细胞功能实验(形态、葡萄糖摄取、线粒体呼吸);④ 体内肿瘤实验(验证肿瘤大小)。
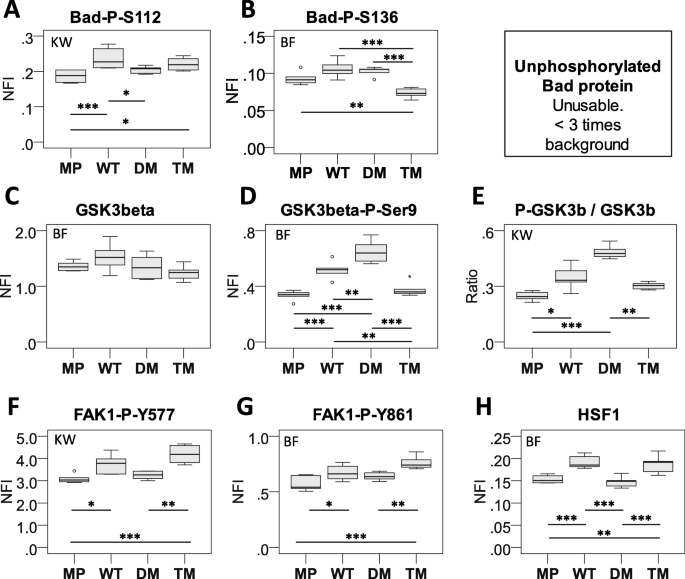
特异性与敏感性数据:① 体内实验中,Y180F突变体(TM)肿瘤体积是WT的1/3(n=13,P<0.001);② 细胞实验中,TM细胞葡萄糖摄取较WT降低40%(n=12,P<0.0001),线粒体最大呼吸速率降低60%(n=5,P<0.05);③ 通路分析显示,TM细胞中AKT底物GSK3β的S9磷酸化水平较WT低50%(Western blot验证),提示Y180磷酸化激活PI3K/AKT通路。
核心成果
本研究首次发现PGRMC1的Y180磷酸化位点是调控细胞行为及肿瘤生长的关键开关,其磷酸化状态可作为胰腺癌预后的潜在Biomarker。具体成果包括:① Y180磷酸化通过激活PI3K/AKT通路,促进细胞增殖与运动;② Y180磷酸化调控线粒体融合/分裂(如Opa1表达),维持线粒体功能;③ 抑制Y180磷酸化(如TM突变体)可显著抑制体内肿瘤生长。这些结果为针对PGRMC1磷酸化位点的癌症治疗提供了新靶点。
