1. 领域背景与文献引入
文献英文标题:Whole genome transcriptome polymorphisms in Arabidopsis thaliana;发表期刊:Genome Biology;影响因子:未公开;研究领域:植物基因组学/拟南芥自然转录组多态性研究。
植物自然转录组变异是形态、生理多样性及适应进化的分子基础,涉及基因表达水平变异(受顺式/反式调控)、可变剪接(产生转录本多样性)和等位基因特异性表达(ASE)(直接反映顺式调控)三大维度。此前研究多采用基因表达阵列解析基因表达变异,但依赖已知注释,无法覆盖未注释区域;全基因组tiling阵列虽能无偏覆盖整个基因组,却因SFP(单特征多态性)探针的序列多态性干扰,未充分用于自然种群的转录组多态性分析。当前研究空白:缺乏全基因组层面多维度解析自然转录组变异(转录水平、可变剪接、ASE)的研究,tiling阵列的SFP校正及de novo转录组分析方法未完善。
本研究以拟南芥Col(哥伦比亚)和Van(温哥华)两个生态型为材料,利用全基因组tiling阵列结合基因组杂交校正SFP,多维度解析转录组多态性,开发广义隐马尔可夫模型(HMM)进行de novo分析,填补了tiling阵列在自然转录组变异中的应用空白,为植物适应进化的分子机制提供了新见解。
2. 文献综述解析
作者按“转录组变异类型(基因表达、可变剪接、ASE)+ 技术应用(基因表达阵列vs tiling阵列)”的逻辑综述现有研究,核心结论如下:
现有研究的关键结论与局限性
- 基因表达变异:自然种群中基因表达变异普遍(拟南芥中4%-32%基因差异表达),顺式调控(通过ASE检测)是重要驱动因素,但此前研究受基因表达阵列的注释bias限制,无法解析未注释区域的变异。
- 可变剪接:是转录组多样性的核心来源,植物中内含子保留更常见,但传统方法依赖注释,难以检测de novo剪接事件。
- 技术局限:基因表达阵列仅覆盖已知基因,tiling阵列的SFP探针会干扰表达定量,其校正方法未系统评估;de novo转录组分析缺乏高效工具。
本研究的创新价值
- 技术校正:结合基因组DNA杂交数据过滤SFP探针,减少tiling阵列的技术bias;
- 多维度解析:用线性模型区分基因表达的加性、显性和母性效应,全面评估遗传调控;
- de novo分析:开发广义HMM,无需注释即可检测差异转录片段;
- 功能关联:将转录组变异与环境适应通路(光、盐、激素响应)关联,揭示适应进化的分子机制。
3. 研究思路总结与详细解析
整体框架
以拟南芥Col和Van生态型为材料,通过全基因组tiling阵列分析基因组DNA(检测SFP和结构变异)与cDNA(检测转录组变异)的杂交数据,多维度解析:① 基因表达的遗传效应(加性、显性、母性);② 等位基因特异性表达(ASE);③ 可变剪接(外显子差异、内含子保留);④ de novo差异转录片段。技术路线为“样本制备→SFP/结构变异检测→转录组变异分析→de novo验证”的闭环。
3.1 植物材料与样本制备
实验目的:获得Col、Van及其杂交后代的高纯度gDNA和cDNA样本,用于tiling阵列杂交。
方法细节:Col和Van种子经春化(4°C,5天)后温室培养,取3天幼苗分别提取gDNA(Qiagen DNeasy kit)和总RNA(Qiagen RNeasy kit);总RNA富集poly(A) RNA后合成双链cDNA,用Invitrogen BioPrime系统标记gDNA和cDNA。
结果解读:成功制备4个母本重复的gDNA和cDNA样本,满足tiling阵列杂交的质量要求。
实验所用关键产品:Qiagen DNeasy植物mini kit、RNeasy植物mini kit、Invitrogen BioPrime DNA标记系统。
3.2 SFP与基因组结构变异检测
实验目的:识别Col和Van之间的序列多态性(SFP)及结构变异(缺失/重复),为后续转录组分析校正SFP干扰。
方法细节:对gDNA杂交的.CEL文件进行空间校正和log转换,用Bioconductor包Siggenes检测SFP(5% FDR);用segmentation算法分析探针水平p值(H₁: μVan > μCol),识别缺失(median p>0.99)和重复(median p<0.02)。
结果解读:共检测到125,043个SFP(5% FDR),其中118,381个Col信号更强;识别到1,645个缺失和136个重复(Van相对于Col),长度集中在500 bp左右,且富集在着丝粒周围区域(图1)。
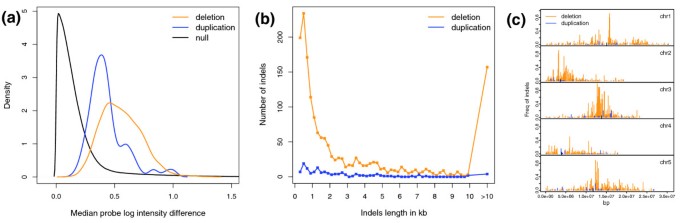
实验所用关键产品:Bioconductor包Siggenes、自定义segmentation算法。
3.3 基因表达变异的遗传效应分析
实验目的:解析Col、Van及其正反交F₁的基因表达变异,区分加性(父母本差异)、显性(F₁偏离中亲值)和母性(正反交F₁差异)效应。
方法细节:过滤cDNA杂交数据(排除低质量、SFP、间区探针),用线性模型Intensity = Additive + Dominant + Maternal + Error分析,通过1000次permutation计算FDR;用k-means聚类分析基因型间的表达模式。
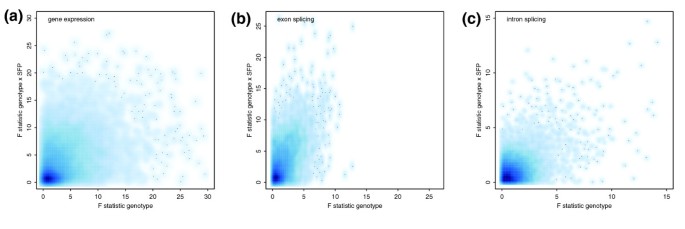
结果解读:8%(1,925个)基因存在加性效应(2% FDR),66% Col中上调;3%(667个)基因存在显性效应(6% FDR),多表现为Van的显性负效应(F₁表达低于中亲值);<1%(163个)基因存在母性效应(17% FDR)。功能富集显示,Col中上调基因富集在叶绿素合成、盐胁迫响应通路,Van中上调基因富集在类黄酮合成通路(图2)。
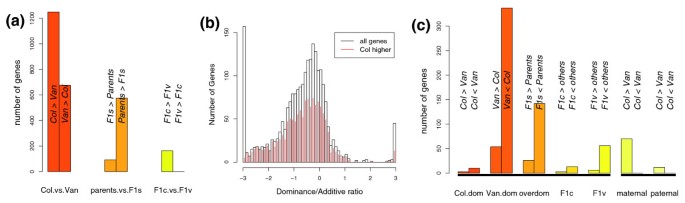
实验所用关键产品:自定义R脚本(线性模型、permutation)、R stats包(k-means聚类)。
3.4 等位基因特异性表达(ASE)检测
实验目的:识别顺式调控变异(通过ASE直接检测),区分顺式与反式调控的贡献。
方法细节:筛选含≥5个非SFP探针和≥1个SFP探针的基因,用非SFP探针估计基因表达水平,校正SFP探针的强度;以基因组DNA杂交的中亲值为参考,分析F₁杂交种的RNA杂交数据,通过线性回归检测ASE,permutation计算FDR。
结果解读:共检测到540个Col-ASE基因(1% FDR)和209个Van-ASE基因(0.1% FDR),ASE基因富集在差异表达基因中(fold enrichment>3)。例如AT4G29950表现为Col等位基因优先表达(图3)。
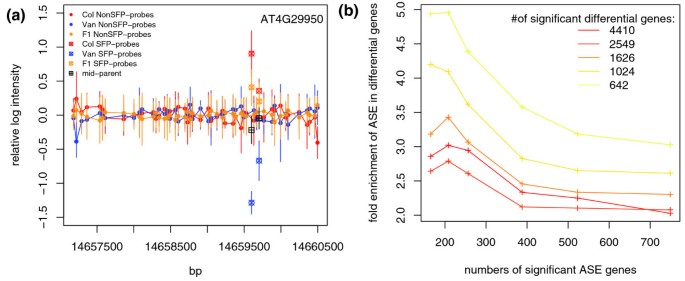
实验所用关键产品:自定义R脚本(线性回归、permutation)。
3.5 可变剪接变异分析
实验目的:检测外显子差异表达与内含子保留,解析转录组多样性的来源。
方法细节:对外显子探针,用基因表达水平校正后分析差异;对内含子探针,直接分析强度差异(内含子保留的信号更高);用线性模型检测加性效应,RT-PCR验证(Primer3设计引物)。
结果解读:0.35%(236个)外显子存在差异剪接(41% FDR),0.73%(459个)内含子存在差异保留(3% FDR)。内含子保留基因富集在叶绿体类囊体膜(p<4.51E-04),涉及光系统功能。RT-PCR验证显示,61%的差异内含子保留为真阳性(图4)。
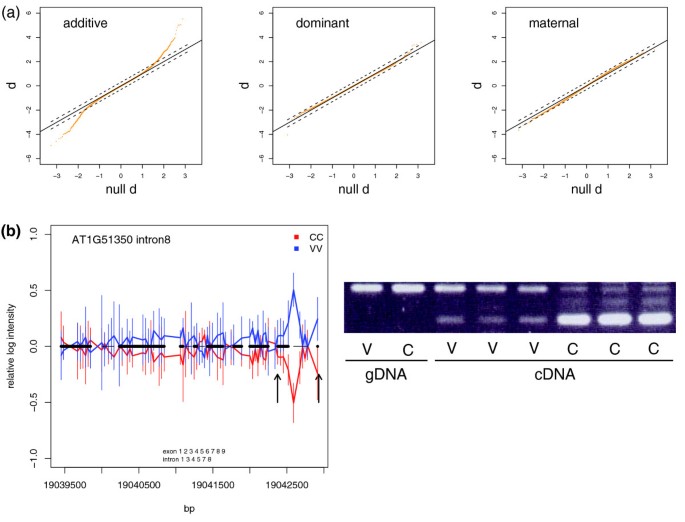
实验所用关键产品:Primer3(引物设计)、琼脂糖凝胶电泳系统。
3.6 de novo转录组变异分析
实验目的:无注释检测Col和Van之间的差异转录片段,发现未注释基因与新剪接事件。
方法细节:对cDNA杂交的探针p值(H₁: μVan > μCol)构建广义HMM,含3个隐藏状态(相等、Van高、Col高);用Baum-Welch算法估计发射概率与转移概率,Forward-Backward算法计算后验概率,识别差异片段(posterior>0.99)。
结果解读:共检测到6,800个差异片段,其中2,673个对应差异表达基因,1,222个对应差异剪接基因,109个新基因边界,85个非注释转录本(图5、6)。
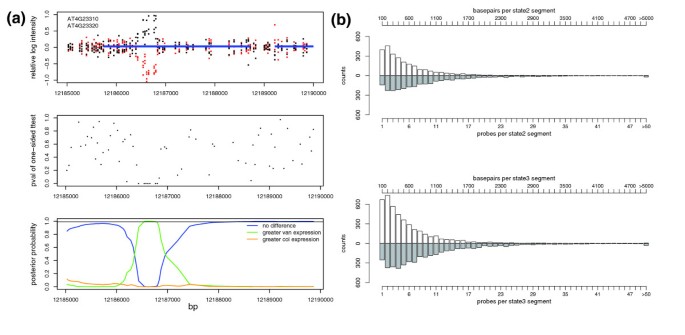
实验所用关键产品:自定义R脚本(广义HMM、Baum-Welch算法、Forward-Backward算法)。
4. Biomarker研究及发现成果解析
Biomarker定位
本研究的Biomarker为拟南芥生态型适应的转录组特征,包括:① 差异表达基因(环境适应的核心通路);② ASE基因(顺式调控的分子标志);③ 差异剪接基因(转录组多样性的来源)。三者共同作为生态型适应不同生境的分子标志物。
筛选与验证逻辑
- 差异表达基因:通过tiling阵列检测加性效应基因(FDR=2%),结合功能富集分析(parametric gene set enrichment)验证其与环境响应的关联;
- ASE基因:通过SFP探针检测等位基因优先表达(FDR=1%),富集分析验证其与差异表达基因的关联;
- 差异剪接基因:通过内含子/外显子探针检测差异保留(FDR=3%),RT-PCR验证真阳性率(61%)。
核心成果提炼
- 差异表达基因:环境适应的Biomarker
- 8%的差异表达基因富集在光、盐、激素响应通路(如Col中叶绿素合成基因上调,适应光照环境;Van中类黄酮合成基因上调,适应生物胁迫);
统计学结果:分析基因数n=24,756,加性效应FDR=2%,功能富集p<0.05(如叶绿素合成通路p<0.01)。
ASE基因:顺式调控的Biomarker
- 540个Col-ASE基因和209个Van-ASE基因,富集在差异表达基因中(fold enrichment>3);
首次在Col和Van中系统揭示ASE与顺式调控的关联,为解析基因表达变异的遗传基础提供了直接证据。
差异剪接基因:转录组多样性的Biomarker
- 0.73%的内含子保留基因富集在叶绿体类囊体膜(光系统组分),首次发现内含子保留与植物光合作用适应的关联;
- RT-PCR验证的真阳性率为61%,表明内含子保留是植物适应环境的重要转录调控方式。
本研究通过全基因组tiling阵列的多维度分析,为植物自然转录组变异的研究提供了范式,其发现的转录组Biomarker为解析植物适应进化的分子机制提供了关键线索。
