1. 领域背景与文献引入
文献英文标题:Virulence determinants, drug resistance and mobile genetic elements of Laribacter hongkongensis: a genome-wide analysis;发表期刊:Cell Bioscience;影响因子:未公开;研究领域:肠道病原菌基因组学与感染性疾病
肠道病原菌是全球社区获得性胃肠炎的主要致病原之一,2001年香港拉里克特菌(Laribacter hongkongensis)作为β-变形菌门奈瑟菌科的新属新种,首次在肝硬化患者的血液及脓胸脓液中被分离鉴定。后续流行病学研究证实,该菌可通过淡水鱼传播,引发社区获得性胃肠炎及旅行者腹泻,临床症状与沙门氏菌、弯曲菌感染类似,但此前针对该病原菌的研究多聚焦于分离鉴定、流行病学调查及单个基因的功能验证,缺乏全基因组层面的系统解析,其感染宿主的关键分子机制、耐药性遗传基础及可移动遗传元件的进化特征等核心问题尚未明确。本研究通过全基因组注释与功能分析,系统解析该病原菌的毒力决定因子、耐药基因谱及可移动遗传元件,为其感染防控及分子溯源提供关键理论依据。
2. 文献综述解析
作者围绕香港拉里克特菌感染过程的关键环节、耐药机制、可移动遗传元件三个核心维度,对现有研究进行分类评述。现有研究已明确该病原菌与淡水鱼传播的胃肠炎关联,初步鉴定出部分表面黏附蛋白,但毒力机制研究仅局限于个别基因的初步探索,未系统解析其适应宿主肠道环境、逃避宿主防御的分子网络;耐药性研究仅报道了单个C类β-内酰胺酶基因,未全面揭示其多药耐药的遗传基础;可移动遗传元件的研究几乎处于空白状态,无法解释该病原菌的遗传进化特征。现有研究的局限性在于样本量较小、研究维度单一,缺乏全基因组层面的整合分析,且未明确各毒力因子间的协同作用机制。本研究的创新价值在于首次通过全基因组视角,系统鉴定了该病原菌感染各环节的关键基因、全面梳理其耐药基因谱,并发现大量前噬菌体及转座子等可移动遗传元件,填补了该病原菌全基因组研究的空白,为其致病性及进化研究提供了全新框架。
3. 研究思路总结与详细解析
本研究以香港拉里克特菌HLHK9株为研究对象,核心目标是系统解析其基因组中与毒力、耐药及可移动遗传元件相关的基因及基因盒;核心科学问题聚焦于该病原菌如何通过基因组编码的分子机制适应宿主肠道环境、逃避宿主防御、产生毒力及耐药性;技术路线遵循“全基因组注释→功能分类筛选→靶向基因解析→进化特征分析→结论整合”的闭环逻辑。
3.1 全基因组注释与功能分类筛选
实验目的:对香港拉里克特菌HLHK9株基因组进行全面功能注释,筛选出与毒力、耐药及可移动遗传元件相关的编码序列。
方法细节:采用Clusters of Orthologous Groups(COG)方法对基因组编码序列进行功能分类,去除染色质结构、管家代谢等管家功能相关基因,保留与胞内转运、分泌、防御机制等潜在毒力相关的COG簇;结合Virulence Factor Database(VFDB)数据库及关键词搜索(包括virulence、toxin、resistance等)进一步筛选目标基因;通过BLAST序列比对、保守域搜索(CD-search)、InterProScan等工具进行手动注释与功能确认。
结果解读:共鉴定出大量与酸抵抗、胆汁抵抗、肠道定植、毒力因子、耐药及可移动遗传元件相关的编码序列,为后续分模块的深入解析提供了基础数据支撑。
产品关联:文献未提及具体实验产品,领域常规使用基因组注释软件(如Prokka)、序列比对工具(如BLAST)、专业功能数据库(如VFDB、COG)等。
3.2 毒力相关基因系统解析
实验目的:解析香港拉里克特菌感染宿主过程中各关键环节的分子机制,包括酸环境适应、胆汁抵抗、肠道定植、宿主防御逃逸及毒力因子作用。
方法细节:对筛选出的毒力相关基因进行多序列比对,采用邻接法构建系统进化树(Kimura校正,1000次bootstrap验证),结合现有文献分析其功能及进化特征。
结果解读:
- 酸抵抗机制:鉴定出一个完整的脲酶基因盒(包含8个编码序列),可水解胃内尿素产生氨以提升局部pH;同时发现两个相邻的精氨酸脱亚胺酶(arc)基因簇,通过将精氨酸转化为氨以适应酸性环境,进化分析显示该基因簇与紫色色杆菌同源性最高,且该菌是目前已知唯一携带两个相邻arc簇的细菌。
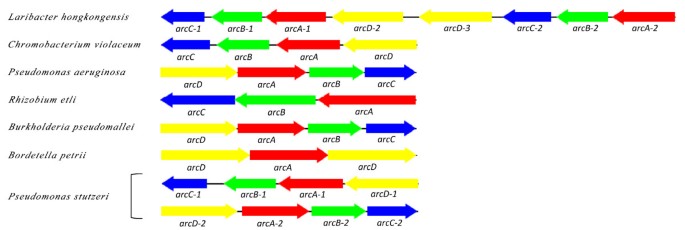
- 胆汁抵抗机制:发现3套完整的acrAB-tolC外排系统,以及emrAB-tolC、acrAD-tolC等4种其他胆汁外排泵基因盒;同时存在维持外膜完整性的tol基因簇(包含tolQ、tolR、tolA、tolB、pal),其中tolQ未与其他tol基因成簇,这一特征与紫色色杆菌类似。
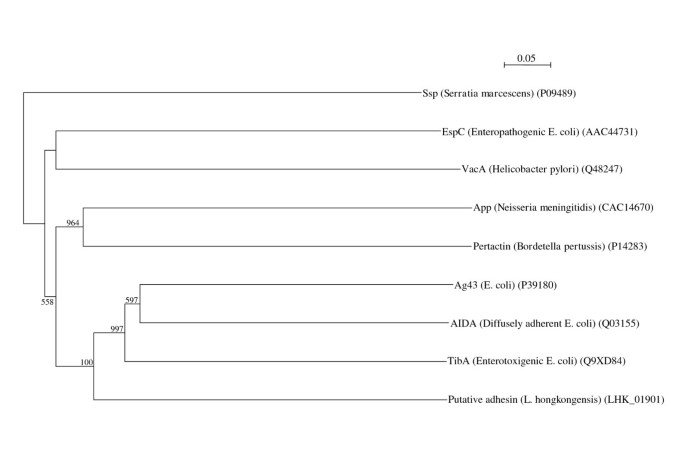
- 肠道定植机制:鉴定出一种自转运蛋白家族的黏附素,与弥散黏附性大肠杆菌(DAEC)的AIDA-I和肠产毒性大肠杆菌(ETEC)的TibA黏附素同源性为27-30%,其 passenger结构域含9个VXSGG重复序列,上游存在假定的庚糖基转移酶基因,推测该黏附素通过庚糖修饰介导肠道黏膜黏附。
- 宿主防御逃逸机制:编码超氧化物歧化酶和3种过氧化氢酶(katE及两个katG),可清除宿主吞噬细胞产生的活性氧;部分多药外排泵还可外排宿主来源的抗菌物质,进一步增强其存活能力。
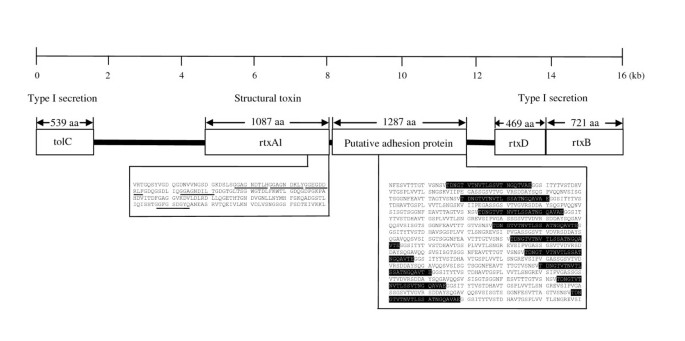
- 毒力因子:脂多糖(LPS)合成基因与大肠杆菌类似,但脂A具有对称酰化模式,由htrB和msbB基因编码的酶催化,进化分析显示这两个基因与奈瑟菌属同源性较高;鉴定出RTX毒素(包含一个基因簇和一个孤立的rtxA2基因)、两种溶血素、3个patatin样蛋白(具有磷脂酶A2活性,可破坏宿主细胞膜)、外膜磷脂酶A(OMPLA,可促进其他毒力因子释放)。
产品关联:文献未提及具体实验产品,领域常规使用系统进化分析软件(如MEGA)、保守域分析工具(如InterProScan)等。
3.3 耐药基因谱全面解析
实验目的:系统解析香港拉里克特菌的耐药基因组成及潜在耐药机制。
方法细节:对COG分类中与防御机制、次级代谢产物转运等相关的编码序列进行手动注释,结合关键词搜索(包括resistance、efflux等)鉴定耐药相关基因。
结果解读:共鉴定出10个β-内酰胺类耐药相关基因,包括6个青霉素结合蛋白(PBP)、1个已表征的C类β-内酰胺酶、2个假定的金属β-内酰胺酶;54个多药外排泵基因,覆盖5个主要家族(RND、MFS、SMR、MATE、ABC),其中RND家族的acrAB-tolC系统可介导多种抗生素耐药,MFS家族的emrAB-tolC系统可介导萘啶酸耐药;还鉴定出卡那霉素、杆菌肽、砷剂等其他耐药相关基因。

产品关联:文献未提及具体实验产品,领域常规使用耐药基因注释工具(如CARD数据库)等。
3.4 可移动遗传元件鉴定与分析
实验目的:鉴定香港拉里克特菌基因组中的前噬菌体、转座子等可移动遗传元件,分析其对基因组进化的影响。
方法细节:使用Prophage Finder工具预测前噬菌体,通过BLASTP比对ISfinder数据库鉴定转座子,利用einverted工具检测反向重复序列以确认插入序列。
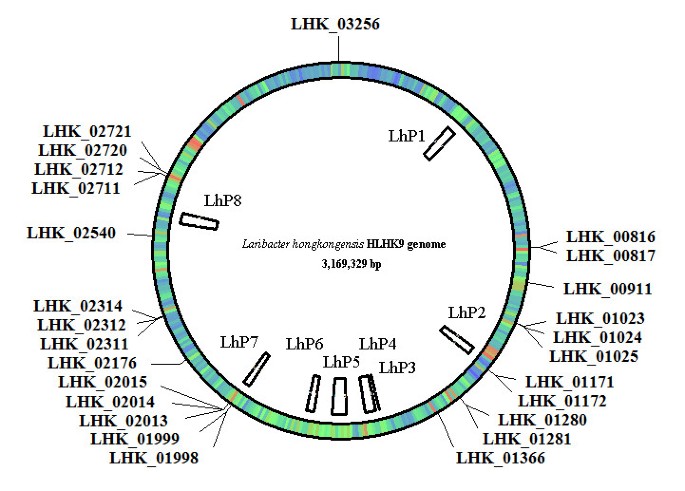
结果解读:基因组中存在8个假定前噬菌体(LhP1-LhP8),其中LhP1和LhP8为P2样噬菌体,其余为Mu样、lambda样等不同类型,前噬菌体数量远高于同科的奈瑟菌属和紫色色杆菌,提示水平基因转移在该菌进化中起重要作用;还鉴定出26个转座酶基因,分属IS3、IS5、IS481家族,其中IS481家族转座子为该菌特有,同科其他细菌中不存在,推测其通过水平基因转移获得。
产品关联:文献未提及具体实验产品,领域常规使用前噬菌体预测工具(如Prophage Finder)、转座子分析工具(如ISfinder)等。
4. Biomarker研究及发现成果
本研究未涉及传统临床诊断或预后生物标志物的研究,但鉴定的香港拉里克特菌毒力相关基因、耐药基因及特异性遗传元件可作为该病原菌的分子标志物。
Biomarker定位:本研究鉴定的分子标志物包括毒力相关基因(如脲酶基因盒、arc基因簇、黏附素基因、RTX毒素基因)、耐药基因(如β-内酰胺酶基因、多药外排泵基因)及特异性遗传元件(如IS481家族转座子),筛选逻辑为全基因组注释→功能分类→进化分析→特异性验证。
研究过程详述:这些标志物来源于香港拉里克特菌HLHK9株的基因组序列,通过序列比对、进化分析及功能预测进行验证。其中脲酶基因盒和arc基因簇可作为该菌适应酸性环境的特异性标志物,与其他肠道病原菌相比,该菌是唯一携带两个相邻arc簇的细菌;黏附素基因的VXSGG重复序列及上游庚糖基转移酶基因可作为肠道定植能力的标志物;耐药基因中的C类β-内酰胺酶基因已通过实验验证其功能,可作为β-内酰胺类耐药的分子标志物;IS481家族转座子为该菌特有,可作为病原菌溯源的特异性标志物。
核心成果提炼:这些分子标志物揭示了香港拉里克特菌的毒力机制、耐药性及遗传进化特征,其中两个相邻arc基因簇、IS481家族转座子为该菌特有,为该病原菌的鉴定、溯源及防控提供了特异性分子靶点;但本研究未进行临床样本验证,这些标志物的临床应用价值需进一步实验证实,且未明确其在不同菌株中的保守性,需扩大菌株样本量进行验证。
